Поиск мотивов, программы MEME и MAST
Поиск мотивов (блоков достоверного выравнивания) среди гомологов белка CISY_BACSU
В отличие от предыдущих разделов, связанных с выравниваниями многих белковых последовательностей, здесь мы будем работать не с программами полного (тотального) множественного выравнивания, а с программой MEME (Multiple Em for Motif Elicitation), позволяющей искать блоки - участки множественного выравнивания без гэпов. Таким образом, это программа множественного локального выравнивания.Параметры запроса: Запуск проводился через команду ememe пакета Emboss. В качестве исходных данных для работы программы использовался файл с последовательностями гомологов из выборки (см. раздел "Множественное выравнивание"), сделанной на основе результатов поиска BLAST: sequences.fasta.
Результаты: Выполнена команда ememe sequences.fasta memeout -nmotifs 3 , где -nmotifs 3 указывает на то, что программа будет находить максимально три мотива (но может быть и меньше). Выдача программы доступна в формате html по ссылке ФАЙЛ. Некоторые параметры, характеризующие найденные мотивы, представлены в таблице 1.
Таблица 1. Некоторые характеристики найденных MEME мотивов
| Мотив | Сколько последовательностей содержит | E-value | LOGO |
| MOTIF 1 | 37 (из 38) | 3.0e -490 | 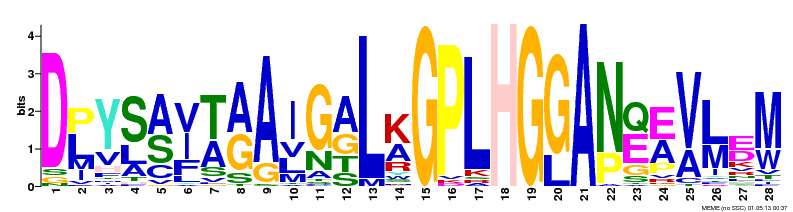 |
| MOTIF 2 | 29 (из 38) | 1.1e -364 | 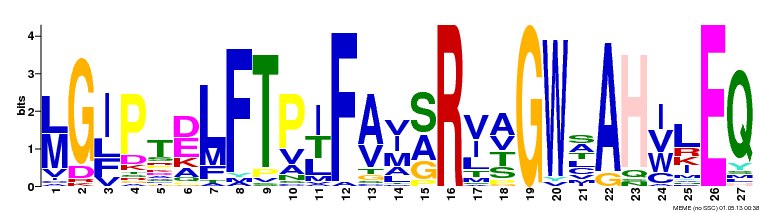 |
| MOTIF 3 | 38 (из 38) | 1.7e -328 | 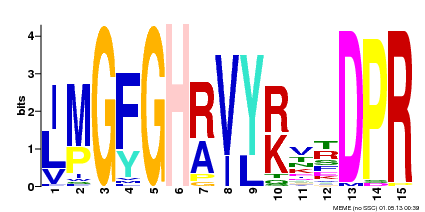 |
Сравнение блоков, найденных MEME, c полным выравниванием - результатом MUSCLE
Сначала из выдачи программы ememe были получены три файла в FASTA-формате, содержащие участки последовательностей, формирующие три вышеописанных мотива. Это было сделано с помощью команды seqret, транслирующей последовательности из формата .aln (выровненные последовательности) в формат .fasta. В Jalview открывались одновременно тотальное выравнивание всей выборки и найденный мотив. Пример проведения визуального сравнения представлен на рисунке 1 (чтобы приблизить, щёлкните левой кнопкой мыши). |
| Рис. 1 Визуальное сравнение участка локального выравнивания и мотива №1, обнаруженного с помощью MEME |
На рисунке 1 выделена единственная последовательность, которая не была включена в состав мотива программой МЕМЕ. Интересно отметить, что при построении дерева последовательностей по проценту идентичности эта последовательность митохондриальной цитратсинтазы оказывается наиболее внешней (т. н. outgroup): TREE. В остальном же выравнивания последовательностей абсолютно идентичны: нет лишних пропусков, все остатки выровнены в колонки одинаково.
 |
| Рис. 2 Визуальное сравнение участка локального выравнивания и мотива №2, обнаруженного с помощью MEME |
На примере этого мотива (см. рисунок 2), содержащего 29 последовательностей, можно ясно видеть, что 9 последовательностей из эукариот, не включённые в него, содержат в соответствующем мотиву участке лишнюю аминокислоту, да и в целом последовательности немного другие. На этом основании, скорее всего, МЕМЕ и не включила эти последовательности в состав мотива. Интересно, что у сравнительно примитивных эукариот наблюдается отсутствие остатка между третьим и четвёртым положениями мотива.
 |
| Рис. 3 Визуальное сравнение участка локального выравнивания и мотива №3, обнаруженного с помощью MEME |
Рисунок 3, на котором производится сравнение мотива 3 с тотальным выравниванием, интересен разницей в представлении последней последовательности >NP_070169.1_Archaeoglobus_fulgidus, которая по-разному выровнена в двух случаях. При глобальном выравнивании был вставлен пропуск во всех остальных последовательностях на месте глицинового остатка, который выбивается из этой последовательности. В это же время в локальном выравнивании пропуски невозможны, и выравнивание получилось явно логически неверным, так как консервативная троица DPR не находится в соответствии с другими последовательностями. Программа локального выравнивания могла бы просто отбросить эту последовательность, что мы могли наблюдать для мотивов 1 и 2, но не сделала этого, что, вероятно, связано с сильным сходством начала мотива последней последовательности с консенсусом для этого мотива.
Поиск найденных мотивов в других последовательностях (программа MAST)
В этом разделе в качестве входных данных использовалось выравнивание, полученное с помощью программы Pfam (см. предыдущий раздел - ССЫЛКА). Это выравнивание доменов, аналогичных найденному в CISY_BACSU среди других белков. Основная же задача этого раздела - поиск мотивов в полученном множественном выравнивании с помощью программы MAST (Motif Alignment & Search Tool). Запуск программы, также как и в предыдущем случае проведём через Putty, воспользовавшись командой emast из пакета EMBOSS.Результаты: Был получен файл с выдачей программы CCЫЛКА.
Краткий анализ результатов: всего на наличие уже известных нам трёх мотивов проанализирована 121 последовательность, суммарная длина выравнивания доменов составила 511 нуклеотидов. Данные о встречаемости обнаруженных мотивов в проанализированных последовательностях представлены в таблице 2.
Таблица 2. Встречаемость мотивов в последовательностях из выравнивания домена
| Комбинация | Количество последовательностей |
| M2 | 1 |
| M1+M2 | 3 |
| M1+M3 | 3 |
| M1+M2+M3 | 114 |
| ИТОГО: | 121 |
* Интересно заметить, что среди последовательностей оказались и те, которые демонстрировали не потерю, а удвоение одного из мотивов: в двух последовательностях удваивался второй мотив, а ещё по одной последовательности демонстрировали дупликации в первом и в третьем мотивах.
Можно заключить, что выравнивание Pfam хорошо соотносится с расположением мотивов в последовательности, т.к. подавляющее большинство из проанализированных последовательностей несут все три найденных мотива, лишь в отдельных случаях один из них не присутствует. Таким образом, понимание того, что найденные белки содержат одинаковый домен, подкреплено и тем, что в них найдены одни и те же консервативные участки - мотивы.
Испытание сервиса MEME Suite на идентичной выборке гомологов
В этом разделе описаны "впечатления" от использования Web-интерфейса для МЕМЕ. Пробный запуск произведён на той же выборке гомологов, что и через Putty (см. выше).- Во-первых, стоит отметить большее по сравнению с запуском через Emboss разнообразие доступных опций. Удобно использовать интерфейс даже без каких-либо предварительных знаний, так как под названием каждой из опций есть краткое пояснение того, на что влияет изменение конкретных параметров. Сразу и единым списком выводятся все характеристики сделанного запроса (см. рис. 4).
 |
| Рис. 4 Характеристики запроса в веб-интерфейсе МЕМЕ |
- Во-вторых, отмечу, что происходит одновременная выдача как результатов МЕМЕ в различных форматах, так и результатов программы MAST, в то время как при запуске через Emboss эти программы запускаются двумя разными командами.
- В целом же выдача через веб-интерфейс не имеет принципиальных отличий по сравнению с результатами обычного запуска программы. Найдены в том числе и те мотивы, о которых уже говорилось выше. В качестве минуса отмечу отсутствие удобных инструментов скачивания и сохранения графической и иной информации. Сложно перенести полученные изображения и таблицы туда, куда необходимо, из полученного отчёта программы.
Анализ карты локального сходства белка с самим собой
В этом разделе я попробую провести анализ карты локального сходства белка TALe (из бактерии Xanthomonas oryzae) с самим собой. Для этого буду использовать сервис DotHelix пакета Genebee. Полученные в результате работы программы (при различных порогах схожести) карты локального сходства представлены на рисунке 5: при получении картинки слева порог сходства составлял 0.01, а для правой картинки он был выбран равным 0.2.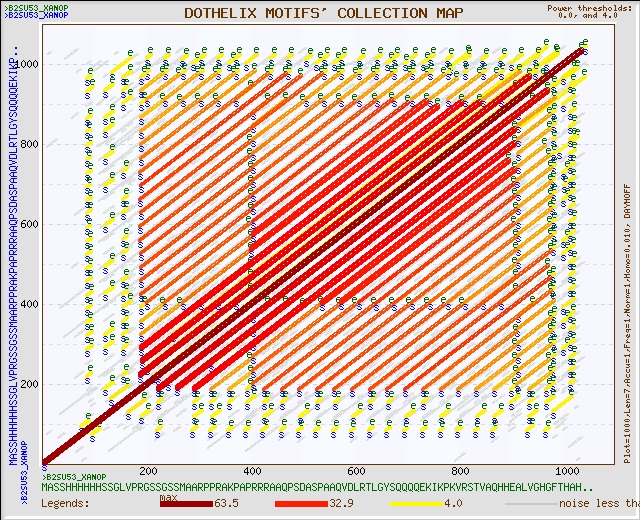 |
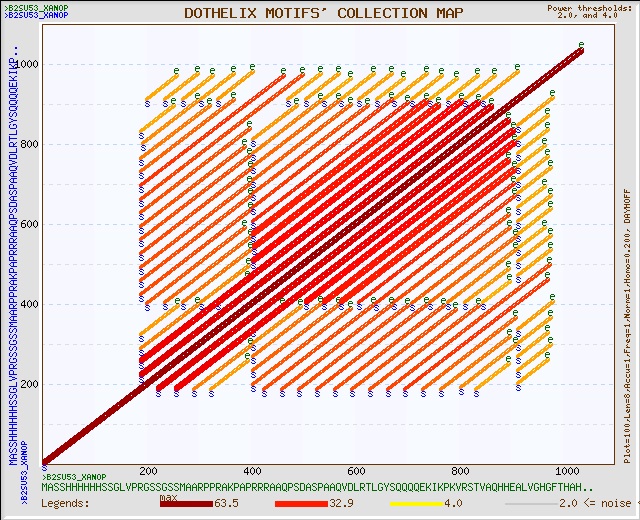 |
| Рис. 5 Карты локального сходства белка TALe с самим собой при различных параметрах запроса | |
Анализ карты: Первое, что надо отметить - полосу полной гомологии по диагонали, наличие которой очевидно, так как белок выравнивался сам с собой. Гораздо более интересно отметить наличие множества участков с высокой степенью гомологии (красные и оранжевые полосы вдоль главной диагонали). Кроме того, интересно, что между соседними двумя полосами расстояние по последовательности одинаковое. Из этого можно сделать предположение о наличии в белке периодичности (некоторой повторяющейся последовательности) на участке примерно от 200 до 970.
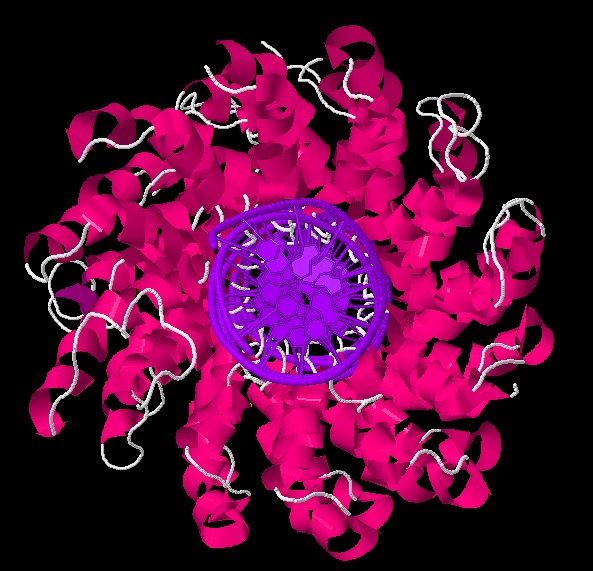
Предположение подтверждено с помощью прямого анализа самой последовательности белка (см. выравнивание повторов) и рассмотрения 3D-структуры белка (см. рисунок 6).
 |
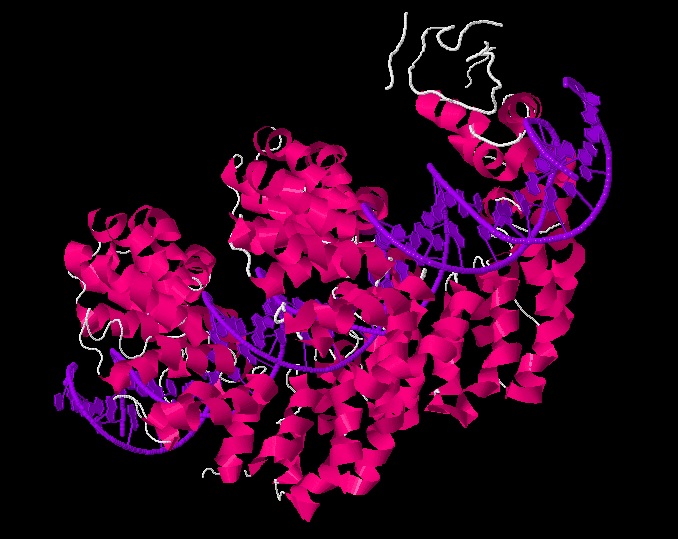 |
| Рис. 6 Структура TALе - эффектора, взаимодействующего с большой бороздкой ДНК. В структуре отчётливо видны повторяющиеся фрагменты из линкера и одной альфа-спирали - это и есть повторы, выявляемые при изучении карты локального сходства. | |
(*) При работе была использована статья: Леонтович А.М., Бродский Д.И., Горбаленя А.Е.; Построение полной карты локального сходства двух биополимеров (программа Dothelix пакета genbee); Биополимеры и Клетка. 1990. Т. 6. № 6.
Дата последнего обновления: 27.05.2013
© Dmitry Travin, 2012