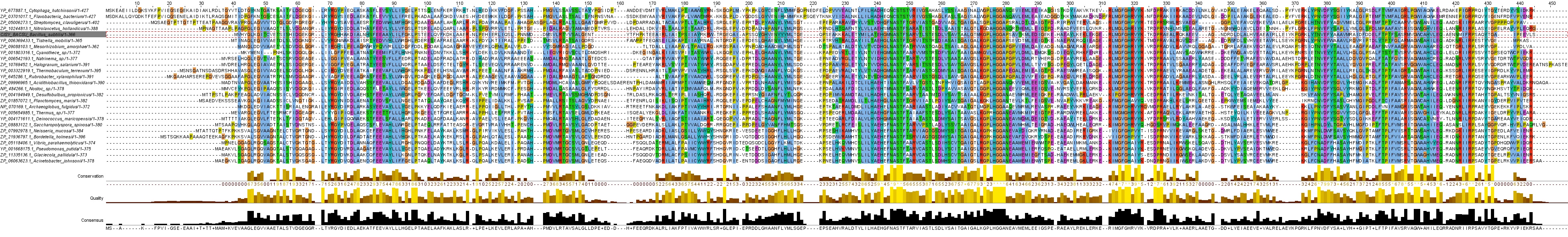
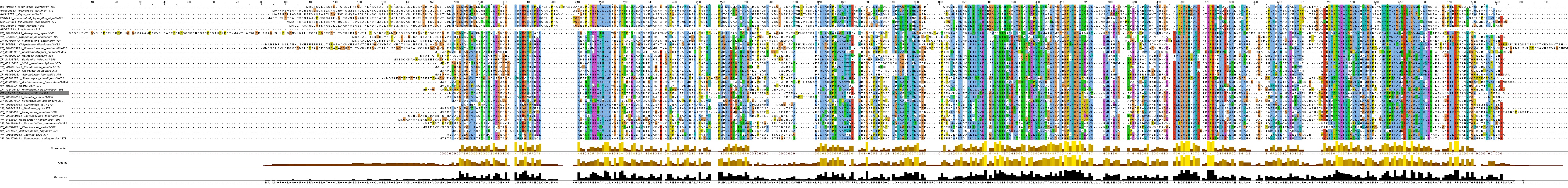
Множественное выравнивание
Создание репрезентативной выборки гомологов белка CISY_BACSU
Часть 1: Из ProcaryotaЗадача этого раздела - создать выборку гомологов белка, представляющих различные группы из Procaryota. В поиске BLAST были исключены все эукариоты, а также представители филума Firmicutes, к которому относится B. subtilis.Так как BLAST не может чисто технически вывести все найденные гомологи до E-value, которое уже можно расценивать как подвергающее сомнению гомологию белков, поисковый запрос был разбит на несколько меньших запросов по крупным группам внутри Procaryota. Были сделаны отдельные запросы по каждому филуму, что, не уменьшая разнообразия результатов, позволяет облегчить работу сервера с большими объёмами данных. Данные о параметрах запросов и их результатах представлены ниже (Табл.1, Рис. 1). Все запросы проводились с пороговым E-value в 0.001, гомологи выбирались из разных родов и субгрупп в филумах Procaryota. Количество выбранных из данной группы гомологов соответствует общему количеству найденного для этой группы исходного материала. ИТОГ: Составлена выборка из 26 последовательностей гомологов (включая исходный белок) из Procaryota. |
 Рис. 1 Распределение гомологов по группам бактерий (репрезентативность выборки) |
Таблица 1. Параметры и результаты поиска для создания выборки гомологов
| Группа из Procaryota | Количество находок | Взято в выборку | Идентификаторы |
| a-proteobacteria | 576 | 2 | YP_006369433.1, ZP_09088103.1 |
| b-proteobacteria | 572 | 2 | ZP_07992978.1, ZP_21936797.1 |
| g-proteobacteria | 1220 | 5 | ZP_11335136.1, ZP_09996965.1, ZP_05118406.1 YP_001668175.1, ZP_06063623.1 |
| d-proteobacteria | 96 | 1 | YP_004194949.1 |
| Actinobacteria | 814 | 3 | YP_645286.1, ZP_08883122.1, ZP_05006273.1 |
| Cyanobacteria | 114 | 2 | YP_001803316.1, NP_484266.1 |
| Bacteroidetes/Chlorobi group | 199 | 2 | YP_677887.1, ZP_03701017.1 |
| Archeae | 201 | 3 | YP_006542193.1, ZP_10769452.1, NP_070169.1 |
| Все остальные | 472 | 5 | YP_003322919.1, YP_005654985.1, ZP_10244813.1, YP_004171611.1, ZP_01857072.1 |
| Итого: | 4264 | 25 |
Последовательности выбранных белков доступны по ссылке: FASTA-файл
Часть 2: Гомологи белка CISY_BACSU среди эукариот
В данном разделе был проведён поиск BLAST по Eucaryota (параметры: E-value threshold = 0.001, база RefSeq). Данные по распределению гомологов среди групп Eucaryota представлены на рисунке 2. Всего обнаружено 889 белков, удовлетворяющих условиям поиска. Среди них выбраны по нескольку из разных групп, для каждого проведена проверка на предмет того, принадлежит ли белок геному митохондрии/хлоропласта или же закодирован в ядре.Филогенетическое дерево распределения гомологов белка среди эукариот доступно по ссылке: Рисунок 2
Всего для выборки взято двенадцать белков из эукариот, их последовательности доступны по ссылке: FASTA-файл .
Анализ расположения генов показал, что 10 из 12 генов являются ядерными, а два оставшихся (из Aspergillus niger и Toxoplasma gondii) - митохондриальными.
Таблица 2. Итоговый набор гомологов белка из разных доменов, вошедших в выборку. Во второй колонке в скобках указано название подцарства/субфилума. Золотым цветом выделены митохондриально закодированные белки. Оранжевым выделен исходный белок CISY_BACSU.
| Домен | Филум/Царство | Организм | Белки |
| Eukaryota | Alveolata (Ciliophora) | Tetrahymena pyriformis | BAF79583.1 |
| Alveolata (Apicomplexa) | Babesia bovis | XP_001610210.1 | |
| Alveolata (Apicomplexa) | Toxoplasma gondii (mitoch.) | ABP37863.1 | |
| Amoebozoa (Mycetozoa) | Dictyostelium discoideum | XP_647596.1 | |
| Opisthokonta (Metazoa) | Schistosoma japonicum | CAX73475.1 | |
| Opisthokonta (Metazoa) | Bos taurus | ABD77273.1 | |
| Opisthokonta (Metazoa) | Homo sapiens | AAC25560.1 | |
| Opisthokonta (Fungi) | Aspergillus niger | XP_001389414.2 | |
| Opisthokonta (Fungi) | Aspergillus niger (mitoch.) | P51044.1 | |
| Viridiplantae (Streptophyta) | Oryza sativa | AAG28777.1 | |
| Viridiplantae (Streptophyta) | Arabidopsis thaliana | AAM62868.1 | |
| Viridiplantae (Chlorophyta) | Chlamydomonas reinhardtii | XP_001695571.1 | |
| Eubacteria | Actinobacteria | Rubrobacter xylanophilus | YP_645286.1 |
| Actinobacteria | Saccharopolyspora spinosa | ZP_08883122.1 | |
| Actinobacteria | Streptomyces clavuligerus | ZP_05006273.1 | |
| Cyanobacteria (Chroococcales) | Cyanothece sp. | YP_001803316.1 | |
| Cyanobacteria (Nostocales) | Nostoc sp. | NP_484266.1 | |
| Proteobacteria (a) | Tistrella mobilis | YP_006369433.1 | |
| Proteobacteria (a) | Mesorhizobium amorphae | ZP_09088103.1 | |
| Proteobacteria (b) | Neisseria mucosa | ZP_07992978.1 | |
| Proteobacteria (b) | Bordetella holmesii | ZP_21936797.1 | |
| Proteobacteria (g) | Pseudomonas putida | YP_001668175.1 | |
| Proteobacteria (g) | Acinetobacter johnsonii | ZP_06063623.1 | |
| Proteobacteria (g) | Glaciecola pallidula | ZP_11335136.1 | |
| Proteobacteria (g) | Acidithiobacillus thiooxidans | ZP_09996965.1 | |
| Proteobacteria (g) | Vibrio parahaemolyticus | ZP_05118406.1 | |
| Proteobacteria (d) | Desulfobulbus propionicus | YP_004194949.1 | |
| Firmicutes | Bacillus subtilis | P39119 | |
| Bacteroidetes/Chlorobi group | Cytophaga hutchinsonii | YP_677887.1 | |
| Bacteroidetes/Chlorobi group | Flavobacteria bacterium | ZP_03701017.1 | |
| Thermobaculum | Thermobaculum terrenum | YP_003322919.1 | |
| Deinococcus-Thermus | Thermus sp. | YP_005654985.1 | |
| Chloroflexi | Nitrolancetus hollandicus | ZP_10244813.1 | |
| Deinococcus-Thermus | Deinococcus maricopensis | YP_004171611.1 | |
| Planctomycetes | Planctomycetes maris | ZP_01857072.1 | |
| Archaea | Euryarchaeota (Halobacteria) | Natrinema spPlanctomyces. | YP_006542193.1 |
| Euryarchaeota (Halobacteria) | Halogranum salarium | ZP_10769452.1 | |
| Euryarchaeota (Archaeglobales) | Archaeoglobus fulgidus | NP_070169.1 |
Множественное выравнивание гомологов белка CISY_BACSU
C помощью программы MUSCLE были построены три множественных выравнивания: изучаемый белок и прокариотические гомологи, изучаемый белок и эукариотические гомологи, вся выборка целиком (см. рис. 3,4,5 соответственно). На рисунках серым выделен CISY_BACSU, {!} чтобы просмотреть увеличенную версию изображения, кликните по интересующему рисунку правой кнопкой мыши, затем увеличьте. Раскраска выравниваний осуществлена по стандартной цветовой схеме Clustalx.Затем был проведён анализ выравнивания в программе Jalview. С последовательностью CISY_BACSU был проассоциирован PDB-файл со вторичной структурой белка, что позволило выделить остатки, которые ранее были определены как функционально важные уже на выравнивании.
К исходному выравниванию были добавлены три новых строки аннотации:
- SECONDARY: отражает информацию об участках вторичной структуры, таких как бета-тяжи и альфа-спирали.
- LIGANDS: Показывает, какие участки участвуют в связывании лигандов - кофермента А и молекулы цитрата. Такие участки помечены буквой "L".
- BLOCKS: в этой строке аннотации отмечены остатки (в строке BLOCKS стоит буква "B"), формирующие блоки - более или менее консервативные участки, хорошо выравниваемые между гомологами.
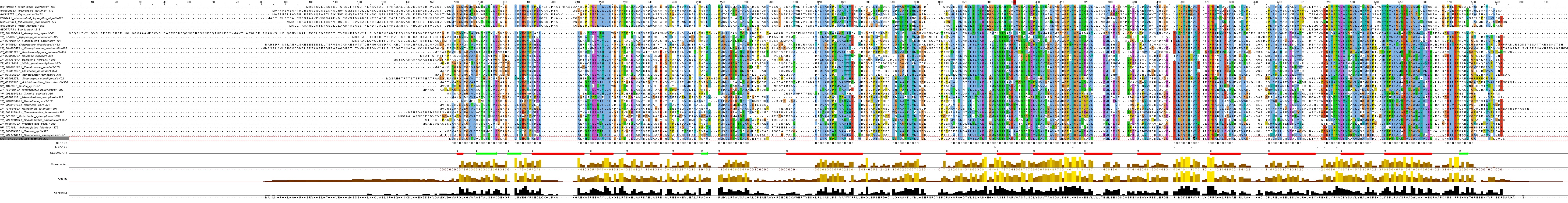 |
| Рис. 6 Аннотированное выравнивание всех гомологов из выборки (комментарии см. в тексте) |
Результаты анализа множественного выравнивания гомологов белка CISY_BACSU
General: Характеризуя выравнивание в целом, можно отметить, что консервативных, хорошо выравненных участков сравнительно много, причём хорошо выравниваются гомологи между всеми тремя доменами живых организмов, а не только среди прокариот или эукариот. Почти все белки имеют сходную длину, что за редким исключением позволяет избежать невыравненных "хвостов".Secondary: Все участки с определённой вторичной структурой входят в состав блоков, хотя консервативность в этих участках может быть в целом и не очень большой. Надо отметить, что блоки затрагивают участки, превосходящие по размерам спирали и тяжи, т. е. хорошо выравниваются даже фрагменты с неупорядоченной вторичной структурой. Выделение блоков я проводил, ориентируясь на строки аннотации conservation и quality, а также на группы последовательно идущих более или менее строго консервативных остатков, которые формируют "скелет" такого блока.
Среди элементов вторичной структуры обнаружены две альфа-спирали, которые прерываются колонками гэпов. Причём интересно, что такая ситуация связана с тем, что у эукариотических гомологов в этом месте есть дополнительный участок, который и даёт колонку гэпов.
Ligands: На рисунке 7 показан белок, на котором чёрным отмечены остатки, участвующие в формировании связей с лигандами. Все белки-гомологи катализируют одну реакцию, поэтому резонно предположить, что такие остатки будут консервативны среди гомологов, так как цитрат - продукт реакции, а кофермент А - субстрат. Так ли это в реальности? В таблице 3 приведена информация по тому, насколько консервативен тот или иной остаток. Консервативность (в процентах) показывает, в каком проценте последовательностей гомологов выравнен такой же остаток, как и в CISY_BACSU.
Таблица 3. Консервативность остатков, участвующих в связывании лиганда
| Остаток | Asp 181 | His 213 | Arg 246 | Arg 253 | Lys 297 | Arg 300 | Asn 305 |
| Консервативность (%) | 97 | 100 | 43(*) | 57 | 23 | 40(**) | 100 |
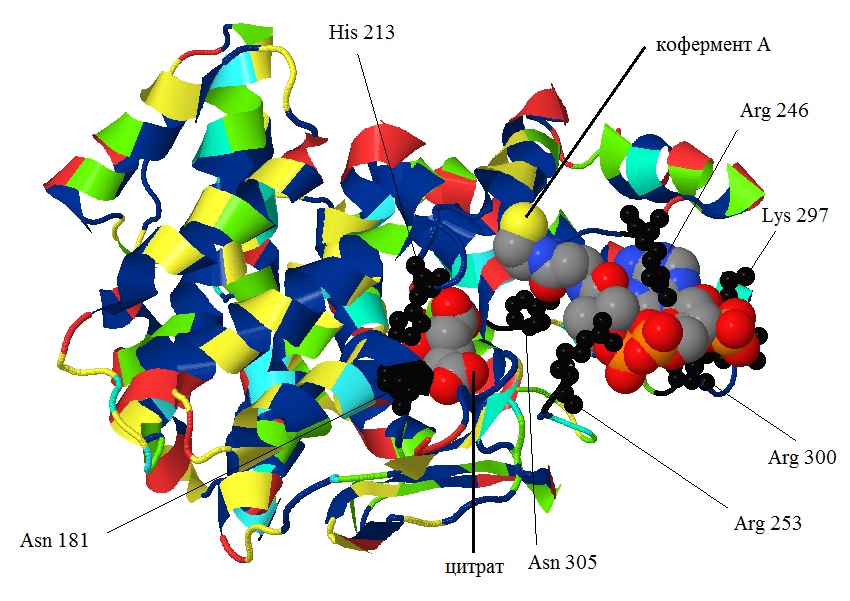 |
| Рис. 7 CISY_BACSU: чёрным показаны остатки, участвующие во взаимодействии с лигандами; лиганды показаны атомными радиусами (cpk); остатки в белке покрашены в соответствии с химической природой радикала (описание см. в практикуме 5). |
Дата последнего обновления: 27.05.2013
© Dmitry Travin, 2012