EMBOSS
Программа getorf пакета EMBOSS
C помощью команды entret embl:D89965 получаем файл (ССЫЛКА) с записью, из которой узнаём, что это последовательность матричной РНК, полученная в эксперименте из желудка крысы Rattus norvegicus.Используя для d89965.entret интересующую нас команду getorf -minsize 30 -table 0 -find 1 получаем набор всех рамок считывания (считая рамкой последовательность между старт и стоп кодонами) длиной более 30 нуклеотидов. При таком варианте записи получаем рамки в транслированном виде, используется стандартные генетический код. Получено всего 9 рамок различной длины. Из них пятая совпадает с той последовательностью, что приведена в записи EMBL для белка. В свою очередь запись EMBL ссылается на запись Swiss-Prot с идентификатором P0A7B8 (ССЫЛКА). Эта запись соответствует белку HSLV_ECOLI из кишечной палочки. Приведённая в записи последовательность соотносится при этом с девятой из найденных рамок считывания.
Как можно объяснить наблюдаемую ситуацию?
Можно предположить следующее: во время эксперимента была сиквенирована последовательность бактериального гена, а такое вполне возможно, так как E.coli населяет различные участки кишечного тракта. Затем (например, по сходству последовательности одной из рамок считывания) было предположено, что это именно такой крысиный ген. Как известно, EMBL - база архивная, в которой за достоверность информации отвечает только тот, кто её туда поместил. В то же время Swiss-Prot база реферируемая, а потому данным из неё можно доверять значительно больше. Таким образом, наблюдаем ошибку интерпретации результатов сиквенирования (по неверной ORF).
Файлы-списки
- Командой seqret sw:adh*_* получаем все последовательности алкогольдегидрогеназ из Swiss-prot (ССЫЛКА).
- C помощью команды infoseq -only -usa получаем список всех универсальных адресов USA (ССЫЛКА).
- Получаем теперь список только из адресов, относящихся к выбранному списку организмов: команда grep adh.txt -f organisms.txt >> selected.txt, таким образом мы осуществляем поиск внутри одного файла по второму и в третий записываем найденные адреса. Имеем файл с выбранными адресами: (ССЫЛКА).
- Теперь наконец получим файл с последовательностями выбранных в предыдущем пункте дегидрогеназ: seqret @selected.txt final.fasta. Такой ввод (используя @) позволят использовать файл selected.txt как список тех белков, последовательности которых, будут в файле final.txt (ССЫЛКА)
EnsEMBL
EnsEMBL - геномный браузер, позволяющий визуализировать информацию о геномах человека и животных (в основном позвоночных).- Сначала я попробовал поиск гена просто по идентификатору (TBA3E_HUMAN) с использованием строки search, получил 2 результата: транскрипт этого гена и сам ген как разные записи. Если выбрать запись гена, то на страничке получаешь краткую о нём информацию. в том числе варианты сплайсинга, расположение в геноме и источники, где всё это описано.
- Если перейти по ссылке Go to Region in Detail то можно получить подробную карту региона, где располагается искомый ген (Рис 1,2).

Рис. 1 Расположение участка с искомым геном во второй хромосоме человека. 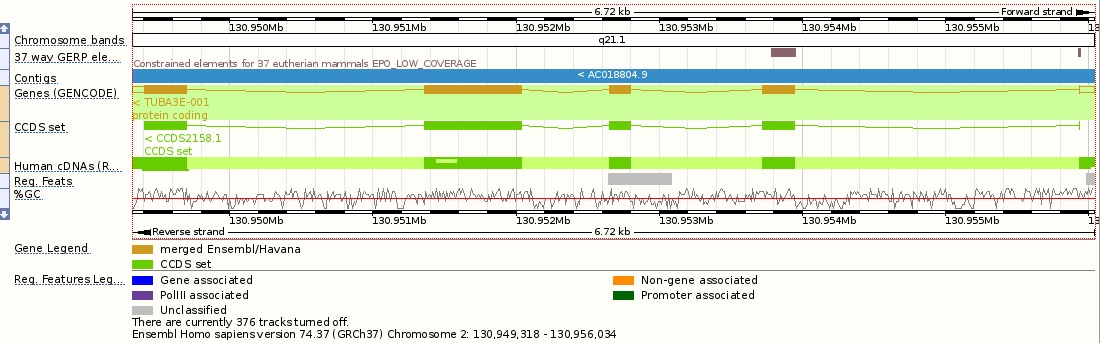
Рис. 2 Cхема расположения интронов и экзонов в гене по различным данным: CCDS set -consensus CDS, проект по выявлению основных кодирующих учасков генома человека и мыши, аннотированных с большой точностью, RefSeq/ENA, GENCODE. - Кроме того со странички гена возможен переход к скачиванию различного рода информации об этом гене (меню Export Data): я сохранил отдельным файлом последовательность всего гена + последовательность каждого интрона/экзона (ССЫЛКА). Возможно скачивание вразличных форматах, есть меню, в котором определяется, что будет входить в итоговый файл, можно захватить например сколько-то нукеотидов вокруг гена по своему желанию.
-
Теперь попробую инструменты поиска BLAST/BLAT в браузере, используя известную последовательность гене субъединицы тубулина. В выдаче программы хочется отметить то, что сразу видно расположение всех найденных участков по геному (Рис 3).Кроме того удобно, что таблица, в которой выводятся результаты, можно форматировать по желанию добавляя и убирая столбцы.
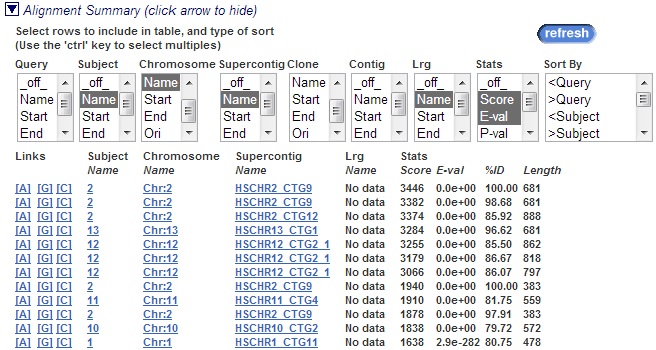
Рис. 3 Расположение находок BLAT в геноме человека, лучшая находка (наш ген) обведён в рамочку. Рис. 4 Фрагмент таблицы с выводом результатов, показана "шапка" форматирования. - Отдельно скажу про сервис доступа к информации по каждому из организмов в Ensembl: если перейти на страничку какого-либо вида, то там доступны такие опции как:броузер кариотипа с возможностью быстрого перехода к отдельным участкам хромосом, примеры построения генетических деревьев от этого организма (Рис 5), и некоторые другие.
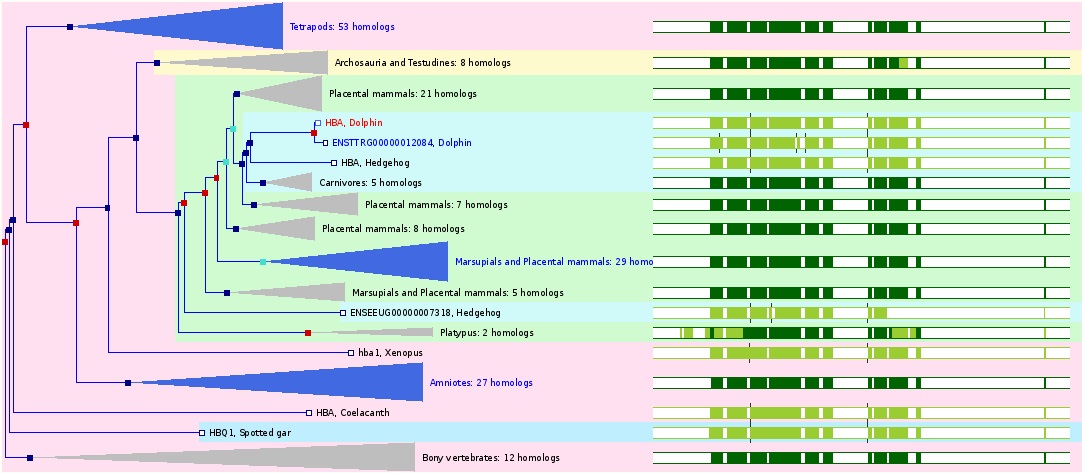
Рис. 5 Пример генетического дерева, построенного по генам дельфина и других животных, информация о геномах которых содержится в базе.
Дата последнего обновления: 09.12.2013
© Dmitry Travin, 2013