Профили (пакет HMMER)
Построения профиля
В качестве выборки, по которой будет строиться профиль, были выбраны последовательности, содержащие домен EF-hand_4 в архитектуре EF-hand_4+Dynamin и принадлежащие к подтаксону Viridiplantae. Для построения профиля выбраны именно эти последовательности, так как они хорошо группируются на филогенетическом дереве, образуя единую кладу (см. практикум Эволюционные домены). Последовательности доменов приведены в файле Выборка для профиля. 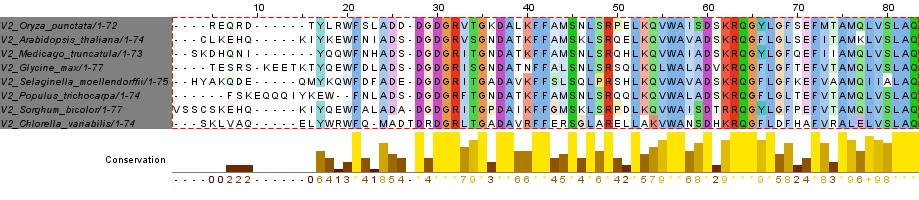 |
| Рисунок 1. Выравнивание доменов EF-hand_4 из последовательностей архитектуры EF-hand_4+Dynamin подтаксона Viridiplantae |
По полученному выравниванию построен HMM (Hidden Markov Model)-профиль с помощью программы hmm2build, затем он был откалиброван командой hmm2calibrate. Полученный профиль: ПРОФИЛЬ.
Проверка построенного профиля
С помощью команды hmm2search был проведён поиск по полученному профилю. В качестве документа, где производился поиск, использовался файл в FASTA-формате со всеми последовательностями белков из Uniprot, содержащих рассматриваемый домен. Выдача программы была перенаправлена в файл: Результаты_поиска.Теперь проанализируем полученные результаты. Из выдачи программы составим список из идентификаторов последовательностей белков и полученных для данной последовательности значений E-value: Список_результаты_поиска. C полученным списком будем сравнивать список из всех идентификаторов последовательностей из Viridiplantae с данной архитектурой: Список_сравнения. Для рассмотрения выбран порог E-value, равный 1e-8, рассматривались находки только с меньшим E-value. Из 107 белков, входящих в "золотой стандарт", было обнаружено 51 (в пределах рассматриваемого E-value), всего же белков, найденных в пределах такого E-value, 515. Таблица приведена по ссылке: Таблица.
- TP (true positives)=51
- FP (false positives)=515-51=464
- FN (false negatives)=108-51=57
Дата последнего обновления: 23.05.2014
© Dmitry Travin, 2014