Реконструкция деревьев по нуклеотидным последовательностям.
Деревья, содержащие паралоги.
Построение дерева по нуклеотидным последовательностям
В данном задании мы строим филогенетическое дерево бактерий, с которыми работали и в прошлых двух практикумах, но с использованием методов, использующих в качестве входных данных нуклеотидные последовательности. К таким методам относятся Neighbor-Joining, Minimum evolution, Maximum likelihood. В качестве последовательности используем 16S РНК бактерий.В таблице 1 для каждой из рассматриваемых бактерий приведены: AC записи EMBL, в которой обнаружена последовательность 16S rRNA, координаты этой РНК в этой записи, а также то, на прямой или обратной цепи она расположена.
Таблица 1. Информация о полученных записях, содержащих последовательность 16S РНК
| Бактерия | Мнемоника | AC записи EMBL | Координаты | Цепь |
| Bacillus anthracis | BACAN | CP002183 | 9750..11308 | Прямая |
| Clostridium botulinum | CLOB1 | CP000726.1 | 48468..49969 | Прямая |
| Enterococcus faecalis | ENTFA | AE016830 | 248466..249987 | Прямая |
| Geobacillus kaustophilus | GEOKA | BA000043.1 | 10421..11973 | Прямая |
| Lactobacillus delbrueckii | LACDA | CP002341.1 | 33581..35141 | Прямая |
| Listeria monocytogenes | LISMO | CP007492.1 | 242732..244284 | Прямая |
| Streptococcus pneumoniae | STRPN | CP006844.1 | 1696703..1698247 | Обратная |
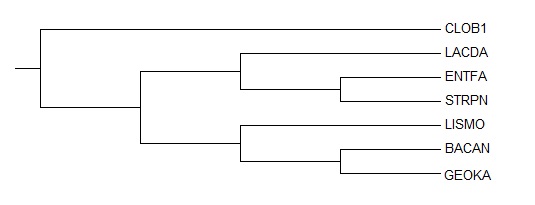
Все полученные последовательности 16S РНК были помещены в файл: FASTA и переименованы в соответствии с мнемоникой конкретного вида. Затем последовательности были выравнены программой Muscle (использовался web-интерфейс). Выравнивание было импортировано в программу MEGA, в которой было построено филогенетическое дерево методом Neighbour joining. На рисунке 1 представлено сравнение правильного дерева и построенного по последовательности 16S РНК.
 |
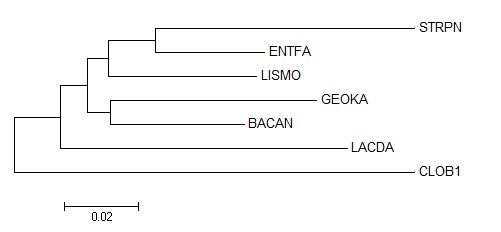 |
| Рисунок 1. Правильное дерево (слева) и дерево, построенное методом Neighbour joining по последовательностям 16S РНК (справа). | |
Реконструированное дерево с правильным не совпадает. Из четырёх ветвей, присутствующих в правильном дереве, две присутствуют и в реконструированном по нуклеотидным последовательностям. Отсутствуют следующие ветви: {BACAN,LISMO,GEOKA}&{others} и {LACDA,ENTFA,STRPN}&{others}.
Вообще-то, исходя из того, что в нуклеотидных последовательностях всего четыре различных элемента алфавита (против 20 в белковых последовательностях), можно предположить, что основанные на них филогенетические деревья будут хуже реконструировать реальность. Это связано с большей вероятностью неправильного выравнивания, а также с тем, что в таких выравниваниях будут учитываться случайные молчащие мутации, которые никак не влияют на последовательность белка.
Построение и анализ дерева, содержащего паралоги
Сначала приведу определения:| Паралоги - последовательности, к разделению которых привело удвоение гена внутри одного организма (в результате хромосомной мутации). В качестве примера паралогов приводятся, например, гены различных форм гемоглобина человека (α, β, γ и др.). |
| Ортологи - последовательности, к разделению которых привел акт видообразования. Примером ортологичных генов могут быть гены гемоглобина β у человека и лемура. |
В данном разделе мы найдем гомологи белка CLPX_BACSU (ATP-dependent Clp protease) в рассматриваемых бактериях. На дереве, построенном по таким гомологам, попробуем указать на два типа эволюционных событий: 1) дупликацию гена; 2) разделение путей эволюции белков в результате видообразования.
С помощью команды blastp (порог E-value 0,001) среди всех белков всех бактерий, упомянутых в первом практикуме, были найдены гомологи, а затем отобраны те, которые относятся к рассматриваемому списку из семи бактерий. полная выдача blastp: Out_blast. Далее были отобраны те, которые относятся к нашим семи бактериям: Out_select. Для 31 белка были получены последовательности в fasta-формате: Protein_seq.
Все полученные последовательности были выравнены с помощью программы Muscle, затем построено дерево гомологов методом Neighbour joining.
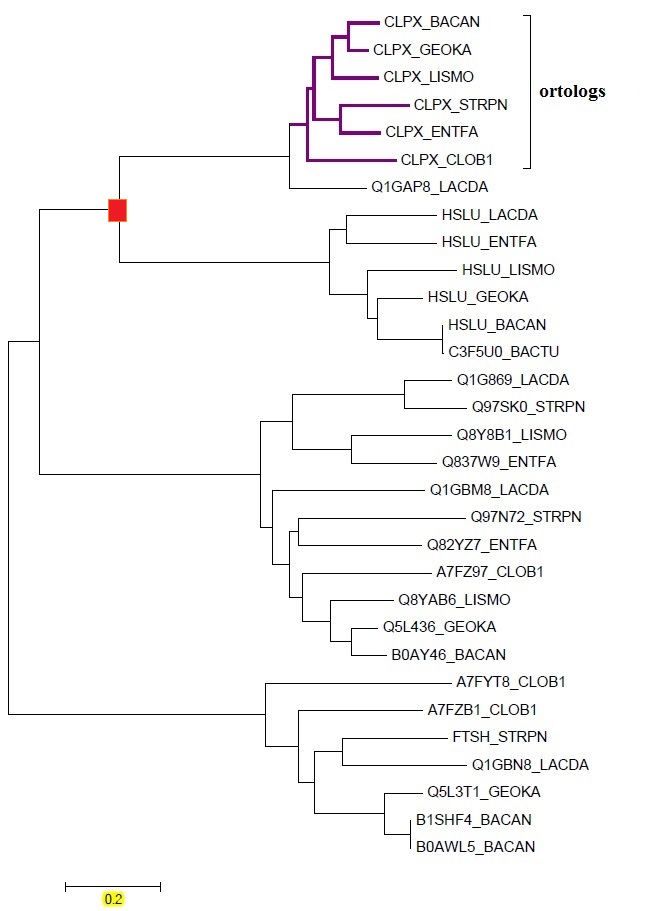 |
| Рисунок 2. Дерево, построенное по последовательностям гомологов белка CLPX_BACSU из семи рассматриваемых бактерий. Квадратной скобкой выделен пример ветви дерева, состоящей из белков-ортологов. |
Пример ортологов: CLPX_LISMO, CLPX_BACAN, CLPX_LACDA, CLPX_CLOB1, CLPX_GEOKA, CLPX_ENTFA и CLPX_STRPN (показаны на рисунке 2 скобкой).
Пример паралогов: B1SHF4_BACAN и B0AWL5_BACAN (образовались в результате дупликации гена).
В качестве примера разделения белков в результате видообразования: разделение на ветвь, содержащую CLPX_X, и ветвь с белками HSLU_X (точка дивергенции обозначена на рисунке красным квадратиком).
Дата последнего обновления: 17.05.2014
© Dmitry Travin, 2014