Особенности мембранных белков
В данном практикуме я буду работать с мембранными белками. Мне достаётся переносчик формиат-аниона FocA из Escherichia coli (PDB: 3KCU).
Таблица 1. Описание трансмембранных белков с известной 3D структурой
Обращает на себя внимание тот факт, что в β-барреле на один трансмембранный участок приходится значительно меньше остатков аминокислот, чем в α-спиральных белках, что в принципе и понятно, исходя из вторичной структуры спирали и плоского β-листа.
Таблица 2. Описание структуры переносчика формиат-аниона FocA (идентификатор PDB: 3KCU)
Расшифровка TC-кода: 1 - каналы/поры, 1.A - каналы, состоящие из α-спиралей, 1.A.16 - cемейство каналов-транспортёров нитрита/формиата (FNT family).
Информация о третичной структуре белка приведена в статье:
Дополнительная информация о белке FocA:
Для гомолога белка FocA из бактерии Vibrio cholerae была получена выдача программы TMHMM (осуществляет предсказание трансмембранных α-спиралей по последовательности белка).
К выравниванию добавлена аннотация TM_PREDICTED, где буквами М обозначены позиции, входящие в состав трансмембранных спиралей, согласно выдаче программы TMHMM. Итоговый файл формата JAR доступен по ссылке: ВЫРАВНИВАНИЕ
Обсуждение результатов и выводы:
База данных OPM (Orientations of Proteins in Membranes) и примеры трансмембранных белков
Известны две основные формы трансмембранных структур, которые наблюдаются у большинства белков, - это α-спирали и β-баррели ("бочонки"). В таблице 1 приведены некоторые параметры трансмембранных структур для ряда белков с известной третичной структурой (по материалам базы данных)Таблица 1. Описание трансмембранных белков с известной 3D структурой
| PDB код | Тип
(спираль, баррель) |
Тип мембраны (внутренняя или внешняя, организм, органелла) |
Толщина гидрофобной части мембраны (Å) | Медиана числа остатков в одном трансмембранном участке |
| 4DKL | Спираль | Плазматическая мембрана Mus musculus (Опиоидный рецептор типа mu) | 32.0 ± 1.0 | 24 |
| 1M0L | Спираль | Мембрана архей Halobacterium salinarum (Бактериородопсин) | 31.8 ± 1.1 | 22 |
| 1KQF | Спираль | Внутренняя мембрана G--бактерий из Escherichia coli (формиатдегидрогеназа) | 33.7 ± 1.1 | 23 |
| 3QRA | Баррель | Внешняя мембрана G--бактерий из Yersinia pestis (белок локуса прикрепления и инвазии) | 25.2 ± 1.4 | 7,5 |
| 1UUN | Баррель | Мембрана G+-бактерий из Mycobacterium smegmatis (порин MspA) | 40.7 ± 2.1 | 8 |
| 4BUM | Баррель | Внешняя мембрана митохондрий Danio rerio (канал VDAC-2) | 23.4 ± 0.9 | 7 |
Обращает на себя внимание тот факт, что в β-барреле на один трансмембранный участок приходится значительно меньше остатков аминокислот, чем в α-спиральных белках, что в принципе и понятно, исходя из вторичной структуры спирали и плоского β-листа.
Создание выборки гомологов белка FocA
Был проведён blasp по последовательности белка 3KCU в базе RefSeq. Получено очень много гомологов - транспортёров формиата из различных организмов. Выборка формировалась на основе последовательностей из разных таксонов: как бактерии, так и эукариоты (по ним был проведён отдельный поиск blastp), имеющих большое (близкое к 100%) покрытие по последовательности и низкие (≤ 1e-30). Последовательности 17-ти белков, вошедших в итоговую выборку, а также исходного белка FocA, доступны по ссылке: выборка.Анализ структуры трансмембранного канала-переносчика формиата FocA
В таблице 1 приведена информация о белке FocA, полученная из баз данных OPM и TCDB (Transporter classification database).Таблица 2. Описание структуры переносчика формиат-аниона FocA (идентификатор PDB: 3KCU)
| PDB ID | Организм | Тип мембраны | TC-код | Наклон спиралей к нормали | Количество трансмембранных спиралей |
| 3KCU | Escherichia coli | Внутренняя мембрана G--бактерий | 1.A.16 | 0 ± 0º | 7 (на каждый из семи мономеров) |
Расшифровка TC-кода: 1 - каналы/поры, 1.A - каналы, состоящие из α-спиралей, 1.A.16 - cемейство каналов-транспортёров нитрита/формиата (FNT family).
Информация о третичной структуре белка приведена в статье:
| Wang Y., Huang Y., Wang J., Cheng C., Huang W., Lu P., Shi Y. (2009). Structure of the formate transporter FocA reveals a pentameric aquaporin-like channel. Nature, 462(7272), 467–72. PubMed |
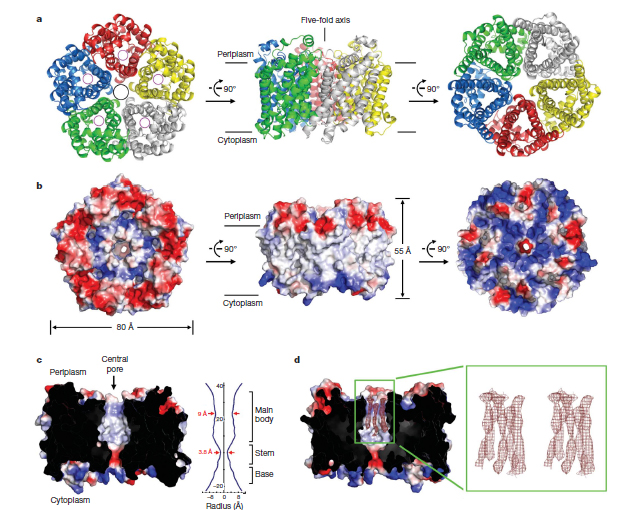 |
Рисунок 1. Структура комплекса FocA (из статьи PMID: 19940917). а: Показаны спирали в составе пентамерного комплекса. b: Раскраска по заряду на поверхности белка (переплазматическая часть заряжена отрицательно - красный цвет, внутренняя - положительно - синий) → работает правило "positive inside" ☺. c: Разрез через белок, показана пора, заряд на поверхности, образующей пору, размеры поры. |
Дополнительная информация о белке FocA:
- Обнаружен в археях, бактериях, водорослях, грибах и некоторых одноклеточных паразитах.
- Переносит короткоцепочечные кислоты, такие как формиат, непроницаем для воды.
- По структуре представляет симметричный пентамерный комплекс, в центре которого формируется пора.
Анализ множественного выравнивания трансмембранных белков
Было проведено выравнивание полученной выборки гомологов с помощью программы Muscle (использовался web-интерфейс). Результат доступен по ссылке: выборка_выров. В выравнивании у трёх белков были удалены сильно выступающие фрагменты, которые ни с чем не выравниваются и не участвуют в образовании трансмембранных структур. К выравниванию добавлена аннотация TM_REAL, в которой буквами M обозначены позиции, входящие в трансмембранные α-спирали белка FocA, для которого известна 3В-структура. Кроме того, с последовательностью FocA была проассоциирована его третичная структура.Для гомолога белка FocA из бактерии Vibrio cholerae была получена выдача программы TMHMM (осуществляет предсказание трансмембранных α-спиралей по последовательности белка).
# WP_001896847.1_Vibrio_cholerae Length: 280 # WP_001896847.1_Vibrio_cholerae Number of predicted TMHs: 6 # WP_001896847.1_Vibrio_cholerae Exp number of AAs in TMHs: 140.35506 # WP_001896847.1_Vibrio_cholerae Exp number, first 60 AAs: 22.46738 # WP_001896847.1_Vibrio_cholerae Total prob of N-in: 0.99366 # WP_001896847.1_Vibrio_cholerae POSSIBLE N-term signal sequence WP_001896847.1_Vibrio_cholerae TMHMM2.0 inside 1 32 WP_001896847.1_Vibrio_cholerae TMHMM2.0 TMhelix 33 55 WP_001896847.1_Vibrio_cholerae TMHMM2.0 outside 56 74 WP_001896847.1_Vibrio_cholerae TMHMM2.0 TMhelix 75 97 WP_001896847.1_Vibrio_cholerae TMHMM2.0 inside 98 115 WP_001896847.1_Vibrio_cholerae TMHMM2.0 TMhelix 116 138 WP_001896847.1_Vibrio_cholerae TMHMM2.0 outside 139 157 WP_001896847.1_Vibrio_cholerae TMHMM2.0 TMhelix 158 180 WP_001896847.1_Vibrio_cholerae TMHMM2.0 inside 181 192 WP_001896847.1_Vibrio_cholerae TMHMM2.0 TMhelix 193 215 WP_001896847.1_Vibrio_cholerae TMHMM2.0 outside 216 253 WP_001896847.1_Vibrio_cholerae TMHMM2.0 TMhelix 254 276 WP_001896847.1_Vibrio_cholerae TMHMM2.0 inside 277 280
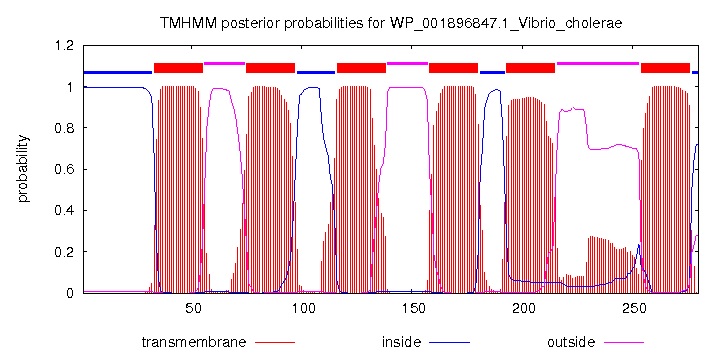 |
| Рисунок 2. Графическая визуализация в выдаче программы TMHMM. Красным показаны предсказанные позиции расположения трансмембранных участков. |
К выравниванию добавлена аннотация TM_PREDICTED, где буквами М обозначены позиции, входящие в состав трансмембранных спиралей, согласно выдаче программы TMHMM. Итоговый файл формата JAR доступен по ссылке: ВЫРАВНИВАНИЕ
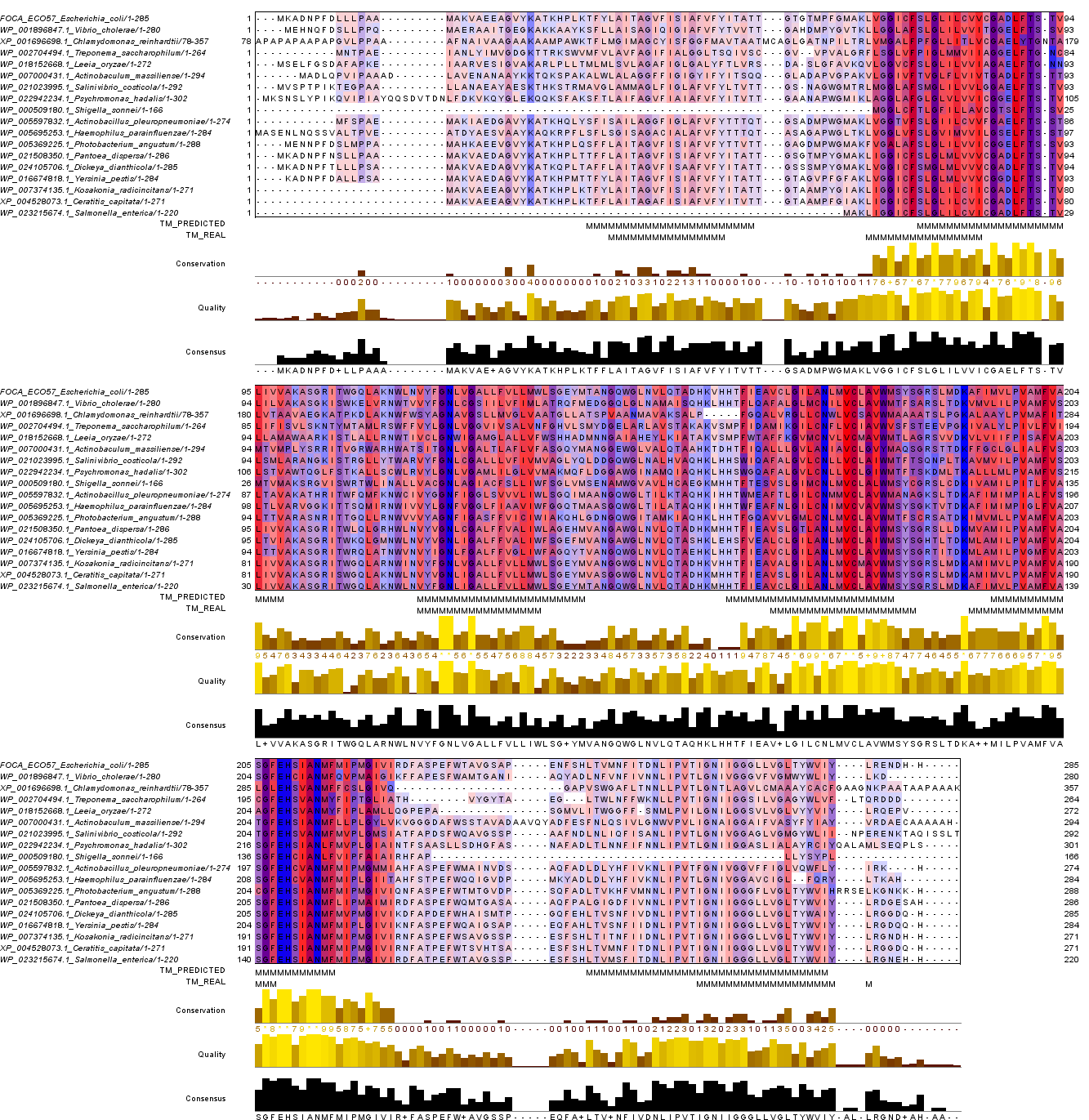 |
Рисунок 3. Выравнивание выборки гомологов белка FocA. Раскраска по гидрофобности с уровнем консервативности 15%. Показаны аннотации TM_REAL, TM_PREDICTED, буквы М индексируют трансмембранные участки. |
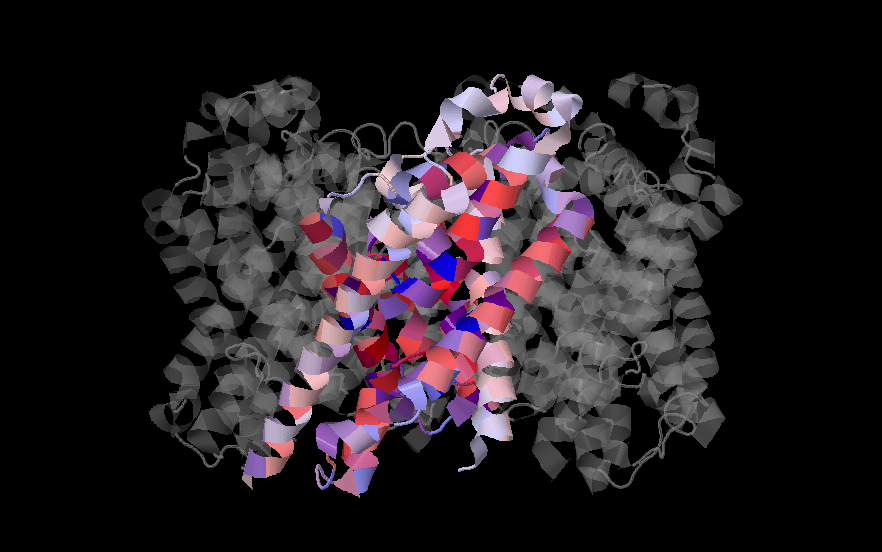 |
Рисунок 4. Третичная структура пентамерного комплекса FocA. Раскраска по гидрофобности с уровнем консервативности 15% для субъединицы А. Сверху периплазматическое пространство, цитоплазматическое внизу. |
Обсуждение результатов и выводы:
- На рисунке 3 видно, что позиции в трансмембранных спиральных участках консервативны, причём более сильно это проявляется в "центральных" по расположению в последовательности спиралях (2-4).
-
Таблица 3. Частоты встречаемости различных аминокислот в трансмембранных спиралях белка FocA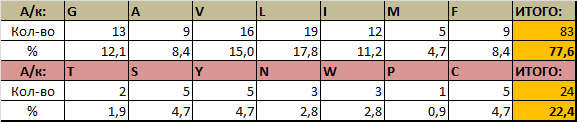
Из приведённой таблицы отчётливо видно, что в спиралях превалируют неполярные аминокислоты с гидрофобными радикалами (77%). - Участки между спиралями сравнительно менее консервативны по сравнению со спиралями, но всё же более консервативны, чем концевые участки белков, более близкие к концам последовательности межспиральные участки менее консервативны по сравнению с внутренними.
- В спиралях есть полярные и даже заряженные остатки (см. таблицу 3), предположительно, эти остатки могут играть роль в спиралях, формирующих в пентамерном комплексе центральную пору, через которую проходит полярная молекула транспортируемой кислоты (формиат, нитрит).
- Программа TMHMM предсказала шесть трансмембранных участков, что соответствует их числу в белке с известной третичной структурой. Во всех случаях они не совпадают идеально (но это же гомолог - такое вполне возможно), а имеют участок перекрывания.
Дополнительные задания:
1) Дыни имеют размер от 16-18 см в длину, 14 см в диаметре (примем дыню за шар диаметра 14 см), размеры эукариотической клетки - 10-50 мкм (пусть будет 30 в среднем). Толщина мембраны - 7-8 нм. Таким образом, для дыни: (8*10-9 * 14*10-2)/30*10-6 м = 3.73*10-5, т.е. 37*10-6 м - мембрану можно сравнить со слоем на поверхности дыни толщиной в одну клетку.Дата последнего обновления: 19.05.2013
© Dmitry Travin, 2014