Эволюционные домены
Выбор домена, архитектур и таксонов, их краткое описание
В качестве семейств доменов, с которым в дальнейшем будет вестись работа, было выбрано семейство доменов EF-hand_4.Краткая информация о домене:
- AC: PF12763
- ID: EF-hand_4 (Страница Pfam)
- Участвует в регуляции цитоскелета, связывает ион кальция (Ca2+).
- В базе Pfam записано 2526 последовательностей, содержащих этот домен, они относятся к 302 видам и 126 архитектурам (Страница архитектур Pfam)
Таблица 1. Характеристика выбранных для работы архитектур Pfam
| Архитектура | Число представителей | Описание второго домена |
| EF-hand_4 | 348 | Однодоменная архитектура, содержащая только домен EF-hand_4 |
| EF-hand_4 + Dynamin_N | 269 | PF00627: Домен, характерный для динаминов - белков, участвующих в процессах эндоцитоза в клетках эукариот. |
В качестве таксона, внутри которого будет проводиться сравнение, был выбран домен Eukaryota, субтаксоны это царства Viridiplantae и Metazoa.
Получение полного выравнивания для домена и формирование выборки для архитектур
Для всех последовательностей (2526), содержащих домен, получено выравнивание, доступное по ссылкам: Проект Pfam или FASTA-формат. В выравнивании произведена покраска по шаблону Clustalx со значением порога консервативности в 10%. Для трёх последовательностей (EHD2_MOUSE, REPS2_HUMAN и REPS1_MOUSE) с выравниванием были проассоциированы файлы с 3D-структурой белков в формате PDB, эти последовательности в выравнивании выделены зелёным цветом.Для всех белков Uniprot, в которых обнаружен рассматриваемый домен, составлена сводная таблица (ССЫЛКА), где для каждой последовательности указаны домены Pfam, в ней встречающиеся, обозначено их количество, а также указано общее число доменов на последовательность. Для каждого домена показано число последовательностей, в которых этот домен встречается. Отдельно жёлтым выделены столбцы тех доменов, которые входят в выбранные архитектуры наряду с рассматриваемым доменом.
Получение выборки последовательностей для дальнейшего анализа
Для двух рассмотренных архитектур были сделаны выборки (ССЫЛКА) последовательностей для дальнейшего выравнивания и построения филогенетических деревьев. Информация о составе выборок представлена в таблице 2. В выборку наряду с другими включена последовательность EHD1_HUMAN, для которой известна трёхмерная структура изучаемого домена.Таблица 2. Состав выборки последовательностей с доменом EF-4_hand по двум архитектурам
| Выборка | Число послед-тей | Metazoa | Viridiplantae | Другие подтаксоны |
| EF-hand_4 | 17 | 9 | 5 | 3 |
| EF-hand_4 + Dynamin_N | 17 | 7 | 8 | 2 |
| Объединённая выборка | 34 | 16 | 13 | 5 |
Для выбранных последовательностей выравненные домены, полученные выше, были отобраны и приведены в отдельное выборочное выравнивание: Проект Pfam или FASTA-формат. В нём отдельные белковые остатки раскрашены по консервативности с порогом в 10%, показано объединение в группы по типу архитектуры. Перед видовым названием каждого организма стоит индекс, буква(ы) в котором обозначают принадлежность к подтаксону (M-Metazoa, V-Viridiplantae, out-другие подтаксоны в роли outgroup), а цифры номер архитектуры (1 или 2).
 |
| Рисунок 1. Выравнивание выборки для двух архитектур домена EF-hand_4 |
Построение филогенетического дерева по выборке доменов из двух архитектур
Для полученной выше выборки последовательностей, содержащих домен EF-hand_4, в программе Mega было построено филогенетическое дерево методом максимального правдоподобия (Maximum Likelthood) с применением метода bootstrep (количество реплик - 100) для подтверждения качества ветвей. Скобочная форма (формат Newick) дерева доступна по ссылке: tree.nwk.Напомню, что последовательности на дереве, как и в выравнивании выше, закодированы следующим образом: для каждой последовательности в названии указана принадлежность к архитектуре (1 - EF-hand_4, 2 - EF-hand_4+Dynamin) и систематическое положение представителя, чьё латинское название приведено далее (M-Metazoa, V-Viridiplantae, out-другие подтаксоны Eucaryota).
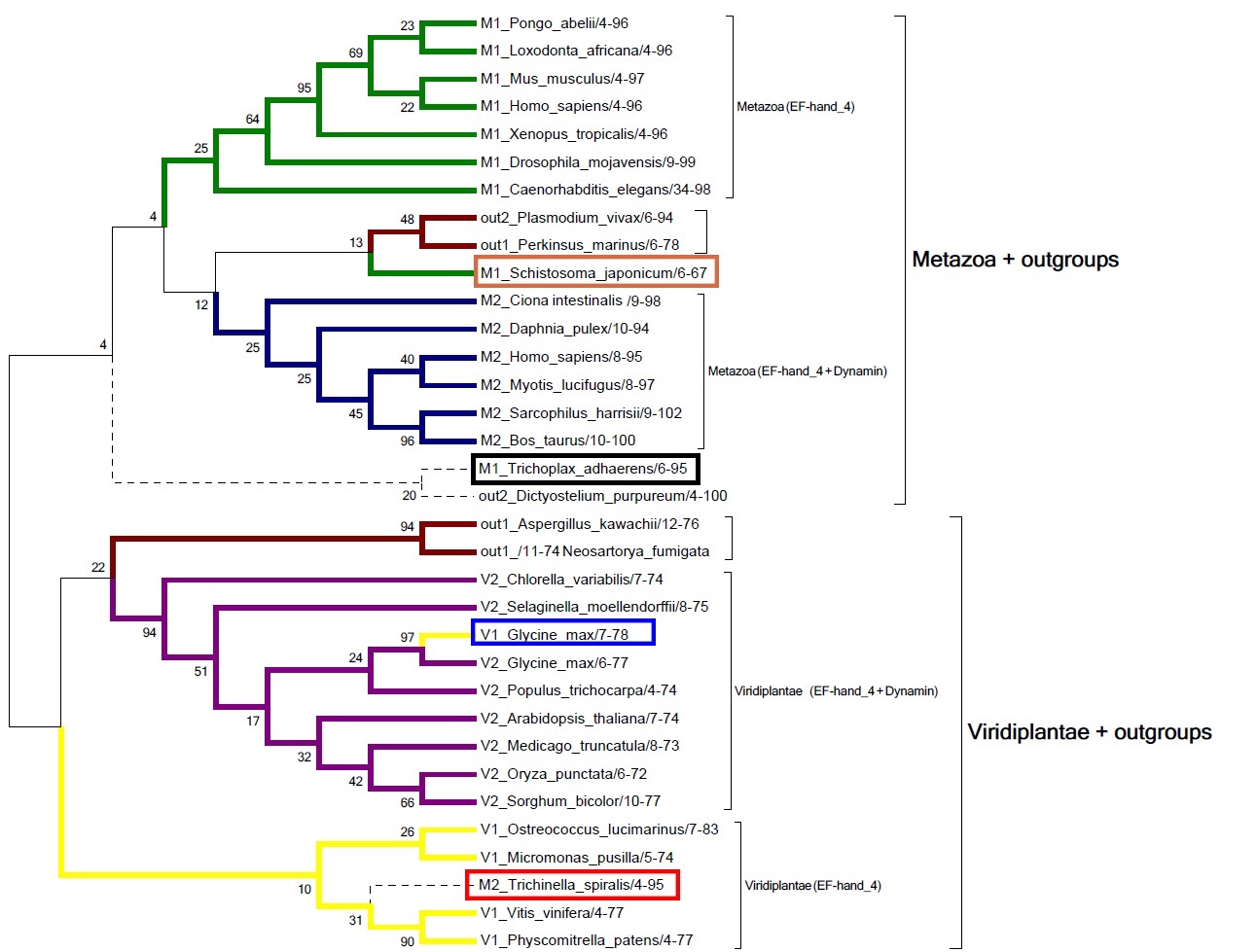 |
| Рисунок 2. Дерево последовательностей домена EF-hand_4, построенное по выборке двух архитектур методом Maximum Likelthood с bootstrep поддержкой (комментарии по раскраске см. в тексте). |
Анализ полученного филогенетического дерева:
- На дереве (рис. 2) видно, что в первую очередь последовательности, содержащие исследуемый домен, группируются по систематическому положению, а потом уже по архитектуре, в которую входит домен, то есть можно чётко выделить ветвь животных и ветвь растений (показаны на рисунке большими скобками). Это говорит о том, что появление архитектуры, содержащей EF-hand_4 совместно с доменом Dynamin, происходило независимо в линиях животных и растений. Такая архитектура не присутствовала у их общего предка.
- В рамках каждого подтаксона последовательности хорошо разделяются по тому, к какой они принадлежат архитектуре. Показаны меньшие скобки, соответствующие определённой архитектуре внутри подтаксона.
- Есть ряд последовательностей, чьё положение выбивается из общей картины, они выделены на рисунке 2 цветными рамочками. Рассмотрим каждый из таких случаев:
- Последовательность из Trichoplax вместе с последовательностью из Dictyostelium сгруппированы в outgroup ко всем остальным последовательностям из Metazoa. В целом это объяснимо, ведь Trichoplax - наиболее примитивный представитель многоклеточных животных, а Dictyostelium относится к Amoebozoa - таксону, группируемому вместе с царством Opisthokonta (включающему и Metazoa) в группу Uniconta.
- Расположение последовательности из трихинеллы (Trichinella spiralis) на ветви с последовательностями первой архитектуры из растений могу объяснить лишь ошибкой построения дерева.
- Две последовательности из сои (Glycine max) группируются вместе, несмотря на разные архитектуры в их составе. Это объясняется их принадлежностью к одному виду, что, видимо, вносит больший вклад, нежели независимая эволюция архитектур с доменом Dynamin и без него.
- Последовательность первой архитектуры из шистосомы (Schistosoma japonicum) группируется внутри ветви Metzoa с представителями outgroup, но с учётом низкой бутстреп-поддержки ранних этапов дивергенции внутри Metazoa, это можно отнести к ошибкам построения дерева.
Дата последнего обновления: 30.04.2013
© Dmitry Travin, 2014