 Последовательности, которые были использованы для построения
множественного выравнивания, довольно консервативны в общем, а не только
в зоне связывания лиганда. Поэтому, возможно, консервативность исследуемой
зоны так высока. Но если брать среднее значение консервативности по
последовательности, то консервативность зоны связывания будет больше
(в ней 18 из 22 остатков в той или иной степени консервативны, а во всем
выравнивании явно консервативна меньшая часть).
Последовательности, которые были использованы для построения
множественного выравнивания, довольно консервативны в общем, а не только
в зоне связывания лиганда. Поэтому, возможно, консервативность исследуемой
зоны так высока. Но если брать среднее значение консервативности по
последовательности, то консервативность зоны связывания будет больше
(в ней 18 из 22 остатков в той или иной степени консервативны, а во всем
выравнивании явно консервативна меньшая часть).
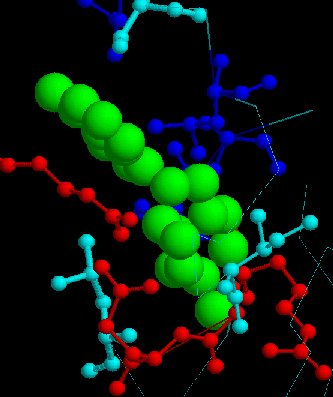
На рисунке справа изображена в большем масштабе зона контакта с одним из лигандов (CMP621).
Данная картина, но в несколько ином представлении, генерируется скриптом, который можно прочитать здесь .
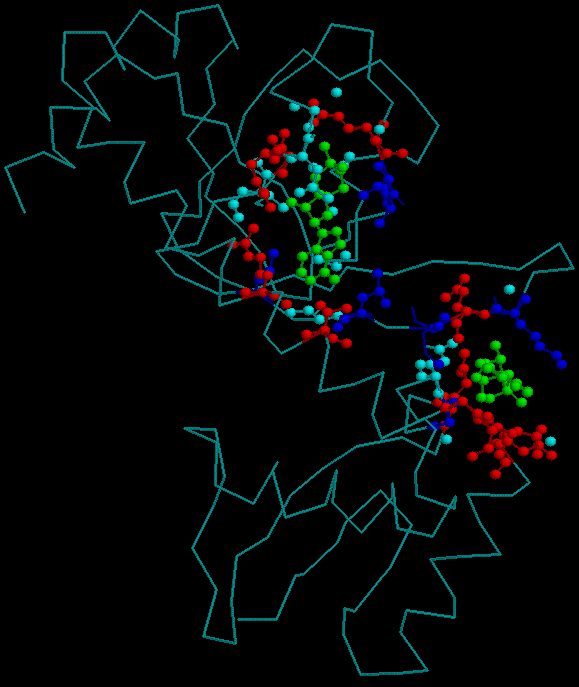
Из полученной картины следует, что зона контакта белка CRP_ECOLI c его лигандом CMP
высоко консервативна.
Это вполне логично, так как очень часто замены на неродственные аминокислоты в таких
участках может привести к потере способности связывания с лигандом, что означало бы
потерю функции белка. Сам алгоритм нахождения консервативных остатков путем поиска
ортологов моего белка наводит на мысль, что функции белков, последовательности
которые будут таким образом найдены и выровнены, должны быть одинаковы.