2. Выберем подсемейство доменов с двудоменной архитектурой: PF06439 - PF06283 (3-keto-disaccharide hydrolase - Trehalose utilisation)
Скачаем последовательности данного подсемейства 3. Выравняем эти последовательности в Jalview с помощью MAFFT.
4. Определим координаты начала и конца двудоменной архитектуры (N-конец первого домена и C-конец второго домена). Для этого воспользуемся данными о координатах доменов из InterPro.
Координаты начала архитектуры в выравнивании: 372
Координаты конца архитектуры в выравнивании: 1726
5. Вырежем этот участок, чтобы получить выравнивание по доменам.
Выравнивание двух доменов 6. Проведём выравнивание ещё раз, чтобы улучшить качество.
Вторая итерация выравнивания двух доменов 7. Удалим фрагменты, имеющие крупные делеции.
Выравнивание без крупных делеций 8. Выберем последовательность из кластеров высокосходных последовательностей так, чтобы она отражала в себе большое количество других последовательностей (Был получен кластер из 4 последовательностей после уборки избыточных последовательностей при пороге 100% и кластер из 18 последовательностей при пороге 95%).
Выбранная последовательность из выравнивания 9. Подготовимся к построению HMM-профиля и поиску по нему.
Материал для построения профиля(выравнивание из пункта 8.) Материал для положительного контроля(Все последовательности подсемейства) Материал для калибровки профиля(Подсемейство с доменом, не пересекающееся с нашим выбраными подсемейством) 10. Строим HMM-профиль, калибруем и проводим по нему поиск последовательностей.
hmm2build HMM.txt seq.fasta
hmm2calibrate HMM.txt
HMM.txt hmm2search -E 0.005 --cpu 1 HMM.txt Urazov-full-189.fasta > full_res.txt
full_res.txt Нашли 152 из 189 последовательностей, порог E-value убрал ещё 2 => осталось 150 последовательностей. Не самый хороший результат. Возможные пути исправления: 1) можно было для материала для построения профиля взять несколько последовательностей, так как видимо hmm2search пропустил последовательности с другими АМК или небольшими делециями. 2) Возможно порог E-value 0.005 слишком низкий.
Теперь проведём hmm2search в материале для калибровки (наверное мы ничего не должны найти)
hmm2search -E 0.1 --cpu 1 HMM.txt cal_PF04721.fasta > calibration.txt
calibration.txt Нашли 2 совпадения из 111 последовательностей, но они не прошли порог E-value => 0 последовательностей.
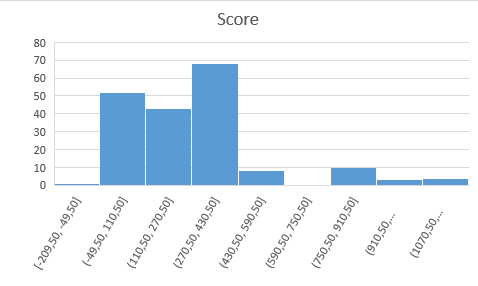
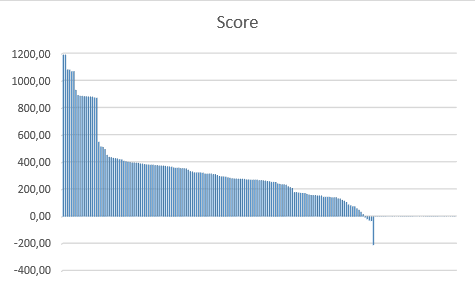
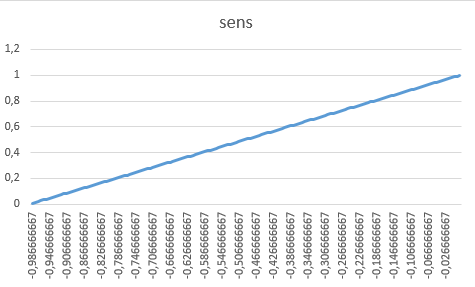
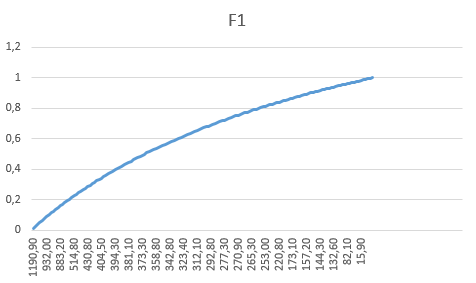
11. Сделаем таблицу из полученных данных.
Таблица 12. Проанализируем данные.