Занятие 9. Эволюционные домены - часть 1.
Домен и архитектуры белков, содержащих его
Для исследования был выбран домен Kringle. В таблице указана основная информация о нём:
| AC | ID | Функция домена | Число разных доменных архитектур с этим доменом | Число последовательностей | Число видов |
| PF00051 | Kringle | Домен сложен в 3 большие петли, которые стабилизируются 3 дисульфидными мостиками, что является очень важным в
белок-белковых взаимодействиях с факторами коагуляции крови. Почти полностью состоит из бета-листов. Обнаружен в плазминогене, факторах роста гепатоцитов, протромбине и липопротеинах плазмы крови. Так же найден в белках коагуляции крови и фибринолитических белках. Считается, что домен играет роль в присоединении медиаторов и регуляции протеолитической активности. |
273 | 2697 | 179 |
Скачали выравнивание из Pfam, раскрасили по консервативности (ClustalX => By conservation 10%). Добавили 3D структуру домена к последовательности UROK_HUMAN из PDB ID 1KDU (правая кнопка => Structure => Associate structure with sequence). Выравнивание было сохранено как проект JalView в файле kringle.jar и в формате fasta в файле kringle.fasta.
Выбор архитектур
С использованием скрипта swisspfam-to-xls.py и файла swisspfam.gz, содержащего информацию об архитектуре всех последовательностей UniProt, была получена таблица с информацией об архитектуре последовательностей, содержащих домен Kringle. На её основе в Excel была составлена сводная таблица, которая содержит AC записей, домены Pfam и таксономическую принадлежность белков(лист "Сводная таблица"). Для получения таблицы использовали команду:
python swisspfam_to_xls.py -p PF00051 -i /srv/databases/pfam/swisspfam.gz -z -o swisspfam_to_xls_out.txt
Для дальнейшего изучения эволюции доменных архитектур, включающих домен Kringle, были выбраны архитектуры:
| Домены | Изображение | Число представителей | Характеристика других доменов |
| Kringle Trypsin |
88 | Trypsin: домен серин-протеазы. Расщепляет белки и пептиды, обладает эстеразной активностью. | |
| PAN_1 Kringle x 4 Trypsin |
115 | PAN_1: содержит ядро из трех дисульфидных связей. Участвует во взаимодействиях белков и белков с углеводами Trypsin: домен серин-протеазы. Расщепляет белки и пептиды, обладает эстеразной активностью. |
Скорее всего каждый домен во второй архитектуре выполняет свою функцию, поэтому всего архитектур получается как бы 5.
Выбор таксона и подтаксонов
Для изучения указанных выше архитектур был выбран таксон Euteleostomi с подтаксонами Mammalia и Actinopterygii для первой архитектуры и Mammalia, Amphibia и Actinopterygii для второй.
Euteleostomi представляет собой кладу, включающую в себя более 90% современных позвоночных.
Класс млекопитающие – Mammalia – относится к подтипу позвоночные (Vertebrata), который является высшим подтипом хордовых. Земноводные – Amphibia — класс позвоночных четвероногих животных, в числе прочих включающий тритонов, саламандр, лягушек и червяг. Actinopterygii – класс лучеперых рыб, к которому относятся около 95% современных видов рыб.
Лист "Distinguished" в таблице содержит идентификаторы выбранных поседовательностей с указанием архитектуры и подтаксонов.
Для каждой архитектуры было отобрано по 21 последовательности. Для архитектуры Kringle+Trypsin было выбрано 11 последовательностей из Mammalia и 10 последовательностей из Actinopterygii. Для второй архитектуры - PAN_1 + Kringle x 4 + Trypsin - было выбрано 8 последовательностей из Mammalia 5 последовательностей из Amphibia и 10 последовательностей из Actinopterygii. Чтобы оставить в выравнивании нужные последовательности из двух групп (был использован скрипт filter_alignment.py, файл с последовательностями - out_selected.fasta).
Полученное выравнивание было загружено в JalView и отредактировано (удалены пустые колонки).В нём были выделены группы согласно архитектуре, в каждой из них была выполнена раскраска последовательностей ClustalX, Conservation (порог на консервативность 10%). Так же были удалены N- и C-концевые участки, в которых выравнивание было явно плохим, удалены некоторые последовательности (E1BCW0_BOVIN и B4DNJ4_HUMAN). Так же к одной из последовательностей - UROK_HUMAN, которая относилась к первой архитектуре, была привязана 3D структура, и в соответствии с ней вторичная структура домена была аннотирована. В конечном итоге файл с выравниванием JalView содержит 40 последовательностей. На рисунках представлено полученное выравнивание:
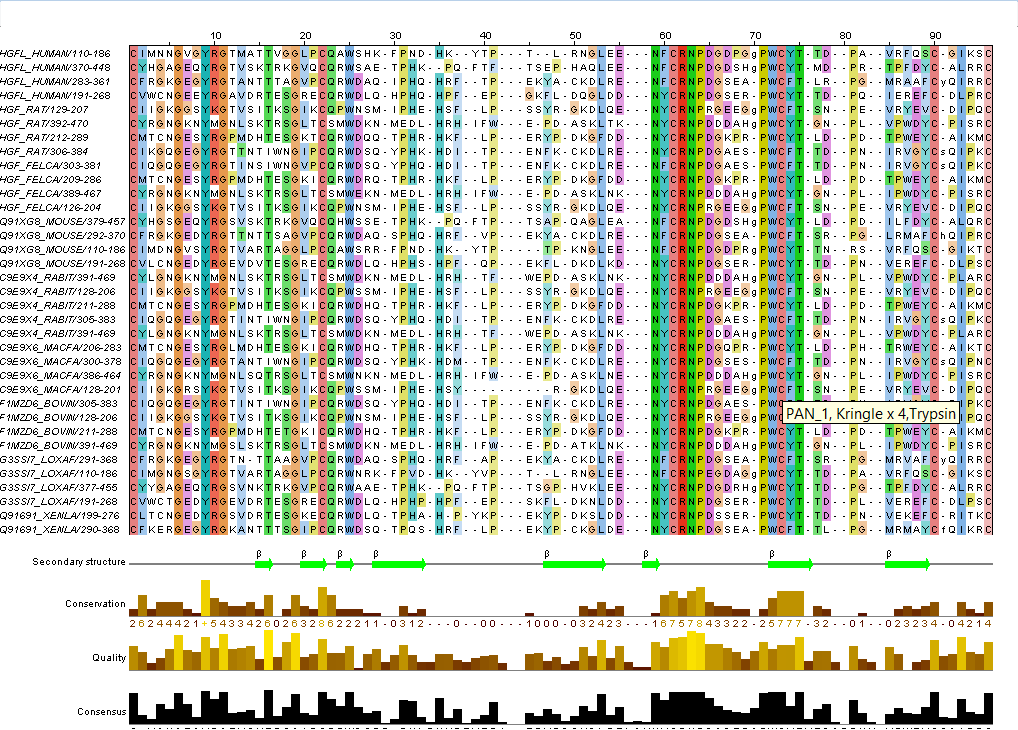
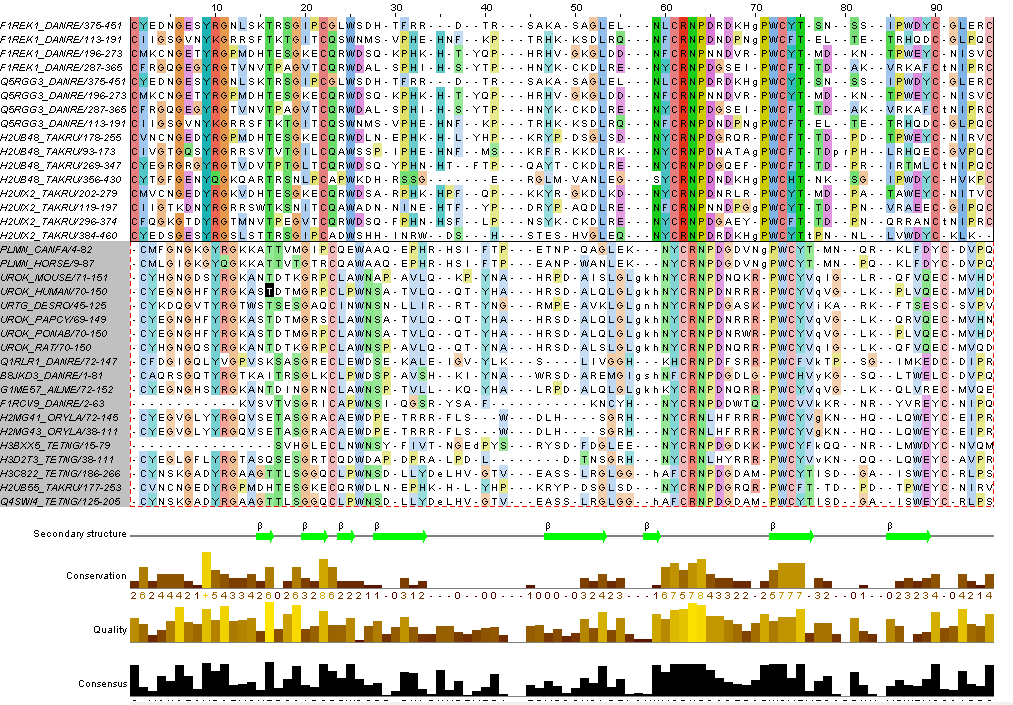
Проект с выравниванием - в файле arch.jar
Занятие 10. Эволюционные домены - часть 2.
Для того, чтобы построить филогенетическое дерево, для удобства доменные структуры и подтаксоны были зашифрованы следующим образом:
| Шифр | Значение |
| 1 | Архитектура Kringle + Trypsin |
| 2 | Архитектура PAN_1 + Kringle x 4 + Trypsin |
| M | Mammalia |
| A | Actinopterygii |
| Am | Amphibia |
Для построения дерева использовались последовательности из файла out_selected.fasta и метод Neighbor-Joining (не использует гипотезу о молекулярных часах и строит неукоренённое дерево) программы MEGA. Скобочная структура дерева - в файле tree.nwk.
Для визуализации дерева была использована программа iTOL. Изображение, полученное с помощью программы iTOL, приведено ниже. Холодными тонами выделены последовательности, относящиеся к архитектуре 1, теплыми - к архитектуре 2. Раскраска таксонов:
- 1 M - сиреневый
- 1 А - синий
- 2 М - желтый
- 2 А - персиковый
- 2 Am - красный
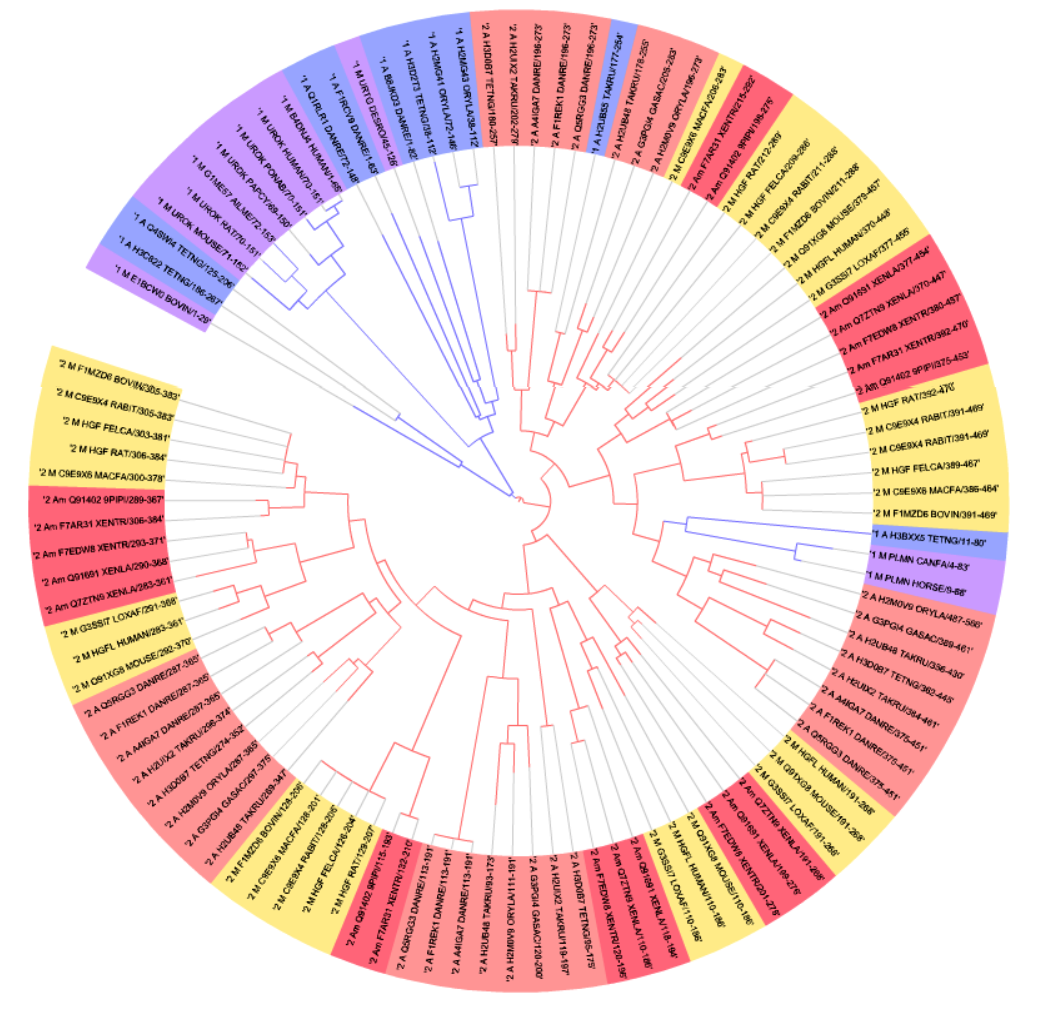
Архитектуры на дереве разделены, что может говорить о том, что общий предок Млекопитающих, Амфибий и Лучеперых рыб имел обе архитектуры. В большинстве случаев разделение на таксоны прослеживается четко.
Исключением для четкого разделения архитектур являются 4 последовательности из 1-й архитектуры попали в другую кладу.
1_A_H2UB55_TAKRU/177-254 объединена в одну ветвь с 2_A_H2UB48_TAKRU/178-255, их последовательности полностью совпадают. Из названий последоватльеностей заключаем, что они из TrEMBL, и скорее всего такой выброс может быть связан с тем, что при получении 1_A_H2UB55_TAKRU/177-254 отсеквенировали неполный кусок, другие архитетуры обрезались и последовательность отнесли к первой архитектуре.
Так же среди в ветвь второй архитектуры попали последовательности 1_M_PLMN_CANFA/4-83, 1_M_PLMN_HORSE/9-88 и 1_A_H3BXX5_TETNG/11-80. Можно предположить, что в ходе эволюции произошла делеция первых трех доменов Kringle, и архитектура 1 сформировалась повторно, но уже из второй.
Для второй доменной структуры можно отметить, что есть хорошее разделение последовательностей по положению домена (то есть группы с доменами, расположенными примерно на ~ 110-186 остатках или ~ 283-361 объединяются в свои ветви). Это подтверждает независимость доменов Kringle.
Для оценки достоверности полученных деревьев был использован бутстрэп-анализ. Изображение полученного дерева приведено ниже. В рамки обведены группы последовательностей, для которых координаты для домена на белке приблизительно одинаковы.
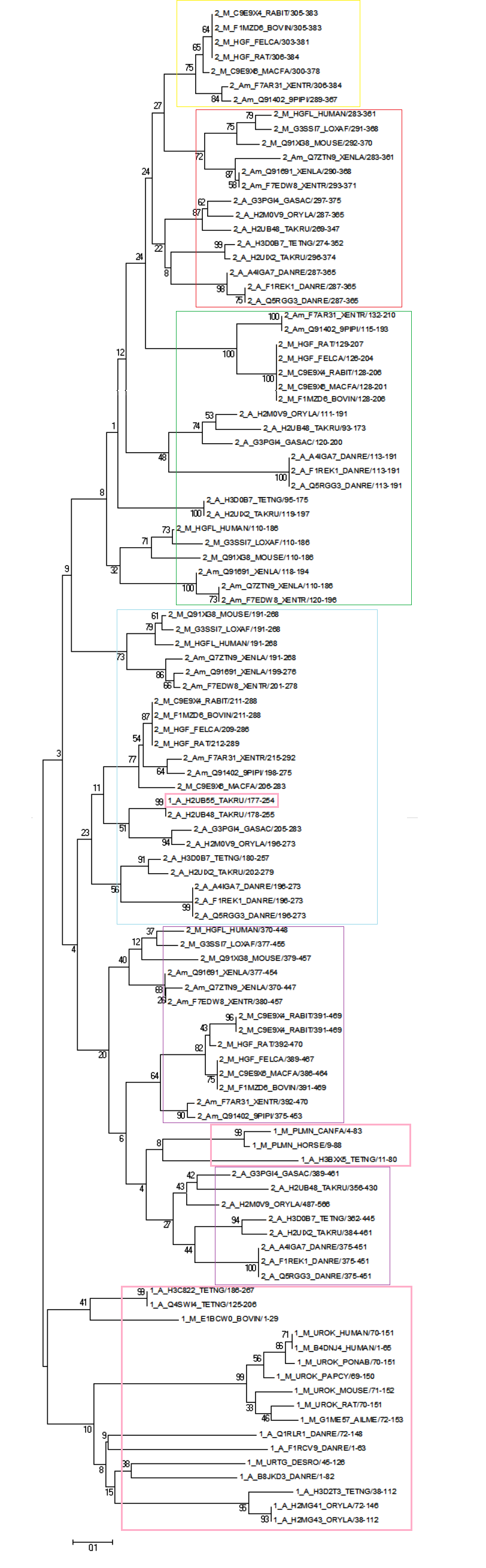
Бросается в глаза, что ветви, отделяющие большие группы, например последовательности с двумя разными архитектурами, имеют очень слабую поддержку. Однако Сильную поддержку имеют только ветви, отделяющие последовательности, принадлежащие одному таксону и содержащие домены, сходные по положению на последовательности.
Таким образом, общий предок рассмотренных таксонов имел обе архитектуры. В процессе эволюции первая архитектура была получена повторно из второй.