Домены и профили
Выбор домена и доменной архитектуры
Для выбора домена я случайным образом выбрала букву, затем скопировала в таблицу и отфильтровала по нужным параметрам.
Из полученной отфильтрованной таблицы мной был выбран домен GAGA (GAGA factor), связывающийся с консенсусным сайтом 5'-GAGAG-3' на ДНК и содержащий ядро цинкового пальца: цистеин-гистидин. Характеристики домена:
- AC: PF09237
- число последовательностей в: seed – 2; full – 186; uniprot – 378
- cредняя длина домена – 46.1 а/к
- cреднее сходство (identity) – 42%
- Средний процент покрытия последовательности белка доменом (coverage) – 9.8%
Для данного домена известны 26 различных архитектур. Для изучения была выбрана двудоменная архитектура,
состоящая из домена GAGA и BTB (сначала идет BTB, затем GAGA) и встречающаяся в 77 последовательностях из 186 известных. Ее схема на рисунке 1:
 Рис.1 Двудоменная архитектура GAGA и BTB
Рис.1 Двудоменная архитектура GAGA и BTB
Все последовательности, содержащие домен GAGA, были скачаны в файл. Далее из полученного файла был составлен список AC, Их получилось 170 из-за того, что в скрите использовались множества и дубликаты были удалены (обнаружила это уже когда смотрела таблицу результатов hmmsearch, надеюсь, что это не критично)
Из раздела "Domain organisation" были скопированы описания с AC последовательностей, соответствующих выбранной двудоменной архитектуре (полученный файл с АС). Далее из файла full.fasta были выгружены последовательности, соответствующие выбранной архитектуре, по АС. Результат в fasta формате.
Полученный fasta файл был выровнен в Jalview, после чего над выравниванием была произведена ревизия: удалены субъективно-лишние последовательности и фрагменты выравнивания до первого домена и после второго, также были удалены все полностью-идентичные последовательности. Длина профиля составила 382 а/к, взято 24 последовательности. Результат в файле, а
также на рисунке 2:
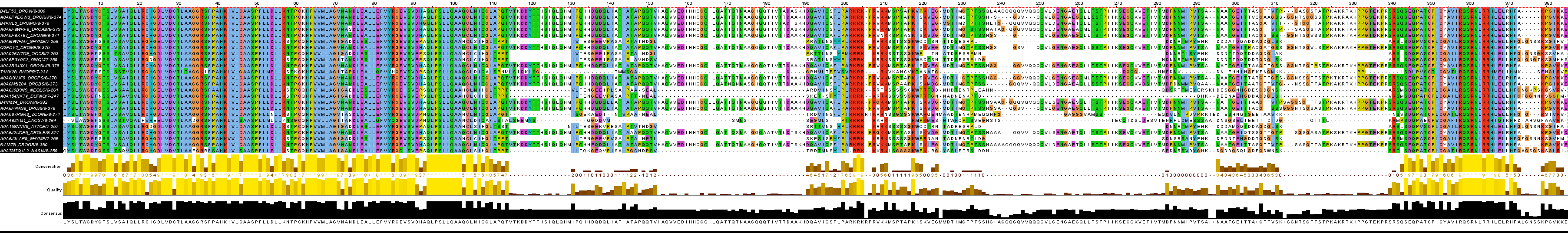 Рис.2 Выравненная учебная выборка
Рис.2 Выравненная учебная выборка
Построение HMM-профиля семейства белков
Для построения профиля, калибровки и поиска по профилю использовались следующие команды:
hmm2build -g hmm_out.txt final.fasta
hmm2calibrate hmm_out.txt
hmm2search --cpu 1 hmm_out.txt full.fasta > hmm2search_out.txt
В результате был получен HMM-профиль двухдоменной архитектуры, именющий длину 384 аминокислот. hmm2search_out.txt — файл с результатами поиска профиля по набору белков с выбранным доменом.
Из полученного log файла была выгружена таблица с результатами поиска. В переданных последовательностях было найдено 158 последовательностей с E-value менее 0.01, в которых можно предположить наличие изучаемой двудоменной стрктуры. В двух последовательностях E-value было меньше порога (они не внесены в таблицу). Еще в десяти последовательностях полученный профиль не был найден
Анализ HMM-профиля
Из файла, полученного командой hmm2search, с помощью скрипта была получена таблица, содержащая информацию о 145 находках: AC, Domain, seq-f, seq-t, hmm-f, hmm-t, score, E-value. Также был добавлен столбец true с ответами yes – если AC встретился в списке белков с данной архитектурой, или no в противном случае.
Данная таблица была экспортирована в Excel, после чего с помощью формул для нее были посчитаны значения specificity, 1 - sensitivity и F1. Таблица с данными, формулами и графиками доступна по ссылке. По полученным данным были построены следующие 3 графика: