-
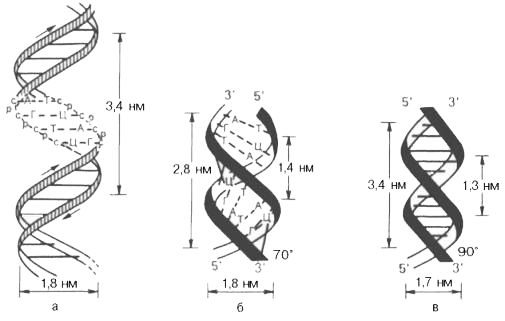
С помощью программы fiber пакета 3DNA построены A- и B- формы дуплекса ДНК, последовательность одной из нитей которого - 4 раза повторенная последовательность "gatc".
| fiber -a gatc-a.pdb |
Для того, чтобы получить структуру дуплекса в B- форме, была использована команда:
| fiber -b gatc-b.pdb |
-
Изучение полученных PDB файлов при помощи программы RasMol.
| A-форма | B-форма | Файл dna12.pdb | |
| Тип спирали (правая или левая) | правая | правая | правая |
| Шаг спирали (Å) | 28.03 (C16A.P - G5A.P) |
33.75 (C16A.P - A6A.P) |
30.57 (G12A.P - G2A.P) |
| Число оснований на виток | 10 | 11 | 10 |
| Ширина большой бороздки | 7.98 (G9A.P - A18B.P) |
17.21 (A18B.P - C12A.P) |
17.12 (A18B.P - G4A.P) |
| Ширина малой бороздки | 16.97 (C8A.P - C28B.P) |
11.69 (T7A.P - A30B.P) |
11.13 (C11A.P - A18B.P) |
| Вид с торца | 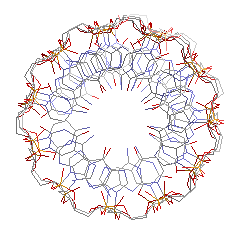 |
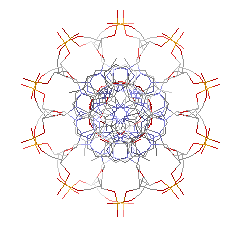 |
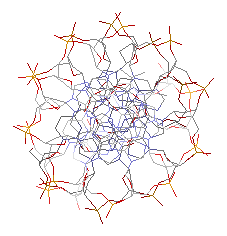 |
Для того, чтобы измерить шаг спирали, выделим все атомы фосфора в структуре и посчитаем расстояние между двумя атомами, отстающими на фазу (удобство этого метода обусловлено тем, что в каждом дезоксирибонуклеотиде только один атом фосфора).
В таблице приведены атомы, между которыми измерялось расстояние. Также в таблице присутствуют изображения A- и B- форм с торца.
По результатам, полученным для dna12 можно сделать вывод о том, что данная структура ближе к B-форме. (Что неудивительно, ведь большинство молекул ДНК находятся в клетке как раз в такой конформации).
-
Провести анализ трёх структур, используя программы find_pair и analyze.
| Команда | Полученный файл |
| find_pair -t gatc-a.pdb stdout | analyze | gatc-a.out |
| find_pair -t gatc-b.pdb stdout | analyze | gatc-b.out |
| find_pair -t dna12.pdb stdout | analyze | dna12.out |
В результате, по данным из полученных файлов была составлена таблица.
Таблица значений конформационно важных торсионых углов (α, β, γ, δ, ε, ζ, χ).
| Угол | α | β | γ | δ | ε | ζ | χ |
| Связь | O3'(i-1)-P-O5'-C5' | P-O5'-C5'-C4' | O5'-C5'-C4'-C3' | C5'-C4'-C3'-O3' | C4'-C3'-O3'-P(i+1) | C3'-O3'-P(i+1)-O5'(i+1) | for pyrimidines(Y): O4'-C1'-N1-C2 for purines(R): O4'-C1'-N9-C4 |
| A-форма | -51.7 | 174.8 | 41.7 | 79.1 | -147.8 | -75.1 | -157.2 |
| B-форма | -29.9 | 136.4 | 31.1 | 143.4 | -140.8 | -160.5 | -98.0 |
| dna12 | -58.8 | 55 | 59.3 | 118.4 | -36.3 | -65.2 | -116.6 |
Поскольку средние значения конформационно важных углов сильно разнятся (порой даже до знака), сравним значения углов для A- и B-форм ДНК с единичными углами для заданной мне структуры dna12.
Сравнение значений конформационно важных торсионных углов A- и B- форм ДНК с заданной структурой dna12.
| Угол | α | β | γ | δ | ε | ζ | χ |
| Связь | O3'(i-1)-P-O5'-C5' | P-O5'-C5'-C4' | O5'-C5'-C4'-C3' | C5'-C4'-C3'-O3' | C4'-C3'-O3'-P(i+1) | C3'-O3'-P(i+1)-O5'(i+1) | for pyrimidines(Y): O4'-C1'-N1-C2 for purines(R): O4'-C1'-N9-C4 |
| A-форма | -51.7 | 174.8 | 41.7 | 79.1 | -147.8 | -75.1 | -157.2 |
| B-форма | -29.9 | 136.4 | 31.1 | 143.4 | -140.8 | -160.5 | -98.0 |
| dna12 | 0 -57.0 -61.6 -65.1 -56.8 -66.3 -59.9 -60.9 -59.6 -65.0 -65.1 -90.3 -67.9 -92.2 -92.8 -60.3 -53.6 -61.1 -61.1 -51.6 -58.0 -53.4 -50.8 0 |
0 166.8 173.3 -175.8 173.9 177.7 -179.1 177.2 179.9 173.7 147.2 -167.3 175.8 150.2 -162.1 -179.9 175.1 169.0 176.0 -174.6 -180.0 164.6 158.0 0 |
175.7 47.1 58.3 59.8 64.4 61.0 5.3 65.1 49.3 61.8 46.5 54.9 51.1 42.7 49.0 42.3 57.0 67.7 60.2 54.4 62.7 51.9 47.2 38.3 |
170.5 113.5 107.9 147.5 136.7 126.3 95.8 102.6 99.2 149.5 149.2 100.2 85.7 123.5 155.9 85.6 109.0 100.3 100.7 140.4 146.1 82.8 136.4 154.7 |
-165.3 178.5 -179.8 171.6 -175.9 175.0 173.9 164.3 -171.7 -115.8 -173.2 0 0 -176.4 -74.0 156.8 -177.6 -175.8 179.6 173.6 166.1 172.1 -178.0 -155.9 |
-136.4 -85.4 -94.8 -113.1 -99.6 -89.4 -80.2 -76.7 -76.9 159.7 -88.9 0 0 -108.4 157.9 -49.4 -84.3 -83.3 -78.0 -94.2 -107.1 -79.4 -110.6 -147.4 |
-90.9 -121.1 -133.6 -105.4 -116.6 -118.8 -129.6 -126.8 -132.9 -94.3 -111.8 -107.6 -132.8 -108.3 -90.1 -123.0 -127.9 -140.9 -133.7 -107.9 -100.2 -140.6 -110.0 -94.8 |
Получаем, что "количественно" данная структура ближе всё-таки к A- форме, особенно по значения α, β, γ, ζ, а по значениям углов δ и χ - к B-форме. Значения угла ε не сближают рассматриваемую структуру ни с A-, ни с B-формами.
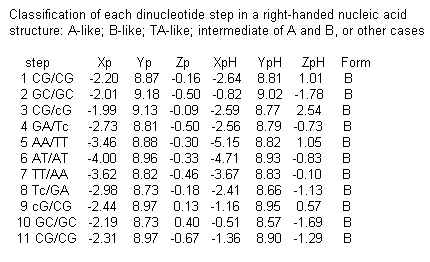
Кроме того, более подробный анализ результатов, полученных при помощи программы analyze, подтвердил сделанные "визуально", то есть при помощи программы RasMol, заключения, отражённые в первой таблице:
| gatc-a.out | gatc-b.out | dna12.out | |
| Тип спирали | This is a right-handed nucleic acid structure | This is a right-handed nucleic acid structure | This is a right-handed nucleic acid structure |
| Ширина большой бороздки |
1 GA/TC 0 2 AT/AT 0 3 TC/GA 15.2 4 CG/CG 15.2 5 GA/TC 15.2 6 AT/AT 15.2 7 TC/GA 15.2 8 CG/CG 15.2 9 GA/TC 15.2 10 AT/AT 15.2 11 TC/GA 15.2 12 CG/CG 15.2 13 GA/TC 15.2 14 AT/AT 0 15 TC/GA 0 |
1 GA/TC 0 2 AT/AT 0 3 TC/GA 17.2 4 CG/CG 17.2 5 GA/TC 17.2 6 AT/AT 17.2 7 TC/GA 17.2 8 CG/CG 17.2 9 GA/TC 17.2 10 AT/AT 17.2 11 TC/GA 17.2 12 CG/CG 17.2 13 GA/TC 17.2 14 AT/AT 0 15 TC/GA 0 |
1 CG/CG 0 2 GC/GC 0 3 CG/cG 17.9 4 GA/Tc 17.8 5 AA/TT 17.1 6 AT/AT 16.1 7 TT/AA 17.4 8 Tc/GA 18.5 9 cG/CG 17.6 10 GC/GC 0 11 CG/CG 0 |
| Ширина малой бороздки | 1 GA/TC 0 2 AT/AT 0 3 TC/GA 18.5 4 CG/CG 18.5 5 GA/TC 18.5 6 AT/AT 18.5 7 TC/GA 18.5 8 CG/CG 18.5 9 GA/TC 18.5 10 AT/AT 18.5 11 TC/GA 18.5 12 CG/CG 18.5 13 GA/TC 18.5 14 AT/AT 0 15 TC/GA 0 |
1 GA/TC 0 2 AT/AT 0 3 TC/GA 11.7 4 CG/CG 11.7 5 GA/TC 11.7 6 AT/AT 11.7 7 TC/GA 11.7 8 CG/CG 11.7 9 GA/TC 11.7 10 AT/AT 11.7 11 TC/GA 11.7 12 CG/CG 11.7 13 GA/TC 11.7 14 AT/AT 0 15 TC/GA 0 |
1 CG/CG 0 2 GC/GC 0 3 CG/cG 13.4 4 GA/Tc 11.7 5 AA/TT 10.2 6 AT/AT 9.4 7 TT/AA 9.5 8 Tc/GA 10.4 9 cG/CG 11.1 10 GC/GC 0 11 CG/CG 0 |
 |
Кроме того, программа analyze выдаёт ещё массу интересной информации, например:
- информация о водородных связях
- расположение комплементарной пары в системе координат заданной структуры
- локальные параметры шага
- классификация структуры
- информация о большой и малой бороздках (приведена выше)
- информация о конформационно важных торсионных углах
- параметры конформации дезоксирибозы
- длины P-P и C1'-C1' связей
- и др
-
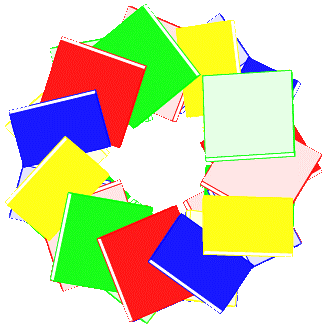
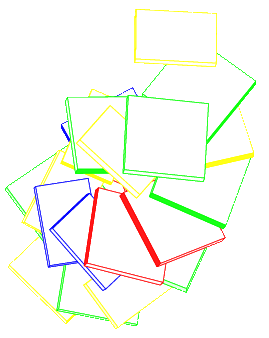
Получить изображение данных структур в виде стопочных моделей.
| A-форма | B-форма | dna12 |
| pdb2img -c -u gatc-a.pdb gatc-a.ps rotate_mol -b gatc-a.pdb gatc-a_view.pdb pdb2img -c -u gatc-a_view.pdb gatc-a_view.ps |
pdb2img -c -u gatc-b.pdb gatc-b.ps rotate_mol -b gatc-b.pdb gatc-b_view.pdb pdb2img -c -u gatc-b_view.pdb gatc-b_view.ps |
pdb2img -c -u dna12.pdb dna12.ps rotate_mol -b dna12.pdb dna12_view.pdb pdb2img -c -u dna12_view.pdb dna12_view.ps |
 |
 |
 |
 |
 |
 |
Протокол к занятию.
Главная Первый семестр Второй семестр Третий семестр