- Информация о pdb-файле.
Предложенная мне структура 2FMT.pdb представляет собой комплекс метилтрансферазы и тРНК.ДНК-метилтрансферазы – группа ферментов, осуществляющих метилирование оснований в ДНК. У прокариот известно несколько ДНК-метилтрансфераз, специфичных в отношении «окружения» метилируемого основания – например, продукт гена dam метилирует аденин в последовательности ГАТЦ, что позволяет ферментам репаративной системы отличать родительскую цепь ДНК от дочерних (неметилированных); модифицирующие ДНК-метилтрансферазы метилируют основания в сайтах рестрикции, делая их устойчивыми к действию соответствующих рестриктаз. У эукариот метилирование цитозина в положении С-5 с участием ДНК-метилтрансферазы является механизмом контроля активности генов (как правило, метилированные гены репрессированы, и наоборот).
Источник: http://www.dictionary.cbio.ru/termin.php?id=285
Таким образом, предложенный для изучения файл содержит две молекулы.
| MOL_ID: 1 | MOL_ID: 2 | |
| Название | FORMYL-METHIONYL-TRNAFMET2 | METHIONYL-TRNA FMET FORMYLTRANSFERASE |
| Цепи | C, D | A, B |
| Синоним | INITIATOR TRNA | 10-FORMYLTETRAHYDROFOLATE L-METHIONYL TRNAFMET FORMYLTRANSFERASE |
| Из какого организма | ESCHERICHIA COLI, K37 | |
| Местоположение в клетке | CYTOPLASM |
Будем работать с цепью C.
-
Последовательность изучаемой цепи тРНК.
| SEQRES 1 C 78
C G C G
G G G 4SU G
G A G
C SEQRES 2 C 78 A G C C U G G H2U A G C U C SEQRES 3 C 78 G U C G G G OMC U C A U A A SEQRES 4 C 78 C C C G A A G A U C G U C SEQRES 5 C 78 G G 5MU PSU C A A A U C C G G SEQRES 6 C 78 C C C C C G C A A C C A FME |
| Сокращённое название | Полное название | Формула | Позиция |
| 4SU | 4-THIOURIDINE-5'-MONOPHOSPHATE | 2(C9 H13 N2 O8 P S) | 8 |
| H2U | 5,6-DIHYDROURIDINE-5'-MONOPHOSPHATE | 2(C9 H15 N2 O9 P) | 20 |
| OMC | O2'-METHYLYCYTIDINE-5'-MONOPHOSPHATE | 2(C10 H16 N3 O8 P) | 32 |
| 5MU | 5-METHYLURIDINE 5'-MONOPHOSPHATE | 2(C10 H15 N2 O9 P) | 54 |
| PSU | PSEUDOURIDINE-5'-MONOPHOSPHATE | 2(C9 H13 N2 O9 P) | 55 |
| FME | N-FORMYLMETHIONINE | 2(C6 H11 N O3 S) | 77 |
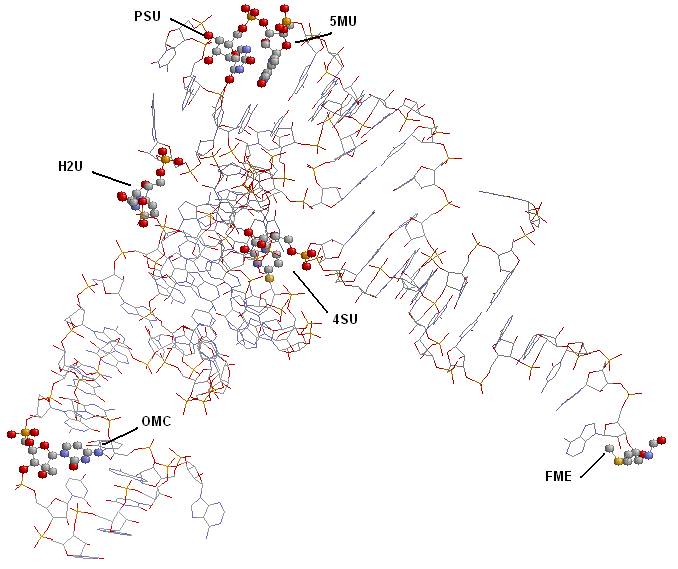
Здесь изображена изучаемая цепь РНК в проволочной модели и выбраны гетероатомы, то есть атомы модифицированных оснований, с раскраской по атомам. Модифицированные основания подписаны.
Начальный нуклеотид изучаемой цепи - 1, конечный - 76, пропусков нет, есть вставка -17A(insertion codes), информация о которых содержится на 27 позиции строки ATOM.
Использовалась команда grep:
| grep -n 'ATOM.*P.*P' 2FMT.pdb > num.txt |
-
Провести анализ структуры с использованием программы find_pair.
| find_pair -t 2FMT1.PDB stdout | analyze |
| find_pair -t -d 2FMT1.PDB stdout | analyze |
| stdout_0001 | 2FMT1.PDB 2FMT1_0001.out 2 # duplex 13 # number of base-pairs 1 1 # explicit bp numbering/hetero atoms 2 71 0 # 1 | C:...2_:[..G]G-----C[..C]:..71_:C 0.60 0.54 11.00 8.94 0.18 3 70 0 # 2 | C:...3_:[..C]C-----G[..G]:..70_:C 1.20 0.99 20.10 8.93 1.68 4 69 0 # 3 | C:...4_:[..G]G-----C[..C]:..69_:C 0.56 0.54 10.64 8.84 0.15 5 68 0 # 4 | C:...5_:[..G]G-----C[..C]:..68_:C 0.78 0.53 11.85 9.08 0.33 6 67 0 # 5 | C:...6_:[..G]G-----C[..C]:..67_:C 0.45 0.36 16.66 8.98 -0.33 7 66 0 # 6 | C:...7_:[..G]G-----C[..C]:..66_:C 1.07 0.53 14.66 9.47 0.62 49 65 0 # 7 | C:..49_:[..G]G-----C[..C]:..65_:C 0.59 0.28 6.44 9.22 -0.34 50 64 0 # 8 | C:..50_:[..U]U-*---G[..G]:..64_:C 2.14 0.85 7.95 8.73 3.83 51 63 0 # 9 | C:..51_:[..C]C-----G[..G]:..63_:C 0.55 0.40 6.57 8.81 -0.14 52 62 0 # 10 | C:..52_:[..G]G-----C[..C]:..62_:C 0.50 0.21 7.06 9.38 -0.58 53 61 0 # 11 | C:..53_:[..G]G-----C[..C]:..61_:C 0.37 0.24 1.67 9.31 -0.66 54 58 0 # 12 | C:..54_:[5MU]u-**--A[..A]:..58_:C 3.69 0.47 12.38 7.65 4.63 55 18 0 # 13 | C:..55_:[PSU]P-**+-G[..G]:..18_:C 6.55 1.33 37.42 9.14 9.21 ##### Base-pair criteria used: 4.00 15.00 2.50 65.00 4.50 7.50 ##### 3 non-Watson-Crick base-pairs, and 1 helix (0 isolated bps) ##### Helix #1 (13): 1 - 13 ***broken O3'[i] to P[i+1] linkage*** |
| stdout_0002 | 2FMT1.PDB 2FMT1_0002.out 2 # duplex 14 # number of base-pairs 1 1 # explicit bp numbering/hetero atoms 35 33 0 # 1 | C:..35_:[..A]A-*---U[..U]:..33_:C 8.81 0.78 45.07 5.78 10.38 38 32 0 # 2 | C:..38_:[..A]A-*---c[OMC]:..32_:C 1.33 0.56 26.40 9.70 2.45 39 31 0 # 3 | C:..39_:[..C]C-----G[..G]:..31_:C 0.57 0.15 20.22 8.60 -0.63 40 30 0 # 4 | C:..40_:[..C]C-----G[..G]:..30_:C 0.78 0.40 11.85 8.89 0.07 41 29 0 # 5 | C:..41_:[..C]C-----G[..G]:..29_:C 0.41 0.35 8.13 9.07 -0.40 42 28 0 # 6 | C:..42_:[..G]G-----C[..C]:..28_:C 0.96 0.87 26.06 8.92 1.19 43 27 0 # 7 | C:..43_:[..A]A-----U[..U]:..27_:C 0.36 0.22 21.12 8.83 -0.69 44 26 0 # 8 | C:..44_:[..A]A-*---G[..G]:..26_:C 1.54 0.10 20.52 10.79 1.74 10 25 0 # 9 | C:..10_:[..G]G-----C[..C]:..25_:C 0.89 0.27 2.35 8.95 -0.08 11 24 0 # 10 | C:..11_:[..A]A-----U[..U]:..24_:C 0.64 0.48 5.68 8.76 0.10 12 23 0 # 11 | C:..12_:[..G]G-----C[..C]:..23_:C 0.39 0.19 12.82 8.85 -0.72 13 22 0 # 12 | C:..13_:[..C]C-----G[..G]:..22_:C 1.29 0.34 9.98 8.73 0.48 14 8 0 # 13 | C:..14_:[..A]A-**--u[4SU]:...8_:C 4.43 0.29 19.08 7.43 5.01 15 48 0 # 14 | C:..15_:[..G]G-**+-C[..C]:..48_:C 2.87 0.15 5.36 8.89 3.16 ##### Base-pair criteria used: 4.00 15.00 2.50 65.00 4.50 7.50 ##### 5 non-Watson-Crick base-pairs, and 1 helix (0 isolated bps) ##### Helix #1 (14): 1 - 14 ***broken O3'[i] to P[i+1] linkage*** |
| stdout_0003 | 2FMT1.PDB 2FMT1_0003.out 2 # duplex 1 # number of base-pairs 1 1 # explicit bp numbering/hetero atoms 19 56 1 # 1 + C:..19_:[..G]G-----C[..C]:..56_:C 1.28 0.50 50.18 9.35 0.78 ##### Base-pair criteria used: 4.00 15.00 2.50 65.00 4.50 7.50 ##### 0 non-Watson-Crick base-pairs, and 1 helix (1 isolated bp) ##### Helix #1 (1): 1 |
CGCGGGG 4SU GGAGCAGCCUGG H2U AGCUCGUCGGG OMC UCAUAACCCGAAGAUCGUCGG 5MU
PSU CAAAUCCGGCCCCCGCAACCA FME
 Скрипт:
Скрипт:select all
center selected
wireframe off
backbone 100
#Выбираем и красим первую спираль
define helix1 2-7:C,66-71:C
select helix1
color green
#Выбираем и красим вторую спираль
define helix2 49-53:C,61-65:C
select helix2
color violet
#Выбираем и красим третью спираль
define helix3 38-44:C,26-32:C
select helix3
color pink
#Выбираем и красим четвёртую спираль
define helix4 10-13:C,22-25:C
select helix4
color blue
#Подписываем 5' и 3' конец
color label black
select c1.p
label 1
select c75.p
label 75
В результате получилась такая картинка, где найденные спирали окрашены в те же цвета, какими размеченычто и на последовательности.Также, на картинке подписаны номера остатков при атомах фосфора 5'-концевого и 3'-концевого нуклеотидов, как и требовалось в задании.
-
Пользуясь средствами RasMol проанализировать структуру.
Note: This structure contains 8[4] non-Watson-Crick base-pairs.
В том же файле находим, какие это взаимодействия:
| Взаимодействие из 2FMT1.out | Изображение RasMol | Химическая структура | Расстояние (Å) |
| 8 (0.009) C:..50_:[..U]U-*---G[..G]:..64_:C (0.007) | 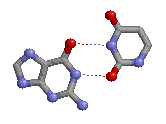 |
 |
3.07 (G64C.O6 - U50C.N5) 2.80 (G64C.N1 - U50C.O2) |
| 12 (0.013) C:..54_:[5MU]u-**-xA[..A]:..58_:C(0.005) | 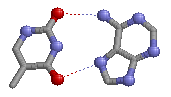 |
3.20(A58C.N6-5MU54C.O2) 2.90(A58C.N7-5MU54C.O4) |
|
| 13 (0.043) C:..55_:[PSU]Px**+xG[..G]:..18_:C(0.007) | 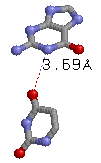 |
3.69(G18C.N1-PSU55C.O4) | |
| 14 (0.005) C:..35_:[..A]Ax*---U[..U]:..33_:C (0.005) | 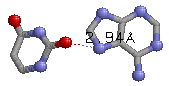 |
2.94(A35C.N7 - U33C.O2) | |
| 15 (0.005) C:..38_:[..A]A-*---c[OMC]:..32_:C(0.009) | 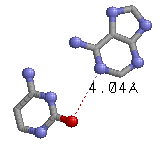 |
4.04(A38C.N1-OMC32C.02) | |
| 21 (0.012) C:..44_:[..A]Ax*---G[..G]:..26_:C (0.010) | 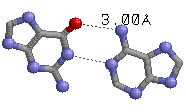 |
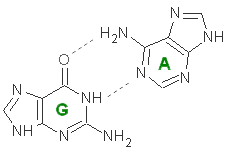 |
3.00(G26C.O6 - A44C.N6) 2.89(G26C.N1 - A44C.N1) |
| 26 (0.008) C:..14_:[..A]A-**-xu[4SU]:...8_:C (0.006) | 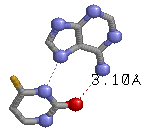 |
3.06(1SU8C.N3-A14C.N7) 3.10(4SU8C.O2-A14C.N6) |
|
| 27 (0.007) C:..15_:[..G]Gx**+xC[..C]:..48_:C (0.006) | 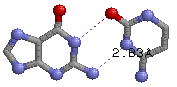 |
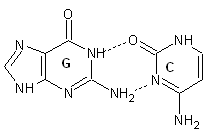 |
3.00(G15C.N1-C48C.O2) 2.83(G15C.N2-C48C.N3) |
Также был создан скрипт, при помощи которого можно последовательно получить изображённые картинки.
Поиск стекинг-взаимодействий:
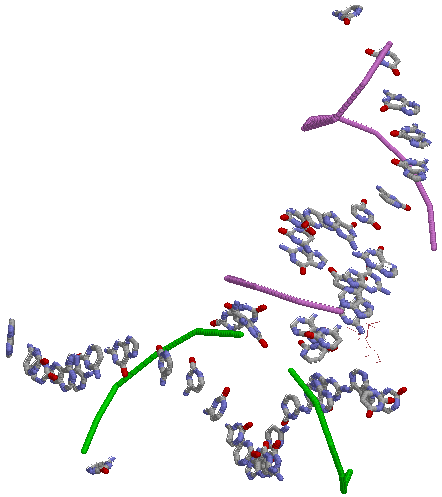 На
картинке изображена изучаемая структура. Спирали выделены теми же
цветами и показаны в той же модели, как и на предидущем рисунке, кроме
третьей спирали, состоящей из одного остатка - она представлена в
проволочной модели. Кроме этого, на рисунке также изображены остальные
остатки данной цепи РНК в проволочной модели, атомы азота и кислорода -
в шариковой модели с раскраской по атомам. Также можно посмотреть скрипт, генерирующий это изображение.
На
картинке изображена изучаемая структура. Спирали выделены теми же
цветами и показаны в той же модели, как и на предидущем рисунке, кроме
третьей спирали, состоящей из одного остатка - она представлена в
проволочной модели. Кроме этого, на рисунке также изображены остальные
остатки данной цепи РНК в проволочной модели, атомы азота и кислорода -
в шариковой модели с раскраской по атомам. Также можно посмотреть скрипт, генерирующий это изображение.Теперь рассмотрим внеспиральные стекинг взаимодействия между основаниями.
По рисунку видно, что "подозрительными" на наличие стекинг взаимодействий можно считать многие пары оснований, но, поскольку данное исследование проводится исключительно визуально, ограничимся одной тройкой и одной четвёркой "идеальных" на мой взгляд примеров внеспирального стекинг взаимодействия.
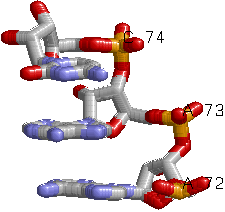 |
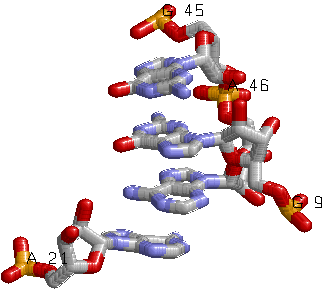 |
Теперь попробуем узнать, на какую из форм ДНК больше всего похожа спираль РНК? Рассмотрим первую спираль (покрашена зелёным цветом).
На один виток спирали приходится примерно 10.5 оснований, спираль правая, чисто визуально малая бороздка шире большой.Поэтому кажется, что это всё же A-форма. Теперь посмотрим на файл 2FMT1.out:
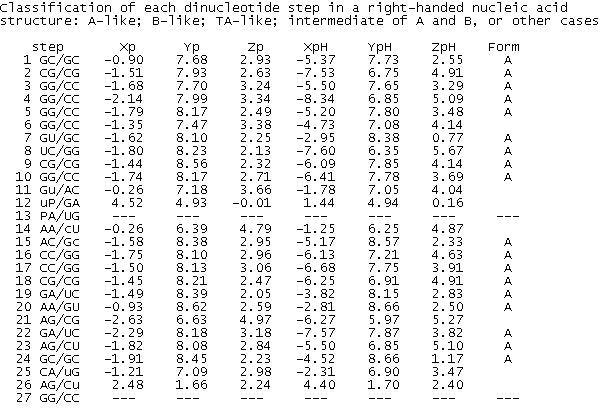
-
Поиск инвертированных последовательностей в НК при помощи программы einverted из EMBOSS.
Работа с программой einverted:
einverted
Finds DNA inverted repeats
Input nucleotide sequence(s): 2FMT.fasta
Gap penalty [12]: 12
Minimum score threshold [50]: 50
Match score [3]: 3
Mismatch score [-4]: -4
Sanger Centre program inverted output file [outfile.inv]: 2FMT.inv
File for sequence of regions of inverted repeats. [outfile.fasta]: regions.fasta
Полученные файлы оказались пустыми.
Пришлось изменить параметр Minimum score threshold
einverted 2FMT.fasta
Finds DNA inverted repeats
Gap penalty [12]: 12
Minimum score threshold [50]: 5
Match score [3]: 3
Mismatch score [-4]: -4
Sanger Centre program inverted output file [outfile.inv]: 2FMT.inv
File for sequence of regions of inverted repeats. [outfile.fasta]: regions.fasta
Полученные данные:
: Score 18: 6/6 (100%) matches, 0 gaps
2 gcgggg 7
| | | | | |
72 cgcccc 67
Несмотря на то, что файл не пуст, выдача программы всё равно не удовлетворительна, т.к. при помощи программы find_pair было найдено 3 участка, содержащих в общей сложности 28 "двойных" кусков, хотя они и соответствуют частично первой сприали, выданной программой find_pair.
2 71 0 # 1 | C:...2_:[..G]G-----C[..C]:..71_:C 0.60 0.54 11.00 8.94 0.18
3 70 0 # 2 | C:...3_:[..C]C-----G[..G]:..70_:C 1.20 0.99 20.10 8.93 1.68
4 69 0 # 3 | C:...4_:[..G]G-----C[..C]:..69_:C 0.56 0.54 10.64 8.84 0.15
5 68 0 # 4 | C:...5_:[..G]G-----C[..C]:..68_:C 0.78 0.53 11.85 9.08 0.33
6 67 0 # 5 | C:...6_:[..G]G-----C[..C]:..67_:C 0.45 0.36 16.66 8.98 -0.33
7 66 0 # 6 | C:...7_:[..G]G-----C[..C]:..66_:C 1.07 0.53 14.66 9.47 0.62
Поэтому я раскрасила их зелёным.
Возможно, частично вина в таком низком результате полученной информации модифицированных основани