Практикум 4
Список гомологичных белков, включающих паралоги
Я взяла протеомы со следующими мнемониками: ACICJ, AROAE, BORPE, BURMA, HAEIN, PARDP, POLAQ.
Записала последовательности в файл proteomes.fasta, произвела поиск программой blastp гомологов с порогом на E-value 0,0001. Использованные команды:
cat ACICJ.fasta AROAE.fasta BORPE.fasta BURMA.fasta HAEIN.fasta PARDP.fasta POLAQ.fasta > proteomes.fasta
makeblastdb -dbtype prot -in proteomes.fasta -out col
blastp -query CLPX.fasta -db col -evalue 0.0001 -out blastp_result.txt
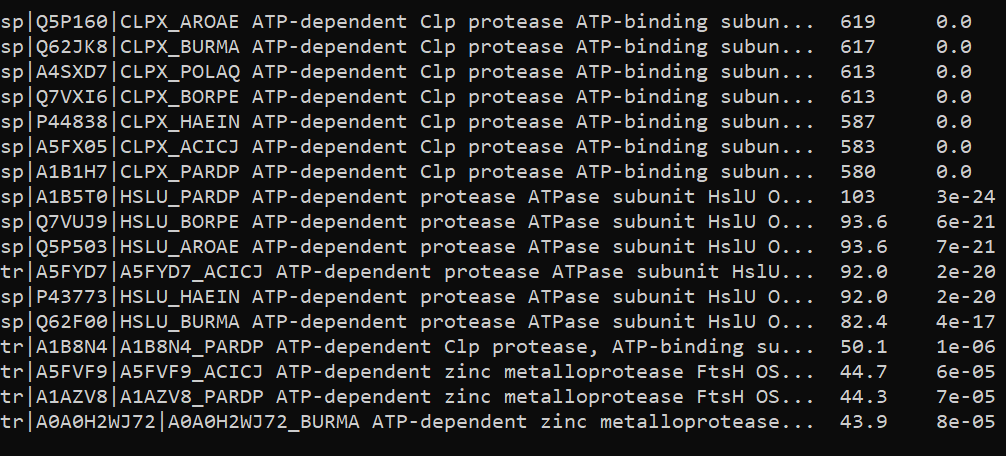
Файл с выдачей blast
Реконструкция и визуализация
Названия находок поменяла на их мнемоники, сделала выравнивание с помощью muscle. Построила на основе выравнивания дерево программой iqtree.
Файл с формулой (Newick)Полученное дерево открыла в iTOL и укоренила в среднюю точку.

Пары ортологов:
- CLPX ACICJ, CLPX PARDP;
- CLPX BURMA, CLPX AROAE;
- HSLU PARDP, A5FYD7 ACICJ;
- HSLU BURMA, HSLU BORPE.
Пары паралогов:
- A1B8N4 PARDP, CLPX PARDP;
- A1AZV8 PARDP, HSLU PARDP;
- A5FVF9 ACICJ, A5FYD7 ACICJ;
- A0A0H2WJ72 BURMA, HSLU BURMA.

Описание состава группы CLPX
Включает в себя белки всех выбранных организмов.
Реконструированная филогения белков сильно отличаестя от филогении бактерий. Клада с POLAQ, BORPE, BURMA, AROAE реконструирована неверно: POLAQ и BURMA должны были образовывать кладу, вместо этого BURMA образует кладу с AROAE. Также должна быть клада из POLAQ, BORPE, BURMA, AROAE и HAEIN, вместо этого образовалась клада из HAEIN с PARDP и ACICJ.
Описание состава группы HSLU
Включает в себя белки всех выбранных организмов за исключением POLAQ.
Реконструированная филогения отличается от филогении бактерий только тем, что HAEIN не образует отдельной клады с BORPE и BURMA, в остальном видно соответствие.