Практикум 6.
Описание входных данных
Мне был предложен следующий список ID генов человека: ссылка.
Список состоит из 19 генов.
Данный перечень генов был проанализирован с помощью баз данных GO и Reactome.
База данных GO
Для того, чтобы выяснить на основе чего была выделена данная группа генов, какая между ними функциональная связь, был проведен анализ обогащения терминами.
GO - инструмент, предназначенный для структурирования информации о продуктах генов. Представляет собой по сути граф с терминами в качестве вершин, ребра этого графа - отношения между терминами, например "A является частью B" ("A part of B").
Термины GO применяются для аннотирования генов, их продуктов, пример - система PANTHER. Для интересующего нас анализа сервисом, предоставленным PANTHER, я и воспользовалась.
Параметры запуска анализа:
метод (Test Type) - тест Фишера,
поправка на множественное тестирование - False Discovery Rate (FDR).
Результаты:
Анализ проводился отдельно по трем группам терминов GO: Biological Process, Molecular Function, Cellular Component.
Biological Process - процессы, состоящие из нескольких молекулярных механизмов, т.е. различные биохимические, метаболические пути и т.п.
Ссылка на файл с выдачей
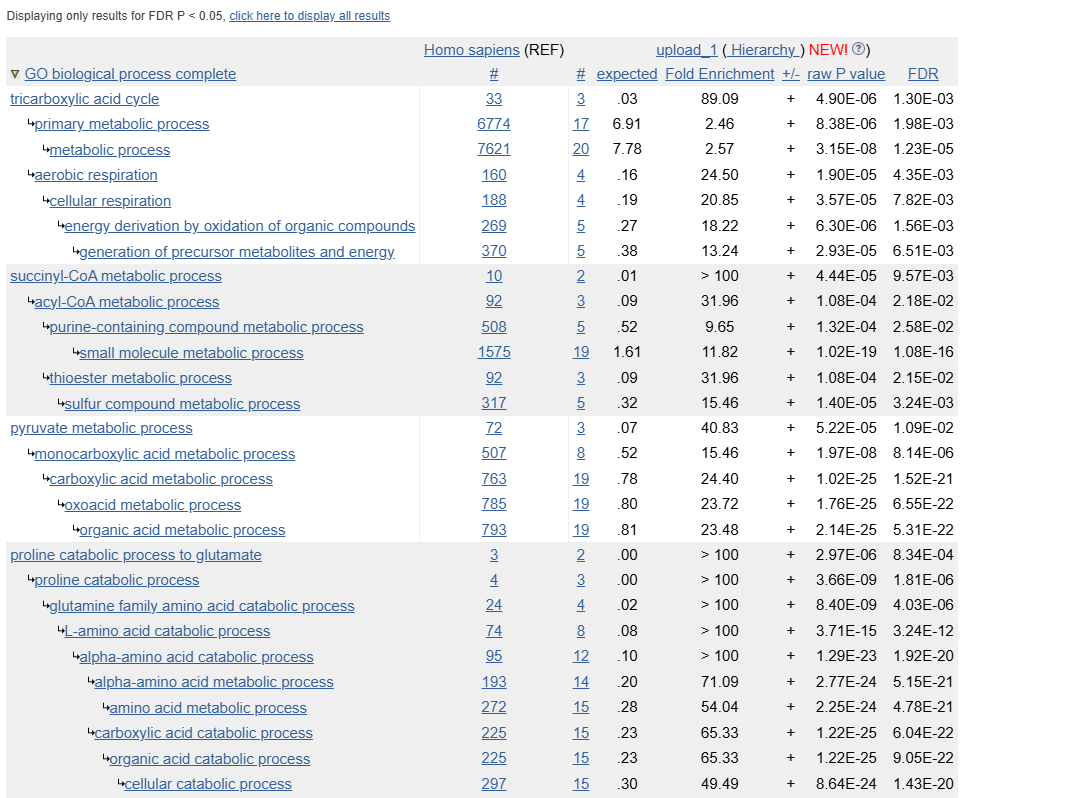
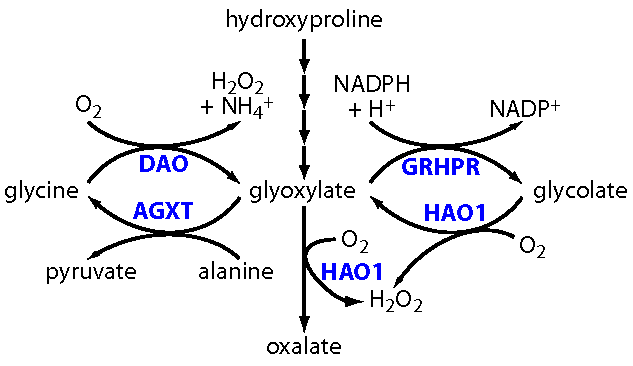
Список оказался довольно объемным, 77 терминов с p-value ниже 0.05, большинство связаны с метаболизмом аминокислот: гидроксипролина, аспартата, аланина, а еще D-аминокислот.
Хочется отдельно отметить термины, связанные с катаболизмом глицина и превращениями глиоксилата.
Остальные термины касаются цикла трикарбоновых кислот.
Molecular Function - механизмы, взаимодействия продуктов генов на молекулярном уровне.
Ссылка на файл с выдачей
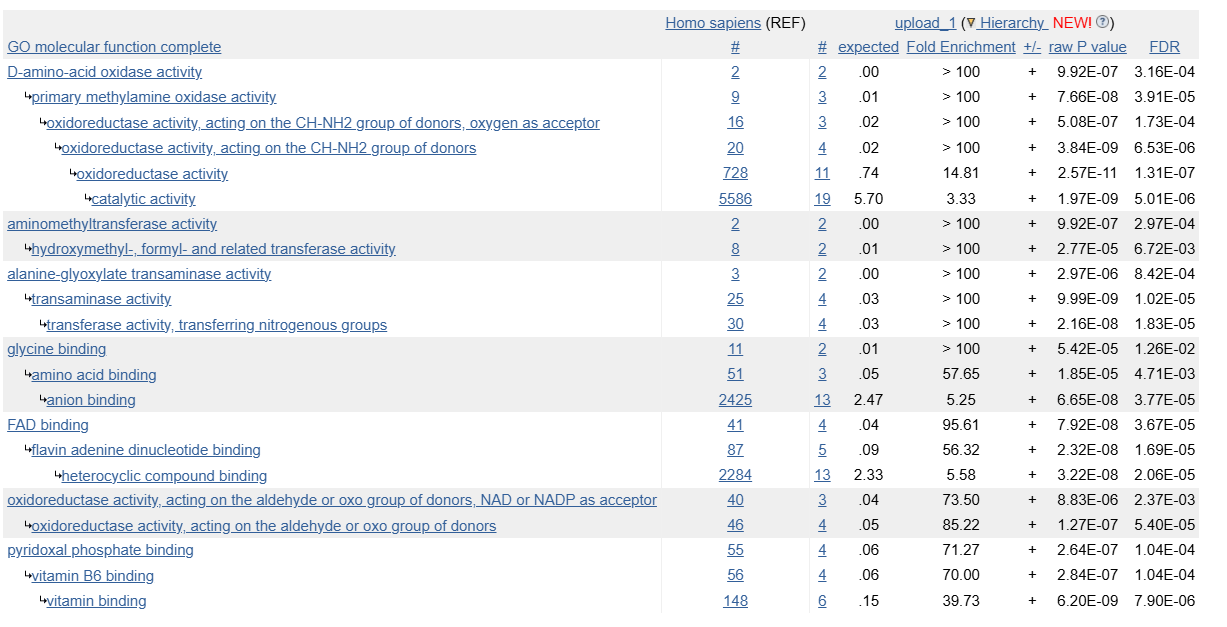
Терминов нашлось 20. Вот тут проясняется связь генов с D-аминокислотами, а именно то, что в представленном наборе встречаются гены, отвечающие за активность оксидазы D-аминокислот (DAAO) и связывание DAAO с флавинадениндинуклеотидом (FAD).
Помимо этого видим, что продукты некоторых генов из перечня являются белками системы расщепления глицина (glycine cleavage system, GCS). Например, связываются с глицином (glycine binding), отвечают за активность аминометилтрансферазы (aminomethyltransferase activity), входящей в GCS.
Так, погуглив, я нашла, что ген GLDC кодирует P белок этой системы. К нему же может относиться "связывание пиридоксальфосфатом" ("pyridoxal phosphate binding"), который является кофактором данного белка.
Cellular Component - локализация процессов.
Ссылка на файл с выдачей
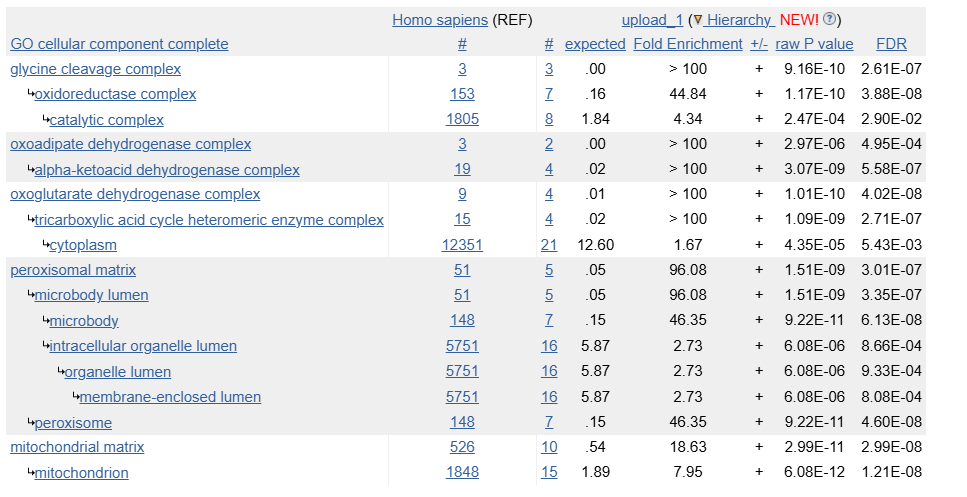
В этой выдаче получили 17 терминов. Собственно подтвердилась связь с GCS. 2-оксоглутаратдегидрогеназный комплекс (oxoglutarate dehydrogenase complex, OGDC) - ключевой фермент в цикле трикарбоновых кислот. 2-оксоадипатдегидрогеназный вообще задействован в катаболизме L-лизина, здесь возник видимо из-за того, что имеет общие компоненты с OGDC, и вовлечен в метаболические процессы в митохондриях, как и многие продукты генов из перечня.
Касательно локализации в клетке - выдача показала митохондриальный матрикс и матрикс пероксисом.
Выводы
Анализ показал, что исследуемые гены связаны метаболическими путями, в которых участвуют их продукты: есть пересечения между циклом трикарбоновых кислот в митохондриях, метаболизмом глицина, а также детоксикацией глиоксилата в пероксисомах.
Как мне кажется, выбранные гены объединены именно деградацией глицина (с помощью GCS, окислением до глиоксилата пероксисомальной оксидазой D-аминокислот).
База данных Reactome
Чтобы подтвердить свое предположение и может быть найти иные возможные связи, да и просто по фану, решила заглянуть в Reactome.
Reactome предоставляет детальные схемы путей биологических процессов (опять же метаболических и т.п.).
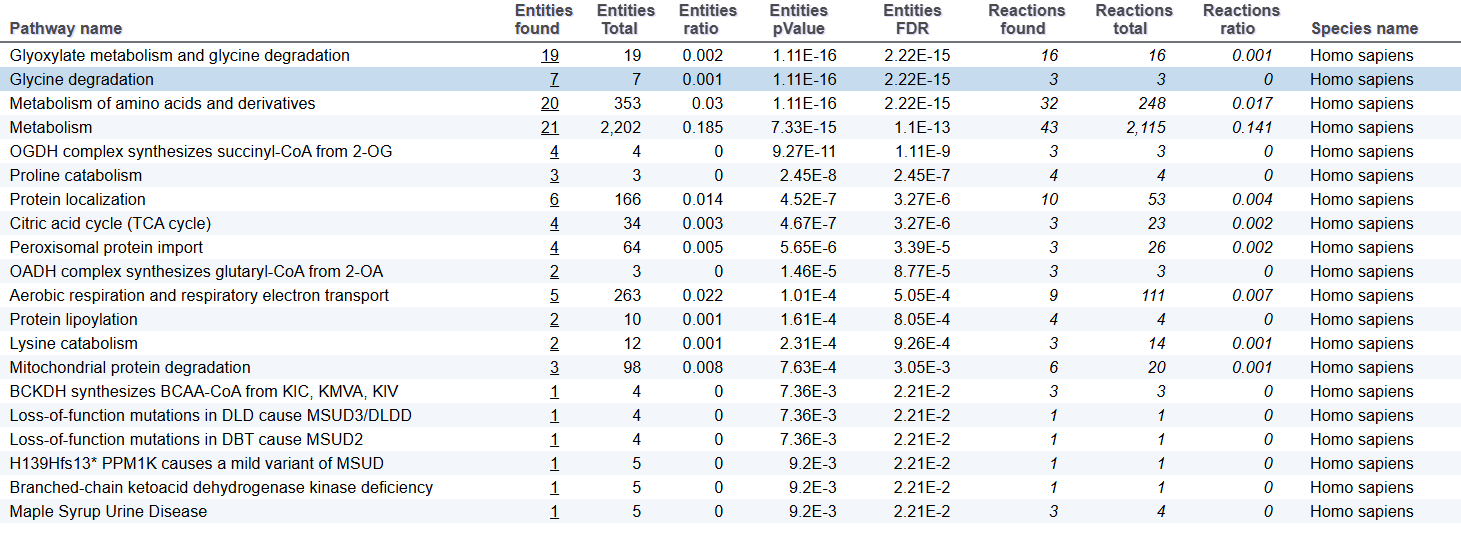
Подтвердилась вовлеченность генов из перечня в деградации глицина, в частности путем превращения в глиоксилат под действием оксидазы D-аминокислот.
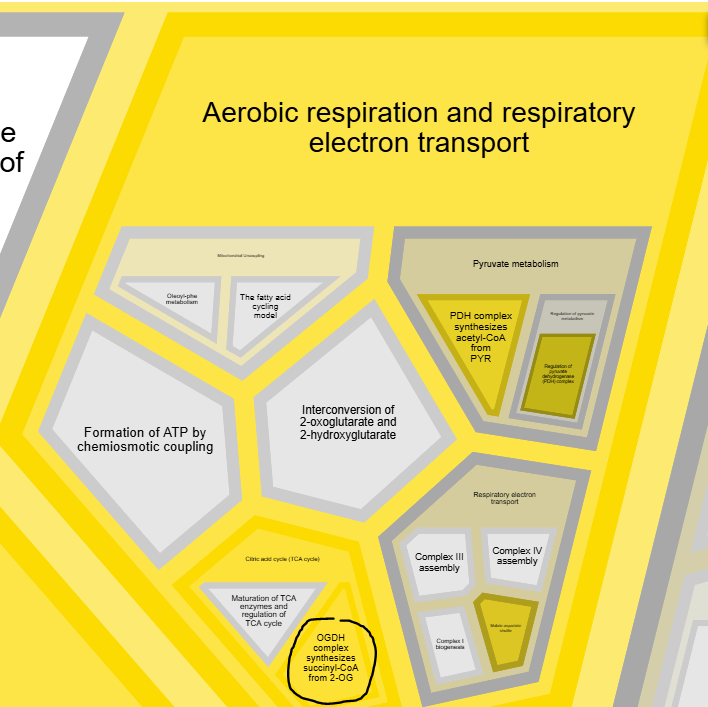
Выяснилась роль конкретного гена OGDH. Он кодирует субъединицу 2-оксоглутаратдегидрогеназного комплекса (OGDHc) - ключевого фермента в митохондриальном матриксе, катализирующего декарбоксилирование альфа-кетоглутарата до suc-CoA в цикле трикарбоновых кислот.
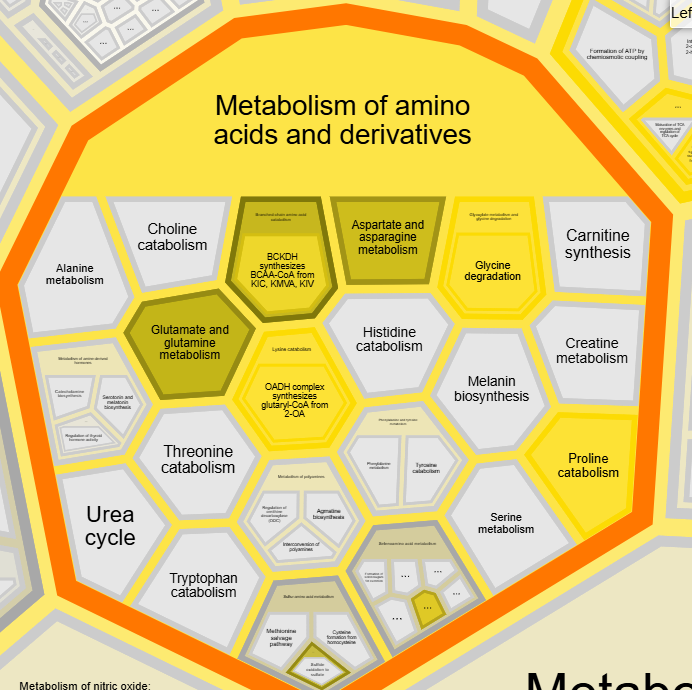
Многие гены вовлечены в метаболизм других помимо глицина аминокислот, например, пролина.
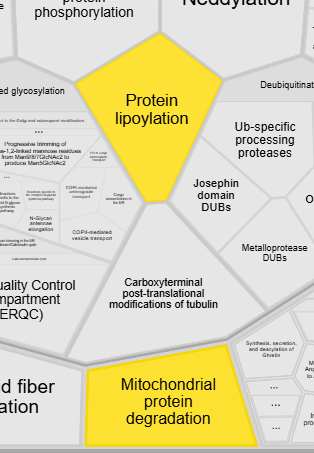
В некоторых метаболических путях, в том числе метаболизме некоторых аминокислот, принимают участие белки, ковалентно связанные с остатком липоевой кислоты. Например в систему GCS входит Н-белок, который содержит липоевую кислоту. Липоилрование происходит преимущественно в митохондриях.

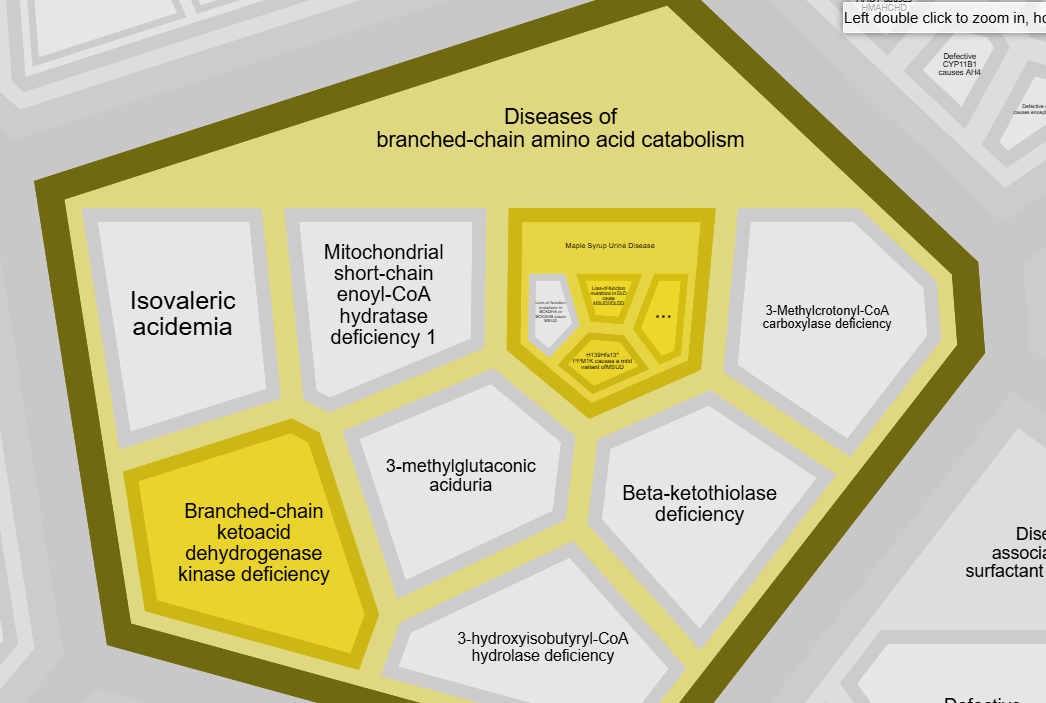
Наконец, была найдена связь с лейцинозом, при котором наблюдается накопление лейцина, изолейцина и валина из-за снижении активности дегидрогеназного комплекса разветвлённых α-кетокислот (BCKDC).
BCKDC состоит из множества субъединиц, одну из которых кодирует ген DLD.
Эта субъединица участвует в декарбоксилировании аминокислот с разветвленной цепью (лейцина, изолейцина, валина), она окисляет липоевый остаток на другой субъединице этого комплекса, благодаря чему поддерживается его активность.
Накопление аминокислот с разветвленной цепью, а следовательно пирувата и молочной кислоты, токисчно и поэтому приводит к проявлению симптомов болезни.
Выводы
Анализ с помощью баз GO и Reactome показал, что данные гены функционально связаны через метаболизм аминокислот, в первую очередь глицина, и участие в митохондриальных процессах.
Ключевыми метаболическими путями, в которых участвуют продукты данных генов, оказались:
- - Деградация глицина через систему расщепления глицина (GCS);
- - Превращение глицина в глиоксилат и его детоксикация в пероксисомах;
- - Цикл трикарбоновых кислот в митохондриальном матриксе;
- - Метаболизм аминокислот с разветвлённой цепью (лейцин, изолейцин, валин).