Анализ белков при помощи алгоритма BLAST
Поиск и анализ гомологов ГТФ-3′,8-циклазы
Был осуществлен поиск белковых последовательностей, сходных с таковой для ГТФ-3′,8-циклазы бактерии Octadecabacter antarcticus 307. Было установлено максимальное количество результатов в выдаче в 5000 последовательностей, пороговое значение e-value - 0.05, размер слова - 5, штраф за открытие инделя - 11, за продолжение - 1, использовалась матрица замен BLOSUM62.
В результате в базе данных были найдены 235 последовательностей, удовлетворявших условиям поиска (см. S1). Среди них были выбраны 6 последовательностей ГТФ-3′,8-циклаз, принадлежащих Caulobacter vibrioides, Gloeobacter violaceus, Synechococcus sp., Methanosarcina acetivorans и Halobacterium salinarum, последовательности белка биосинтеза молибденового кофактора 1 Bos taurus и Dictyostelium discoideum, а также последовательность циклазы pqqA-пептида из протеома бактерии Erwinia tasmaniensis.
Для данных белков было построено множественное выравнивание при помощи алгоритма MAFFT (см. S2). Из него видно, что ГТФ-3′,8-циклазы и белки биосинтеза молибденового кофактора всех организмов имеют консервативные регионы, некоторые из которых можно обнаружить и в последовательности циклазы pqqA-пептида, что может говорить о потенциальной гомологии рассматриваемых ферментов и будет обсуждено далее.
Как можно заметить из построенного выравнивания, последовательности белков биосинтеза молибденового кофактора эукариотических организмов (Bos taurus и Dictyostelium discoideum) значительно длиннее таковых у бактерий и архей, причем на последовательность ГТФ-3′,8-циклазы Octadecabacter antarcticus 307 выравниваются N-концевые части данных последовательностей. Чтобы изучить функцию C-концевых участков данных эукариотических белков был осуществлен поиск последовательностей, сходных с C-концевым участком рассматриваемого фермента Bos taurus. В результате в Swiss-Prot были найдены 248 последовательностей, среди которых большую часть составляли последовательности бактериальных синтаз циклического пираноптеринмонофосфата (cPMP-синтаз).
Для дальнейшего анализа были взяты последовательности cPMP-синтаз, принадлежащих Vibrio cholerae, Yersinia enterocolitica, Serratia proteamaculans и Bacillus pumilus, найденные по последнему запросу белки биосинтеза молибденового кофактора 1, принадлежащие Bos taurus, Homo sapiens, Mus musculus и Drosophila melanogaster, а также последовательности ГТФ-3′,8-циклаз Octadecabacter antarcticus 307, Caulobacter vibrioides, Gloeobacter violaceus, Synechococcus sp., Methanosarcina acetivorans и Halobacterium salinarum. Для данных белков при помощи алгоритма MAFFT было построено множественное выравнивание (см. S3), которое показало наличие гомологии между N-концевой частью белка биосинтеза молибденового кофактора 1 и прокариотическими ГТФ-3′,8-циклазами, а также между его C-концевым участком и прокариотическими cPMP-синтазами, что наводит на мысли о том, что белок биосинтеза молибденового кофактора 1 является продуктом слияния ГТФ-3′,8-циклазы и cPMP-синтазы, белков, осуществляющих 2 последовательные реакции в пути биосинтеза молибдоптерина (Рис. 1).
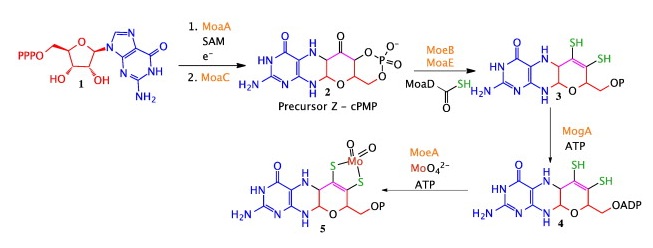
Действительно, ген белка биосинтеза молибденового кофактора 1 (MOCS1) кодирует 2 неперекрывающиеся рамки считывания: MOCS1A, кодирующую ГТФ-3′,8-циклазу, и MOCS1B, кодирующую cPMP-синтазу. При этом благодаря альтернативному сплайсингу для данного гена существуют 2 возможных транскрипта: первый, бицистронный с отдельными ORF, и второй, со слитыми ORF. Причем, как показали исследования, с бицистронной мРНК идет синтез только ГТФ-3′,8-циклазы, тогда как активная cPMP-синтаза образуется только с моноцистронной мРНК в слитом с ГТФ-3′,8-циклазой состоянии [2].
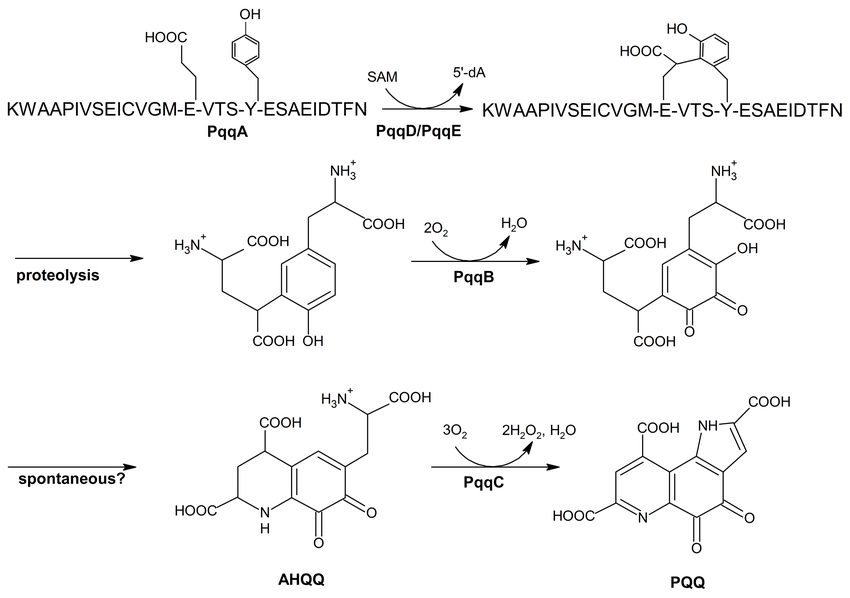
Среди результатов поиска по первому запросу имелись последовательности циклазы pqqA-пептида - фермента, участвующего в биосинтезе пирролохинолинхинона (Рис. 2), который является важным бактериальным окислительно-восстановительным кофактором. Данный фермент осуществляет катализ циклизации пептидного предшественника pqqA по SAM-радикальному механизму (рис. 3). При помощи BLAST были найдены последовательности, сходные с таковой для циклазы pqqA-пептида Erwinia tasmaniensis. Далее при помощи алгоритма MAFFT было построено множественное выравнивание последовательностей циклаз pqqA-пептида Erwinia tasmaniensis, Klebsiella pneumoniae, Pseudomonas protegens, Azotobacter vinelandii, Acinetobacter baylyi и Gluconobacter oxydans, а также ГТФ-3′,8-циклаз Octadecabacter antarcticus 307, Caulobacter vibrioides, Gloeobacter violaceus, Synechococcus sp. и Halobacterium salinarum.
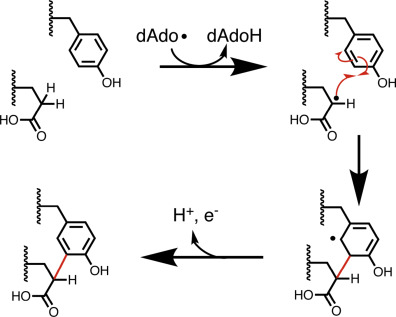
Выравнивание (см. S4) показало, что ГТФ-3′,8-циклаза и циклаза pqqA-пептида имеют общие консервативные участки, например, мотив СxxxCxxC, необходимый для связывания кластера 4Fe-4S [5], который имеется у большинства представителей суперсемейства SAM-радикальных ферментов, к которому данные два белка и относятся. Приведенные факты в совокупности позволяют говорить о вероятной гомологии данных белков.
Анализ вирусного полипротеина и одного из зрелых белков
Среди результатов поиска по базе данных Swiss-Prot с использованием запроса (taxonomy_id:2559587) AND (protein_name:polyprotein) был выбран полипротеин вируса Вессельсброна (WSLV, ID POLG_WSLV). Среди зрелых белков данного вируса был выбран неструктурный белок 1, который участвует в репликации вируса и в противодействии иммунному ответу хозяина, например, взаимодействуя с макрофагами и дендритными клетками и препятствуя передаче сигналов от Toll-подобных рецепторов 3 типа.
При помощи алгоритма BLAST был осуществлен поиск гомологичных последовательностей (см. S5). Для построения множественного выравнивания были взяты последовательности полипротеинов вирусов желтой лихорадки, Эдж Хилл, Зика, японского энцефалита, энцефалита долины Мюррей, лихорадки западного Нила и омской геморрагической лихорадки. В построенном выравнивании (см. S6) наблюдается высокая степень сходства в области, соответствующей рассматриваемому белку. Стоит также отметить, что почти все найденные BLAST последовательности принадлежат вирусам из рода Flavivirus.
Оценка доли вирусных белков в банке NCBI
Был снова осуществлен поиск по последовательности неструктурного белка 1 вируса Вессельсброна, однако в этот раз был указан таксономический фильтр "Viruses". Для выбранных ранее вирусных полипротеинов было рассчитано изменение e-value (Табл. 1).
| AC | Без фильтра | С фильтром | Изменение e-value |
|---|---|---|---|
| P29165.1 | 3E-179 | 1E-180 | 30.0 |
| C8XPB2.1 | 7E-149 | 3E-150 | 23.3 |
| Q32ZE1.1 | 1E-113 | 4E-115 | 25.0 |
| P0DOK8.1 | 3E-109 | 1E-110 | 30.0 |
| P05769.2 | 1E-108 | 5E-110 | 20.0 |
| P06935.2 | 3E-108 | 1E-109 | 30.0 |
| Q7T6D2.1 | 1E-96 | 6E-98 | 16.7 |
| Среднее | 25.0 | ||
В среднем для данных 7 последовательностей e-value уменьшается в 25 раз при добавлении фильтра "Viruses". Поскольку e-value линейно зависит от размера базы данных, можно предположить, что суммарная длина последовательностей вирусных белков составляет 4% от общей длины банка NCBI.
Поиск гомологов бессмысленной последовательности
Алгоритм BLAST был протестирован на двух бессмысленных последовательностях. Для первой при поиске по базе данных NR (Non-redundant protein sequences) не было найдено выравниваний с e-value меньше 10, но при увеличении порога e-value до 100 были найдены 4 выравнивания с e-value 44, 47, 65 и 68 (см. S7). Другая последовательность была получена из последовательности ГТФ-3′,8-циклазы Octadecabacter antarcticus 307 путем случайной перестановки букв методами Python (см. S8). Для запроса с данной последовательностью при поиске по Swiss-Prot было найдено одно выравнивание с e-value 0.084 (см. S9), но при поиске по базе данных NR выравниваний с e-value меньше 10 найдено не было, что является наглядной демонстрацией влияния размера базы данных на e-value выравнивания.
Сравнение интерфейсов BLAST
Помимо NCBI инструмент BLAST имеется также и на других сайтах, таких как EMBL-EBI. При поиске при помощи BLAST на сайте EMBL-EBI (Рис. 4) существует возможность выбрать базы данных, по которым будет проводиться поиск, причем перечни возможных баз данных различаются, так, например, при помощи NCBI BLAST можно осуществлять поиск белковых последовательностей по базам данных NR, RefSeq и PDB, а EMBL-EBI BLAST позволяет выбрать TrEMBL, UniParc, кластеры UniRef, а также некоторые специфические базы данных, такие как IPD (Immuno Polymorphism Database), ChEMBL и базы данных белков SARS-CoV-2. Также для EMBL-EBI BLAST существует возможность выбрать одновременно несколько баз данных для поиска.
И на сайте NCBI, и на сайте EMBL-EBI возможен поиск с использованием основных пяти вариантов BLAST: blastp, blastn, blastx, tblastn и tblastx. Также в обоих случаях существует возможность задания таксономических фильтров, штарфов за открытие и продолжение инделя, матрицы замен, порога e-value, размера слова для алгоритма поиска и максимального количества последовательностей в выдаче. Таким образом по функционалу данные инструменты сходны и выбор между ними зависит по большей степени от желаемой базы данных для поиска.
Также иструмент BLAST имеется на сайте FPbase - базы данных флуоресцентных белков (Fluorescent Protein BLAST). В данном случае интерфейс максимально упрощен (Рис. 5), отсутствуют возможности какой-либо настройки параметров алгоритма. Но данный иструмент вполне подойдет для быстрого поиска последовательностей флуоресцентных белков.
СОПРОВОДИТЕЛЬНЫЕ МАТЕРИАЛЫ
ЛИТЕРАТУРА И ИСТОЧНИКИ
Angad P. Mehta, Sameh H. Abdelwahed, Tadhg P. Begley, Molybdopterin biosynthesis—Mechanistic studies on a novel MoaA catalyzed insertion of a purine carbon into the ribose of GTP, Biochimica et Biophysica Acta (BBA) - Proteins and Proteomics, Volume 1854, Issue 9, 2015, Pages 1073-1077, ISSN 1570-9639, https://doi.org/10.1016/j.bbapap.2015.04.008.
Gray TA, Nicholls RD. Diverse splicing mechanisms fuse the evolutionarily conserved bicistronic MOCS1A and MOCS1B open reading frames. RNA. 2000 Jul;6(7):928-36. doi: 10.1017/s1355838200000182. PMID: 10917590; PMCID: PMC1369970.
Martins, Ana & Latham, John & Martel, Paulo & Barr, Ian & Iavarone, Anthony & Klinman, Judith. (2019). A two-component protease in Methylorubrum extorquens with high activity toward the peptide precursor of the redox cofactor pyrroloquinoline quinone. Journal of Biological Chemistry. 294. jbc.RA119.009684. 10.1074/jbc.RA119.009684.
Wen Zhu, Ana M. Martins, Judith P. Klinman, Chapter Fourteen - Methods for Expression, Purification, and Characterization of PqqE, a Radical SAM Enzyme in the PQQ Biosynthetic Pathway,Editor(s): Vahe Bandarian,Methods in Enzymology,Academic Press, Volume 606, 2018, Pages 389-420, ISSN 0076-6879, ISBN 9780128127940, https://doi.org/10.1016/bs.mie.2018.04.002.
Nicolet, Yvain 2020/04/01 Structure–function relationships of radical SAM enzymes Nature Catalysis https://doi.org/10.1038/s41929-020-0448-7