
В данном практикуме нам предложмлм попробовать собрать геном. В качестве примера была выбрана бактерия Buchnera aphidicola. Для начала работы были скачаны чтения при помощи команды:
wget ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR424/001/SRR4240361/SRR4240361.fastq.gz
Теперь необходимо проверить качество чтений (ссылка). Как видно, качество чтений не очень хорошее, так что необходимо будет произвести триммирование. Для начала необходимо было собрать все адаптеры в 1 файл (файл) и провести триммирование (лог):
cat /mnt/scratch/NGS/adapters/* > adapters.fasta
java -jar /usr/share/java/trimmomatic.jar SE -threads 20 SRR4240361.fastq.gz trimadapt_SRR4240361.fastq.gz
ILLUMINACLIP:adapters.fasta:2:7:7 2> trimadapt.err
Затем была проведена проверка качества (ссылка). Из файла видно, что было удалено 34532 (0.47%) адаптеров (качество улучшилось, но не очень сильно).
Теперь надо удалить с правых концов чтений нуклеотиды с качеством ниже 20 и оставить только такие чтения, длина которых не меньше 32 нуклеотидов:
java -jar /usr/share/java/trimmomatic.jar SE -threads 20 trimadapt_SRR4240361.fastq.gz
trim_SRR4240361.fastq.gz TRAILING:20 MINLEN:32 2> trim.err
Из полученного (файла) видно, что было удалено 403754 (5.58%) последовательностей. Затем, опять же, была проведена проверка качества (ссылка). Качество улучшилось (хотя всё ещё странное).
Теперь, приступаем к самой сборке:
velveth velvet/ 31 -fastq.gz -short trim_SRR4240361.fastq.gz
velvet/ — директория для сборки, 31 — размер k-мера для построения графа, -short — указание на короткие непарные прочтения.
Теперь надо проанализировать качество сборки. Из (log-файла) узнали, что полученный граф имеет 477 узлов и N50=25683. Теперь найдём 3 самых длинных контига (файл) и запишем их покрытие.
| № | Длина | Покрытие |
|---|---|---|
| 6 | 49238 | 26.660851 |
| 2 | 45555 | 26.450466 |
| 34 | 43866 | 23.514977 |
| 352 | 5 | 93.200000 |
| 372 | 1 | 93.000000 |
Также наблюдается 2 контига с аномально большим покрытием, но, учитывая их мерность, это, скорее всего, только шум
Теперь посмотрим, как контиги лягут на хромосому. При помощи "seqret" было создано 3 fasta файла, которые были загружены в megablast.
| № | Координаты участка на хромосоме | Число однонуклеотидных различий | Число гэпов | Ссылка на файл |
|---|---|---|---|---|
| 6 | 127825-140555 153752161738 144368-151796 |
2711 1549 1434 |
50 25809 16429 |
(файл) |
| 2 | 474667-467412 467421-462496 480660-474844 |
14 991 1295 |
168 135 196 |
(файл) |
| 34 | 266073-275551 275566-283706 260224-263784 |
1688 1596 728 |
274 323 83 |
(файл) |
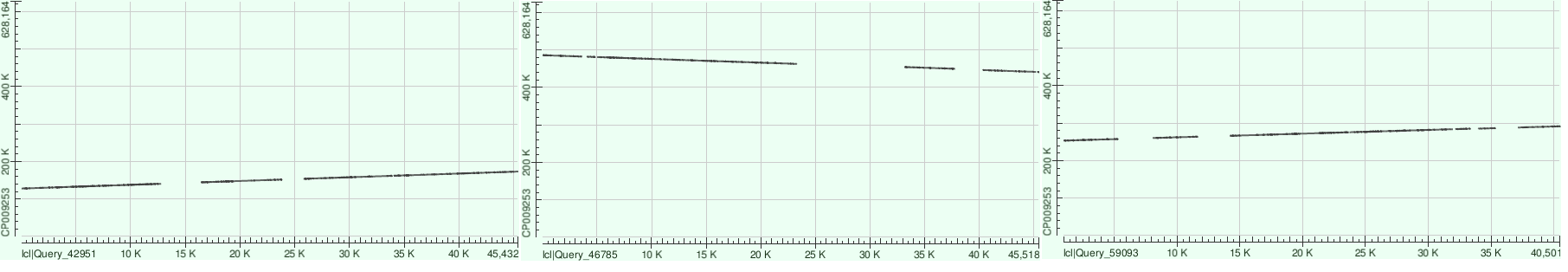
Рисунок 1. Dot plot для 6, 2 и 34 контигов соответственно
Все контиги легли достаточно хорошо. Прерывистость объясняется наличием вариабельных участков.