 8 (926) 907 94 08
8 (926) 907 94 08
Всё на свете является чудом!
Понятие о выравнивании. Эволюционное выравнивание.
- cкопировали пару коротких последовательностей в fasta-формате из таблицы в файл shortseqs.fasta.
- запустили программу GeneDoc и импортировали этот файл.
- выровняли последовательности, стараясь, чтобы было сопоставлено максимальное число одинаковых букв.
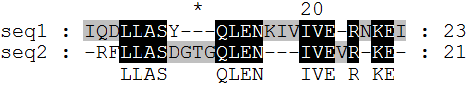
- сохранили выравнивание под именем alignment1.msf.
Рассчитываем процент идентичности двух последовательностей:
A - (общее число колонок выравнивания) -> 27
B - (число колонок с одинаковыми буквами) -> 14
PI - (процент идентичности) -> [B/A] * 100% = [14/27] * 100% = 51.85 %
Рассчитываем процент сходства двух последовательностей, используя матрицу сходства BLOSUM62:
A - (общее число колонок выравнивания) -> 27
B - (число колонок с одинаковыми буквами) -> 14
C - (число колонок с буквами, соответствующими сходным остаткам) -> 1 (колонка Q/R с положительным значением в матрице сходства)
PS - (процент сходства) -> [(B+C)/A] * 100% = [(14+1)/27] * 100% = 55.56 %
2. Построение карты локального сходства последовательностей из задания 1, пользуясь возможностями Excel
- заносим обе последовательности в таблицу Exele.
- чтобы вставить последовательность по букве в каждую ячейку мы можем написать формулу, используя функцию ПСТР (MID); или во вкладке "Данные" выбрать "Текст по столбцам", а для разбиения по строчкам выполнить тоже самое и затем транспонировать.
- для отметки совпадений букв цифрой "1" мы также можем написать формулу, используя функции СОВПАД (EXACT) и ЕСЛИ (IF).
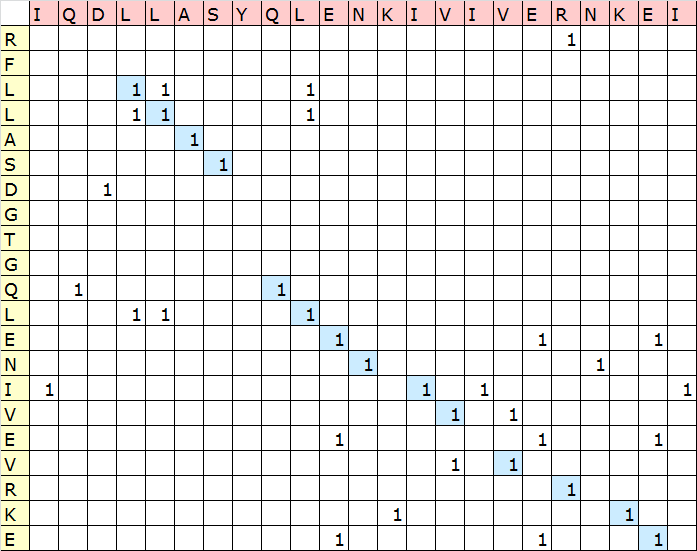
Пояснения к таблице:
Не стоит сильно удивляться карте, которая получилась, так как при 50%-м сходстве мы и не должны ожидать чего-то большего, хотя и хотелось конечно...
Итак, перейдём к пояснениям:
- в идеале, подчёркиваю в идеале, мы должны были получить красивую диагональ из единиц, идущую слева направо, сверху вниз. А на нашей карте мы мало того что не видим красивой диагонали, мы и диагональ то видим с трудом... Но, что есть то есть.
- причина столь неидеальной карты сходства кроется в выравнивании этих двух последовательностей, где количество гэпов почти равно количеству колонок с одинаковыми буквами
- на карте мы видим что "диагональ" начинается лишь с соответствия L-L, перед которым остаётся приличный пробел из-за несоответствия букв R и F буквам I, Q и D из-за чего в выравнивании мы и видим эти гэпы.
Далее идёт один из двух самых лучших участков единиц, целых 4 подряд, следовательно на этом участке в выравнивании было соответствие букв.
Затем на карте мы наблюдаем резкий "обрыв" линии единиц из-за очередного несоответствия букв, на этот раз диагональ сдвинулась на четыре клетки вниз, что напрочь убило надежды на то что наша "диагональ" хоть немного приблизится к идеальной...
После этого идёт второй замечательный участок единиц, а вот дальнейшее продолжение настолько грустно и печально, что из-за обилия слова гэп в соответствующем описании, я решил закончить объяснении карты на последнем замечательном участке из 4-х единиц.
3. Выравнивание первого фрагмента из задания 1 с последовательностью белка THIS_BACSU, пользуясь программой bl2seq
Используя программу bl2seq, мы можем построить частичные выравнивания, но программу имеет смысл применять только для выравнивания одинаковых или очень сходных последовательностей, что мы собственно и делаем.
- переходим на сайт NCBI BLAST, где bl2seq реализована как сервис
- переходим по ссылке Align внизу страницы, а затем выбираем вкладку blastp (blast protein - выровнять белковые последовательности)
- далее в верхнее окошко ("Enter Query Sequence") копируем последовательность моего белка в fasta-формате, а в окошко "Enter Subject Sequence" вписываем первую последовательность из задания 1, которая является фрагментом последовательности моего белка
- нажимаем на большую синюю кнопку "BLAST" и получаем результат, из которого нам необходима только информация о выравнивании
>lcl|64185 unnamed protein product Length=23 Score = 77.4 bits (175), Expect = 3e-24 Identities = 23/23 (100%), Positives = 23/23 (100%), Gaps = 0/23 (0%) Query 18 IQDLLASYQLENKIVIVERNKEI 40 IQDLLASYQLENKIVIVERNKEI Sbjct 1 IQDLLASYQLENKIVIVERNKEI 23Из строчки "Query" делаем вывод, что координаты последовательности фрагмента в полной последовательности белка: 18 - 40.
4. Выравнивание последовательности моего белка с последовательностью гомологичного (родственного) белка, пользуясь сервисом bl2seq
- для выполнения этого выравнивания снова переходим на сайт NCBI BLAST с сервисом bl2seq
- теперь в окошко "Query" вписываем AC моего белка THIS_BACSU (O31617), а в "Subject" - ID его гомолога AP1G_DICDI (Q8I8U2).
Но, как оказалось, не всё так просто. Всеми любимый сервис bl2seq устроил нам небольшую проблему, не выдав на наш запрос ничего, хотя кое-что он всё-таки предоставил...
Я просто не могу не прокомментировать полученный результат, хоть результатом это и трудно назвать...
Во-первых, он (сервис) всё-таки понял, что необходимо выровнять две последовательности, это не может не радовать:)
Во-вторых, он дважды написал о предупреждении, непонятно только для чего.
В-третьих, он таки не смог найти каких-либо существенных сходств у последовательностей.
Но на этом мы не остановились, так как выровнять нам их в любом случае нужно.
- запишем в соответствующие окошки не идентификаторы, а напрямую сами последовательности белков в fasta-формате
- на этот раз сервис "сдался" и выдал нам то что нужно:
>lcl|32701 unnamed protein product Length=895 Score = 30.4 bits (67), Expect = 5e-06, Method: Composition-based stats. Identities = 14/45 (31%), Positives = 25/45 (56%), Gaps = 0/45 (0%) Query 18 IQDLLASYQLENKIVIVERNKEIIGKERYHEVELCDRDVIEIVHF 62 + DL +Y + K ++ ERN +I +E+C+ D +I+HF Sbjct 166 VPDLTENYIPKIKALLSERNHAVILTALTLIIEICEMDSTQIIHF 210Но, честно говоря, многим сервис нас не обеспечил, как минимум описанием обоих белков.
На самом деле для задания необходимо лишь само выравнивание и информация о нём, поэтому ограничимся тем, что есть.
|
ID |
THIS_BACSU |
AP1G_DICDI |
|
Организм, из
которого синтезирован
белок |
Bacillus subtilis
(Сенная палочка) |
Dictyostelium discoideum
(Slime
mold - плесень покрытая слизью) |
|
Процент идентичности
(Identities) |
31 % |
|
|
Процент сходства (Positives) |
56 % |
|
|
Число
символов разрыва (гэпов) |
0 |
|
|
Число символом разрыва, идущих подряд |
||
|
Суммарное число гэповых
колонок |
||
|
Координаты выровненного
участка |
18-62 |
166-210 |
Нажав на "Dot Matrix View" нам раскрывается вкладка с картой локального сходства, которую мы сохраняем в виде gif-файла:
5*. Создание матрицы, в ячейке которой стоит вес замены соответствующих остатков в строке и столбце, для
последовательностей из задания 1
- для выполнения задания веса берём из матрицы весов замен BLOSUM62
- строим матрицу, используя Exele
| I | Q | D | L | L | A | S | Y | Q | L | E | N | K | I | V | I | V | E | R | N | K | E | I | |
| R | -3 | 1 | -2 | -2 | -2 | -1 | -1 | -2 | 1 | -2 | 0 | 0 | 2 | -3 | -3 | -3 | -3 | 0 | 5 | 0 | 2 | 0 | -3 |
| F | 0 | -3 | -3 | 0 | 0 | -2 | -2 | 3 | -3 | 0 | -3 | -3 | -3 | 0 | -1 | 0 | -1 | -3 | -3 | -3 | -3 | -3 | 0 |
| L | 2 | -2 | -4 | 4 | 4 | -1 | -2 | -1 | -2 | 4 | -3 | -3 | -2 | 2 | 1 | 2 | 1 | -3 | -2 | -3 | -2 | -3 | 2 |
| L | 2 | -2 | -4 | 4 | 4 | -1 | -2 | -1 | -2 | 4 | -3 | -3 | -2 | 2 | 1 | 2 | 1 | -3 | -2 | -3 | -2 | -3 | 2 |
| A | -1 | -1 | -2 | -1 | -1 | 4 | 1 | -2 | -1 | -1 | -1 | -2 | -1 | -1 | 0 | -1 | 0 | -1 | -1 | -2 | -1 | -1 | -1 |
| S | -2 | 0 | 0 | -2 | -2 | 1 | 4 | -2 | 0 | -2 | 0 | 1 | 0 | -2 | -2 | -2 | -2 | 0 | -1 | 1 | 0 | 0 | -2 |
| D | -3 | 0 | 6 | -4 | -4 | -2 | 0 | -3 | 0 | -4 | 2 | 1 | -1 | -3 | -3 | -3 | -3 | 2 | -2 | 1 | -1 | 2 | -3 |
| G | -4 | -2 | -1 | -4 | -4 | 0 | 0 | -3 | -2 | -4 | -2 | 0 | -2 | -4 | -3 | -4 | -3 | -2 | -2 | 0 | -2 | -2 | -4 |
| T | -1 | -1 | -1 | -1 | -1 | 0 | 1 | -2 | -1 | -1 | -1 | 0 | -1 | -1 | 0 | -1 | 0 | -1 | -1 | 0 | -1 | -1 | -1 |
| G | -4 | -2 | -1 | -4 | -4 | 0 | 0 | -3 | -2 | -4 | -2 | 0 | -2 | -4 | -3 | -4 | -3 | -2 | -2 | 0 | -2 | -2 | -4 |
| Q | -3 | 5 | 0 | -2 | -2 | -1 | 0 | -1 | 5 | -2 | 2 | 0 | 1 | -3 | -2 | -3 | -2 | 2 | 1 | 0 | 1 | 2 | -3 |
| L | 2 | -2 | -4 | 4 | 4 | -1 | -2 | -1 | -2 | 4 | -3 | -3 | -2 | 2 | 1 | 2 | 1 | -3 | -2 | -3 | -2 | -3 | 2 |
| E | -3 | 2 | 2 | -3 | -3 | -1 | 0 | -2 | 2 | -3 | 5 | 0 | 1 | -3 | -2 | -3 | -2 | 5 | 0 | 0 | 1 | 5 | -3 |
| N | -3 | 0 | 1 | -3 | -3 | -2 | 1 | -2 | 0 | -3 | 0 | 6 | 0 | -3 | -3 | -3 | -3 | 0 | 0 | 6 | 0 | 0 | -3 |
| I | 4 | -3 | -3 | 2 | 2 | -1 | -2 | -1 | -3 | 2 | -3 | -3 | -3 | 4 | 3 | 4 | 3 | -3 | -3 | -3 | -3 | -3 | 4 |
| V | 3 | -2 | -3 | 1 | 1 | 0 | -2 | -1 | -2 | 1 | -2 | -3 | -2 | 3 | 4 | 3 | 4 | -2 | -3 | -3 | -2 | -2 | 3 |
| E | -3 | 2 | 2 | -3 | -3 | -1 | 0 | -2 | 2 | -3 | 5 | 0 | 1 | -3 | -2 | -3 | -2 | 5 | 0 | 0 | 1 | 5 | -3 |
| V | 3 | -2 | -3 | 1 | 1 | 0 | -2 | -1 | -2 | 1 | -2 | -3 | -2 | 3 | 4 | 3 | 4 | -2 | -3 | -3 | -2 | -2 | 3 |
| R | -3 | 1 | -2 | -2 | -2 | -1 | -1 | -2 | 1 | -2 | 0 | 0 | 2 | -3 | -3 | -3 | -3 | 0 | 5 | 0 | 2 | 0 | -3 |
| K | -3 | 1 | -1 | -2 | -2 | -1 | 0 | -2 | 1 | -2 | 1 | 0 | 5 | -3 | -2 | -3 | -2 | 1 | 2 | 0 | 5 | 1 | -3 |
| E | -3 | 2 | 2 | -3 | -3 | -1 | 0 | -2 | 2 | -3 | 5 | 0 | 1 | -3 | -2 | -3 | -2 | 5 | 0 | 0 | 1 | 5 | -3 |
6*. Сравнение выравниваний последовательностей из задания 4, построенных с разными параметрами программы
bl2seq
- на этот раз перед тем как нажать на кнопку "Blast" и произвести выравнивание, мы откроем вкладку "Algorithm parameters", которая находится как раз под кнопкой начала выравнивания
- открыв её, изменим матрицу BLOSUM62 на матрицу PAM30 в разделе "Scoring Parameters". Мы получили два участка выравнивания вместо одного как было ранее, и на втором участке, например, процент идентичности увеличился до 60%, а сходства до 100%
- произведя то же выравнивание, но уже с матрицей BLOSUM80, процент идентичности останется равным 31% как и было с матрицей BLOSUM62, а процент сходства уменьшится до 53%
- изменив штрафы за гэпы, никаких изменений мы не увидим, так как в нашем выравнивании гэпов нет
Чтобы определить насколько одинаковы два выравнивания (одних и тех же последовательностей), можно использовать процент согласованных столбцов двух выравниваний относительно числа столбцов в каждом из выравниваний. В данном случае при изменении матрицы BLOSUM62 на BLOSUM80 процент согласованных столбцов равен [(A/B)*100%] = [(24/25)*100%] = 96% (где A - число столбцов с одинаковыми буквами для матрицы BLOSUM80, а B - для матрицы BLOSUM62).
7*. Импорт последовательности, которую мы выравнивали в обязательном упражнении 4, в GeneDoc.
Воспроизведение вручную выравнивания, полученного программой bl2seq.
Посмотрим выравнивание последовательностей, выполненное при помощи сервиса bl2seq:
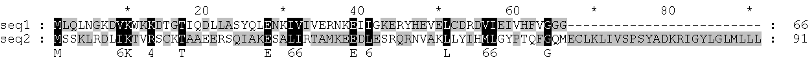
Программа bl2seq выдала нам частичное выравнивание, а нам нужно сделать полное, поэтому мы импортируем последовательности обоих белков в GeneDoc и против всех остатков, не вошедших в частичные выравнивания, вручную ставим гэпы.

Добавив гэпы, мы получили дополнительно 9 соответствий букв L-L, D-D, A-A, Q-Q, R-R, K-K, E-E, R-R, V-V. Это увеличило процент идентичности и сходства выравниваемых последовательностей и, следовательно, дало нам более полное выравнивание.
| Главная | Об авторе | Учебные семестры | Проекты автора | Друзья | Ссылки партнеров | Extra | Контакты |
Mneff © 2011-2012