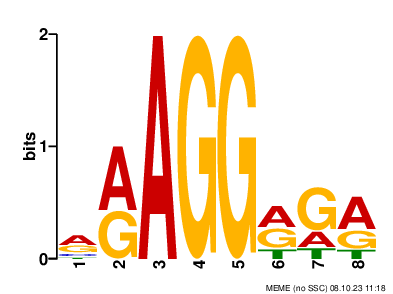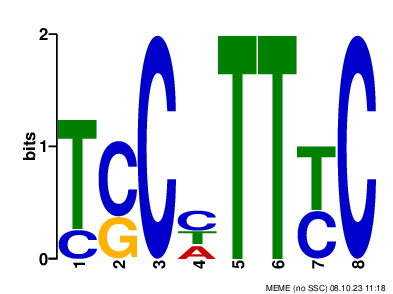Сигналы и мотивы, часть 2
Подготовка данных
Для решения данной задачи был использован геном бактерии Bacillus subtilis штамм DSM 10. При помощи онлайн-сервиса Operon-mapper были получены координаты оперонов этой бактерии. В качестве промотора были выбраны 100 нуклеотидных последовательностей расположенные перед оперонами. Был использован скрипт, написанный Петром Милейко, который на основе ключевых слов в описании оперона находит гены домашнего хозяйства и сохраняет 100 областей промоторов оперонов с этими генами в файл материала обучения. Также скрипт сохраняет в отдельный файл тестовой выборки 2286 промоторных последовательностей всех оперонов. Для негативного контроля были выбраны 2286 нуклеотидных последовательностей в 100 нуклеотидов расположенных после каждого промотора.
MEME
Для запуска использовался локальный MEME:
meme house_keeping_promoters.txt -dna -mod zoops -nmotifs 3 -minw 6 -maxw 50
В результате работы этой команды были получены три мотива, logo которых представлены ниже:
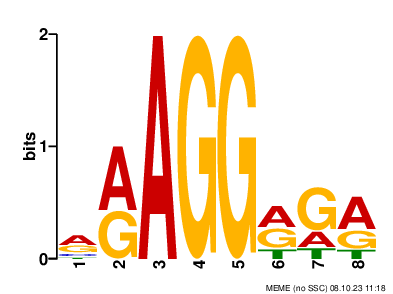 |
| Рис. 1: Logo мотива RRAGGRGR, E-value: 2.0e-014 |
 |
| Рис. 2: Logo мотива MNTTTTTNDYMWTTKTTYTWWMDAWHC, E-value: 1.0e-002 |
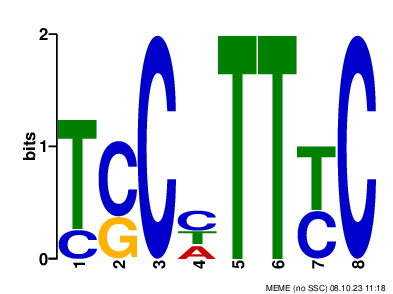 |
| Рис. 3: Logo мотива TSCHTTYC, E-value: 8.0e+000 |
Только первый и второй мотивы имеют p-value меньше 0.05, поэтому дальнейшая работа проводилась с ними.
FIMO
В начале, в положительном и отрицательном контролях, производился поиск первого мотива:
fimo --norc -motif RRAGGRGR -thresh 0.001 meme_out/meme.txt all_promoters.fasta
fimo --norc -motif RRAGGRGR -thresh 0.001 meme_out/meme.txt negative_cont.fasta
В результате в 1350 последовательностях тестовой выборки был обнаружен сигнал, то есть более чем в половине всех промоторов. И лишь 318 сигналов было найдено в выборке негативного контроля.
Так же в мотиве RRAGGRGR по его logo можно углядеть AGGagg (последовательность, схожая с последовательностью Шайна — Дальгарно), так что ради интереса был произведён поиск по этой последовательности:
fimo --norc -motif AGGAGG -thresh 0.001 meme_out/meme.txt all_promoters.fasta
fimo --norc -motif AGGAGG -thresh 0.001 meme_out/meme.txt negative_cont.fasta
И в итоге было найдено 0 таких сигналов в обоих случаях.
Далее производился поиск второго мотива:
fimo --norc -motif MNTTTTTNDYMWTTKTTYTWWMDAWHC -thresh 0.001 meme_out/meme.txt all_promoters.fasta
fimo --norc -motif MNTTTTTNDYMWTTKTTYTWWMDAWHC -thresh 0.001 meme_out/meme.txt negative_cont.fasta
В результате в 1084 последовательностях тестовой выборки был обнаружен сигнал, то есть почти в половине всех промоторов. Однако 455 сигналов было найдено в выборке негативного контроля, что многовато.
Из-за этого был произведён поиск с меньшим порогом:
fimo --norc -motif MNTTTTTNDYMWTTKTTYTWWMDAWHC -thresh 0.0001 meme_out/meme.txt all_promoters.fasta
fimo --norc -motif MNTTTTTNDYMWTTKTTYTWWMDAWHC -thresh 0.0001 meme_out/meme.txt negative_cont.fasta
В этот раз в тестовой выборке было найдено 206 сигналов, а в негативном контроле 70 сигналов.
|