- Сравнение нумерации остатков белка-прототипа в UniProt и PDB
На страничке БД PDBsum, предоставляющей краткое схематическое отображение информации о структуре, находим документ с идентификатором PDB 2ZW3. Получили выравнивание последовательности из UniProt и последовательности из PDB.
Импортировали выравнивание в Genedoc: pdbsum.msf.

Нумерация в 2-х БД совпадает. - Построение полного глобального выравнивания заданного белка CXB2_RAT и белка-прототипа CXB2_HUMAN.
Оба белка принадлежат к семейству коннексинов, один, как видно из названия, из организма крысы, другой - из организма человека.
Получили последовательности белков для выравнивания из UniProt: cxb2_rat.fasta, cxb2_human.fasta
Глобальное выравнивание построили с помощью программы needle: tm.needle - Создать по данным БД ОРМ разметку трансмембранных сегментов в белке-прототипе CXB2_HUMAN.
На страничке БД ОРМ,
по PDB ID 2ZW3 нашли описание ТМ-сегментов в белке-прототипе CXB2_HUMAN.
Описание белка: * Тип: 1. трансмембранный белок (2 classes) * Класс: 1.1. Альфа-спиральный трансмембранный белок (57 superfamilies) * Суперсемейство: 1.1.52. Коннексин, формирующий щелевые контакты (нексусы) (1 family) 1.A.24 (TCDB) * Семейство: 1.1.52.01. Коннексин (1 protein) 1.A.24 (TCDB) * Виды: Spodoptera frugiperda (1 protein) * Локализация: Плазматическая мембрана эукариот (199 proteins)Белок состоит из 6 суъединиц, в каждой из которых найдено 6 трансмембранных сегментов.A - Tilt: 20° - Segments: 1( 22- 45), 2( 73- 91), 3( 137- 157), 4( 187- 211) B - Tilt: 19° - Segments: 1( 22- 45), 2( 73- 91), 3( 137- 157), 4( 187- 211) C - Tilt: 19° - Segments: 1( 22- 45), 2( 73- 91), 3( 137- 157), 4( 187- 211) D - Tilt: 19° - Segments: 1( 22- 45), 2( 73- 91), 3( 137- 157), 4( 187- 211) E - Tilt: 19° - Segments: 1( 22- 45), 2( 73- 91), 3( 137- 157), 4( 187- 211) F - Tilt: 19° - Segments: 1( 22- 45), 2( 73- 91), 3( 137- 157), 4( 187- 211)
Интересно, что изображение в Jmol показывает фрагмент 1-8 аминокислоты как трансмембранный, тогда как по данным OPM это не так.Разметка была сделана согласно OPM: позиции мембранных сегментов отмечены буквой "Н", позиции цитоплазматических петель знаком "+", остальные — знаком "-".
Выравнивание сохранено в файл: mark.msf
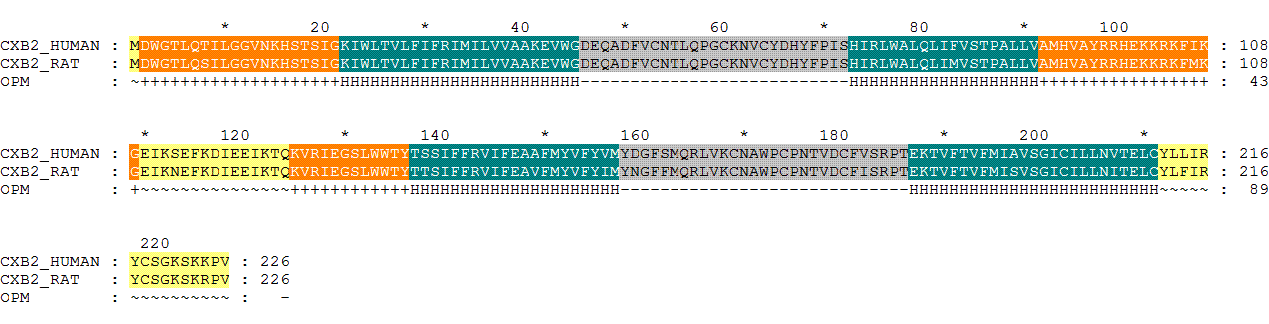
Оранжевым раскрашены цитоплазматические позиции, зеленым - трансмембранные сегменты, желтым - последовательность, которой нет в OPM и PDBsum, остальное - серым. - Предсказать топологию заданного белка с помощью наиболее популярной программы (TMHMM)
Для предсказания топологии заданного белка CXB2_RAT используем сервер TMHMM. (опции по умолчанию).
На вход программе нужно подать последовательность белка в fasta-формате.
Полученный результат, искусственную последовательность, с разметкой трансмембранных спиралей в соответствии с результатами предсказания добавили к файлу с выровненными последовательностями: mark.msf
Посмотреть выравнивание (пояснения к выбранным цветам и обозначениям см.выше).
CXB2_HUMAN 1 MDWGTLQTILGGVNKHSTSIGKIWLTVLFIFRIMILVVAAKEVWGDEQAD 50
|||||||:||||||||||||||||||||||||||||||||||||||||||
CXB2_RAT 1 MDWGTLQSILGGVNKHSTSIGKIWLTVLFIFRIMILVVAAKEVWGDEQAD 50
CXB2_HUMAN 51 FVCNTLQPGCKNVCYDHYFPISHIRLWALQLIFVSTPALLVAMHVAYRRH 100
||||||||||||||||||||||||||||||||.|||||||||||||||||
CXB2_RAT 51 FVCNTLQPGCKNVCYDHYFPISHIRLWALQLIMVSTPALLVAMHVAYRRH 100
CXB2_HUMAN 101 EKKRKFIKGEIKSEFKDIEEIKTQKVRIEGSLWWTYTSSIFFRVIFEAAF 150
||||||:|||||:||||||||||||||||||||||||:||||||||||.|
CXB2_RAT 101 EKKRKFMKGEIKNEFKDIEEIKTQKVRIEGSLWWTYTTSIFFRVIFEAVF 150
CXB2_HUMAN 151 MYVFYVMYDGFSMQRLVKCNAWPCPNTVDCFVSRPTEKTVFTVFMIAVSG 200
|||||:||:||.|||||||||||||||||||:||||||||||||||:|||
CXB2_RAT 151 MYVFYIMYNGFFMQRLVKCNAWPCPNTVDCFISRPTEKTVFTVFMISVSG 200
CXB2_HUMAN 201 ICILLNVTELCYLLIRYCSGKSKKPV 226
||||||:||||||.|||||||||:||
CXB2_RAT 201 ICILLNITELCYLFIRYCSGKSKRPV 226
Идентичность: 212/226 (93.8%)
Сходство: 222/226 (98.2%)
Гэпов нет