Ещё работа с BLAST и карты локального сходства
Поиск отличий между двумя разными выравниваниями одних и тех же последовательностей
Для сравнения выравниваний была выбрана метиониновая аминопептидаза I - фермент,который отщепляет N-концевой аминокислотный остаток метионина у пептидов. Для
сравнения была выбрана аминопептидаза палочки Коха(Mycobacterium tuberculosis) и человека(
Homo sapiens). Было произведено два выравнивания с помощью
двух разных программ.
Первое выравнивание было произведено в рамках множественного выравнивания в программе muscle - для этого были выбраны ещё две последовательности, принадлежащие
сенной палочке(Bacillus subtilis (strain 168)) и пекарским дрожжам (Saccharomyces cerevisiae). После множественного выравнивания из проекта JalView были удалены все последовательности, кроме белков палочки Коха и человека.
Второе выравнивание было сделано с помощью опции Align в UniProt. После выполнения этой опции был скачан fasta-файл с результатами и также визуализирован в JalView.
Поиск отличий в выравниваний производился их сравнением в JalView. Проект с обоими выравниваниями можно скачать здесь


В таблице ниже приведены места, в которых обнаружены отличия в выравниваниях.
| Выравнивание в Uniprot | Выравнивание в muscle | |
| Отличие №1 | Glu134 - Gly19 | Arg105 - Gly19 |
| Отличие №2 | гэп - Glu170 | Gly284 - Glu170 |
| Отличие №3 | Met94 - гэп | Met94 - Arg8 |
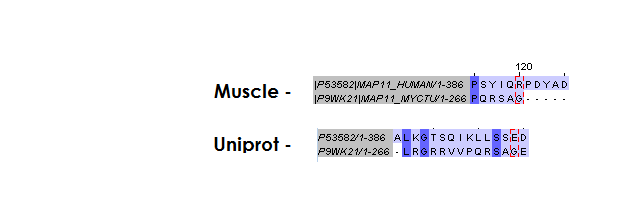

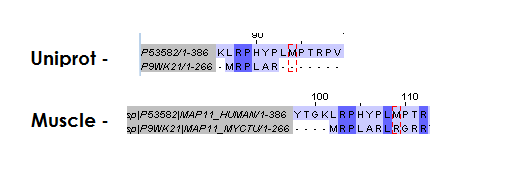
Вообще, выравнивания достаточно сильно похожи(что логично, т.к. последовательности одинаковые), единственные отличия заключаются в разном расположении гэпов, из-за которых смещается соответствие между аминокислотами.
Карта локальных сходств двух белков
Для построения карты локального сходства были выбраны полипротеины полиовируса
Poliovirus type 1 (strain Mahoney) и ящура Foot-and-mouth disease virus (strain A10-61) (Aphthovirus A). Их аминокислотные последовательности были выровнены в BLAST.
Ниже представлена таблица с основной информацией об этих белках.Выдачу BLAST можно посмотреть
здесь.
| Poliovirus type 1 | Foot-and-mouth disease virus | |
| UniProt ID | POLG_POL1M | POLG_FMDV1 |
| UniProt AC | P03300; A0A142KD04; P03299; Q84879; Q84880; Q89679; | P03306; Q64768; Q84750; Q84751; Q84752; Q84753; Q84754; Q84760; Q84761; Q84762; Q84763; Q84764; Q84765; Q84766; Q84767; Q84768; Q84769; Q89824; |
| Full name | Genome polyprotein | |
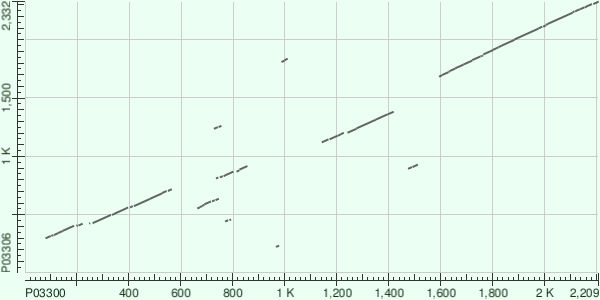
BLAST выдал 10 локальных выравниваний с разным весом и E-value. Они представлены на рисунке 7. Можно видеть 4 достаточно больших участка, на которых были выровнены
последовательности и 6 маленьких участков, которые, по большому счёту, не нужны, т.к. они короткие и имеют большое значение E-value( от 1.1 и выше)
Поэтому выравнивание было проведено ещё раз, но с другим порогом Expect threshold, равным 0.0001. Полученную выдачу можно посмотреть здесь. С таким строгим значением мы получили более чистую карту.
Ниже представлено лучшее локальное выравнивание из BLAST.
Score:250 bits(638), Expect:5e-70,
Method:Compositional matrix adjust.,
Identities:189/659(29%), Positives:322/659(48%), Gaps:59/659(8%)
Query 1594 GVHDNVAILPTH--ASPGESIVIDGKEVEILDAKALEDQ---AGTNL--EITIITLKRNE 1646
GV ++P H A + I++DG+ + D + E + G ++ + +I L R
Sbjct 1684 GVFGTAYLVPRHLFAEKYDKIMLDGRAMTDSDYRVFEFEIKVKGQDMLSDAALIVLHRGN 1743
Query 1647 KFRDIRPHI--PTQITETNDGVLIVNTSKYPNMYVPVGAVTEQG-YLNLGGRQTARTLMY 1703
RDI H ++ + V +VN + + A+T + + + G Y
Sbjct 1744 CVRDITKHFRDTARMKKGTPVVGVVNNADVGRLIFSGEALTYKDIVVCMDGDTMPGLFAY 1803
Query 1704 NFPTRAGQCGGVITCTGK----VIGMHVGGNGSHGFAAALKRSYFTQSQGEIQWMRPSKE 1759
TRAG CGG + ++G H G G+ + + RS + + + P E
Sbjct 1804 KAATRAGYCGGAVLAKDGADTFIVGTHSAGGNGVGYCSCVSRSMLQKMKAHVD-PEPHHE 1862
Query 1760 VGYPI--------INAPSKTKLEPSAFHYVFEGVKEPAVLTKNDPRLKTD--FEEAIFSK 1809
G + ++ KTKL P+ + VF PA L+ DPRL ++ IFSK
Sbjct 1863 -GLIVDTRDVEERVHVMRKTKLAPTVAYGVFNPEFGPAALSNKDPRLNEGVVLDDVIFSK 1921
Query 1810 YVGN-KITEVDEYM-KEAVDHYAGQLMS-LDINTEQMCLEDAMYGTDGLEALDLSTSAGY 1866
+ G+ K+TE D+ + + YA +L S L + + +A+ G DGL+A++ T+ G
Sbjct 1922 HKGDAKMTEEDKALFRRCAADYASRLHSVLGTANAPLSIYEAIKGVDGLDAMEPDTAPGL 1981
Query 1867 PYVAMGKKKRDILNKQTR----DTKEMQKLLDTYGINLPLVTYVKDELRSKTKVEQGKSR 1922
P+ GK++ +++ + + + KL++ T++KDE+R KV GK+R
Sbjct 1982 PWALQGKRRGALIDFENGTVGPEVEAALKLMEKREYKFACQTFLKDEIRPMEKVRAGKTR 2041
Query 1923 LIEASSLNDSVAMRMAFGNLYAAFHKNPGVITGSAVGCDPDLFWSKIPVLMEE--KLFAF 1980
+++ + + +M G A H N G GSAVGC+PD+ W + + ++
Sbjct 2042 IVDVLPVEHILYTKMMIGRFCAQMHSNNGPQIGSAVGCNPDVDWQRFGTHFAQYRNVWDV 2101
Query 1981 DYTGYDASLSPAWFEALKMVLEKI-----GFGDRVDYI-DYLNHSHHLYKNKTYCVKGGM 2034
DY+ +DA+ +A+ ++ E++ GF ++I L ++ H Y+NK V+GGM
Sbjct 2102 DYSAFDANHCS---DAMNIMFEEVFRTDFGFHPNAEWILKTLVNTEHAYENKRITVEGGM 2158
Query 2035 PSGCSGTSIFNSMINNLIIRTLLLKTYKGIDLDHLKMIAYGDDVIASYPHEVDASLLAQS 2094
PSGCS TSI N+++NN+ + L + Y+G++LD MI+YGDD++ + +++D L
Sbjct 2159 PSGCSATSIINTILNNIYVLYALRRHYEGVELDTYTMISYGDDIVVASDYDLDFEALKPH 2218
Query 2095 GKDYGLTMTPADKS----ATFETVTWENVTFLKRFFRADEKYPFLIHPVMPMKEIHESIR 2150
K G T+TPADKS +++T +VTFLKR F D F PVM K + +
Sbjct 2219 FKSLGQTITPADKSDKGFVLGQSIT--DVTFLKRHFHMDYGTGFY-KPVMASKTLEAILS 2275
Query 2151 WTKDPRNT-QDHVRSLCLLAWHNGEEEYNKFLAKIRSVPIGRALLLPEYSTLYRRWLDS 2208
+ + R T Q+ + S+ LA H+G +EY + P +P Y +LY RW+++
Sbjct 2276 FAR--RGTIQEKLISVAGLAVHSGPDEYRRLFE-----PFQGLFEIPSYRSLYLRWVNA 2327
В поле FT было сказано, что в участках полипротеинов, которые попали в выравнивание, содержатся:
У полиовируса: Protein 3CD, Protease 3C, RNA-directed RNA polymerase
У вируса ящура: Protease 3C, RNA-directed RNA polymerase
То есть выровненные участки кодируют функционально одинаковые белки, это может свидетельствовать о гомологии этих полипротеинов.
К тому же в других локальных выравниваниях полипротеинов тоже оказываются функционально схожие белки. Не очень понятно, как обозначить выравнивания из выдачи
BLAST, поэтому в качестве критерия был использован Range. Поиск белков осуществлялся с помощью записи о полипротеинах в UniProt.
| Range | Белки полиовируса,попавшие в выравнивание | Белки вируса ящура, попавшие в выравнивание |
| Range 1 | Protein 3CD, Protease 3C, RNA-directed RNA-polymerase | Protease 3C, RNA-directed RNA-polymerase |
| Range 2 | Protein 2C | Protein 2C |
| Range 3 | Капсидные белки VP3, VP4 | Белки VP3, VP2 |
| Range 4 | Капсидный белок VP1 | Белок VP1 |
Затем было проведено локальное выравнивание белков программой water [ссылка]. Важно отметить, что штрафы за открытие и продолжение гэпа были выставлены по умолчанию, то есть 10.0 и 0.5 за открытие и продолжение гэпа соответственно.
########################################
# Program: water
# Rundate: Fri 1 May 2020 10:28:38
# Commandline: water
# -asequence sw:P03300
# -bsequence sw:P03306
# Align_format: srspair
# Report_file: polg_pol1m.water
########################################
#=======================================
#
# Aligned_sequences: 2
# 1: POLG_POL1M
# 2: POLG_FMDV1
# Matrix: EBLOSUM62
# Gap_penalty: 10.0
# Extend_penalty: 0.5
#
# Length: 2494
# Identity: 616/2494 (24.7%)
# Similarity: 957/2494 (38.4%)
# Gaps: 655/2494 (26.3%)
# Score: 1533.5
#
#
#=======================================
POLG_POL1M 2 GAQVSSQKVGAHENSNRAYGGSTINYTTINYYRDSASNAASKQ------- 44
||..||...|:...|... ||.|| |||.....|:.|.|
POLG_FMDV1 202 GAGQSSPATGSQNQSGNT--GSIIN----NYYMQQYQNSMSTQLGDNTIS 245
POLG_POL1M 45 ----DFSQD-------------------PSKFTEPIKDVLIKTAPMLNSP 71
:.|.| .|.||. :..|.:.:..
POLG_FMDV1 246 GGSNEGSTDTTSTHTTNTQNNDWFSKLASSAFTG------LFGALLADKK 289
POLG_POL1M 72 NIEACGYSDRVLQLTLGNSTITTQEAANSVVAYGRWPEYLRDSEANPVDQ 121
..|.....||:|....|::|.|||.:.. |.||...| .|..|.|...
POLG_FMDV1 290 TEETTLLEDRILTTRNGHTTSTTQSSVG--VTYGYSTE--EDHVAGPNTS 335
POLG_POL1M 122 PTEPDVA-ACRFYTLDTVSWTKESR-GWWWKLPDALRDMGLFGQNM-YYH 168
..|..|. |.||:......||.:.. |:..||.......|:||..: .|.
POLG_FMDV1 336 GLETRVVQAERFFKKFLFDWTTDKPFGYLTKLELPTDHHGVFGHLVDSYA 385
POLG_POL1M 169 YLGRSGYTVHVQCNASKFHQGALGVFAVPEMCLAGDSNTTTMHTSYQNAN 218
|: |:|:.|.|....::|:.|.|.|..||| ::..:
POLG_FMDV1 386 YM-RNGWDVEVSAVGNQFNGGCLLVAMVPE---------------WKAFD 419
POLG_POL1M 219 PGEKGGTFTGTFTPDNNQTSPARRFCPVDYLLGNGTLLGNAFVFPHQIIN 268
..|| |.| || ||||.|:
POLG_FMDV1 420 TREK-------------------------YQL---TL------FPHQFIS 435
POLG_POL1M 269 LRTNNCATLVLPYVNSLSIDSMVKHNNWGIAILPLAPLNFASESSPEIPI 318
.|||..|.:.:||:.....|...||..|.:.::.|:||..::.::|:|.:
POLG_FMDV1 436 PRTNMTAHITVPYLGVNRYDQYKKHKPWTLVVMVLSPLTVSNTAAPQIKV 485
POLG_POL1M 319 TLTIAPMCCEFNGLRNITLPRLQGL-PVMNTPGSNQYLTADNFQSPCALP 367
...|||......| .||..:|: ||....|....:|.|...:.....
POLG_FMDV1 486 YANIAPTYVHVAG----ELPSKEGIFPVACADGYGGLVTTDPKTADPVYG 531
POLG_POL1M 368 EFDVTPPIDIPGEVKNMMELAE-IDTMIPFDLSATKKNTMEMYRVRLSDK 416
:....|..:.||...|::::|| ..|.:.|| ..|
POLG_FMDV1 532 KVYNPPKTNYPGRFTNLLDVAEACPTFLRFD----------------DGK 565
POLG_POL1M 417 PHTDDPILCLSLSPASDPRL-------------SHTMLGEILNYYTHWAG 453
|:. ::.|.|.|| |:|.|..|..|||.::|
POLG_FMDV1 566 PYV--------VTRADDTRLLAKFDVSLAAKHMSNTYLSGIAQYYTQYSG 607
POLG_POL1M 454 SLKFTFLFCGFMMATGKLLVSYAPPGAD-PPKKRKEAMLGTHVIWDIGLQ 502
::...|:|.|...:..:.:|:|.|||.: ||...:||....|..||.||.
POLG_FMDV1 608 TINLHFMFTGSTDSKARYMVAYIPPGVETPPDTPEEAAHCIHAEWDTGLN 657
POLG_POL1M 503 SSCTMVVPWISNTTYRQTIDD---SFTEGGYISVFYQTRIVVPLSTPREM 549
|..|..:|::|...|..|..| :....|::.|:..|.......|
POLG_FMDV1 658 SKFTFSIPYVSAADYAYTASDTAETTNVQGWVCVYQITHGKAENDT---- 703
POLG_POL1M 550 DILGFVSACNDFSVRLLRDTTHIEQKALAQGLGQMLESMIDNTVRETVGA 599
:|...||..||.:||..|
POLG_FMDV1 704 -LLVSASAGKDFELRLPID------------------------------- 721
POLG_POL1M 600 ATSRDALPNTEASGPTHSKEIPALTAVETGATNPLVPSDT-VQTRHVVQH 648
|.|:.:....|.: |..|.||. ...|| ||.||
POLG_FMDV1 722 -------PRTQTTTTGESAD-PVTTTVEN------YGGDTQVQRRH---- 753
POLG_POL1M 649 RSRSESSIESFFARGACVTIMTVDNPASTTNKDKLFAVWKITYKDTVQLR 698
..:..|.....|.| |..|.|:...|.. |:|..: :.
POLG_FMDV1 754 ------HTDVGFIMDRFVKI----NSLSPTHVIDLMQ----THKHGI-VG 788
POLG_POL1M 699 RKLEFFTYSRFDMELTFVVTANFTETNNGHALNQVYQIMYVPPGAPVPEK 748
..|...||...|:|:......|.| :||.|||
POLG_FMDV1 789 ALLRAATYYFSDLEIVVRHDGNLT---------------WVPNGAP---- 819
POLG_POL1M 749 WDDYTWQTSSNPSIFYTYGTAP-ARISVPYVGISNAYSHFYDGFSKVPLK 797
:.....:|||: .|..|| .|:::||.......:..|||.:|....
POLG_FMDV1 820 --EAALSNTSNPT---AYNKAPFTRLALPYTAPHRVLATVYDGTNKYSAS 864
POLG_POL1M 798 DQSAALGDSLYGAASLNDFGILAVRVVN------DHNPTKVTSKIRVYLK 841
|..: .|.|.:|.||.. ::...:..:...:.::
POLG_FMDV1 865 DSRS------------GDLGSIAARVATQLPASFNYGAIQAQAIHELLVR 902
POLG_POL1M 842 PKHIRVWCPRPPRAVAYYGPGVDYKDGTLTPLSTKDLTTY------GFGH 885
.|...::||||..|:...... .||...:.| .|.|..: |...
POLG_FMDV1 903 MKRAELYCPRPLLAIKVTSQD-RYKQKIIAP--AKQLLNFDLLKLAGDVE 949
POLG_POL1M 886 QNKA------VYTAGYKICNYHLATQDDLQNAVNVMWSRDLLVTESRAQG 929
.|.. |.:...|:.:.....|:|:.......::|.:...|..|.|
POLG_FMDV1 950 SNLGPFFFADVRSNFSKLVDTINQMQEDMSTKHGPDFNRLVSAFEELATG 999
POLG_POL1M 930 TDSI-----------------ARCNCNAGVYYCESRRKYYPVSFVGPTFQ 962
..:| :|.:|.|.|
POLG_FMDV1 1000 VKAIRTGLDEAKPWYKLIKLLSRLSCMAAV-------------------- 1029
POLG_POL1M 963 YMEANNYYPARYQSHMLIGHGFASPGDCGGILRCHHGVIGIITAGGEGLV 1012
.||.:..:|:....|..|
POLG_FMDV1 1030 --------AARSKDPVLVAIMLADTG------------------------ 1047
POLG_POL1M 1013 AFSDIRDLYAYEEEAMEQGITNYIESLGAAFGSGFTQQISDKITELTNMV 1062
:|.|.:.| ..::.||.::.|.::.
POLG_FMDV1 1048 -----------------------LEILDSTF---VVKKSSDSLSSLFHVP 1071
POLG_POL1M 1063 TSTIT---EKLLKNLIKIISSLVIITRNYEDTTTVLATLALLGCDASPWQ 1109
....: ..||..|:|:.||....|. ||.......|
POLG_FMDV1 1072 APAFSFGAPVLLAGLVKVASSFFRSTP--EDLERAEKQL----------- 1108
POLG_POL1M 1110 WLRKKACDVLEIPYVIKQGDSWLKKFTEACNAAKGLEWVSNKISKFIDWL 1159
||.|:.:|..::|.| ||:...|....||:
POLG_FMDV1 1109 ----KARDINDIFAILKNG-----------------EWLVKLILAIRDWI 1137
POLG_POL1M 1160 KEKIIPQARDKLEFVTKLRQLE-MLENQISTIHQSCPSQEHQEILFNNVR 1208
|..|..:.: |||....:. :||.| ..::.....:|.:|.| :|.|
POLG_FMDV1 1138 KAWIASEEK----FVTMTDLVPGILEKQ-RDLNDPGKYKEAKEWL-DNAR 1181
POLG_POL1M 1209 WLSIQS--KRFAPLYAVEAKRIQKLEHTINNYIQFKSKHRIEPVCLLVHG 1256
...::| ...|.|..|.|.. .||.|.|||.:.:.|
POLG_FMDV1 1182 QACLKSGNVHIANLCKVVAPA--------------PSKSRPEPVVVCLRG 1217
POLG_POL1M 1257 SPGTGKSVATNLIARAIAER---ENTSTYSLPPDPSHFDGYKQQGVVIMD 1303
..|.|||...|::|:||:.. ...|.:..||||.|||||.||.||:||
POLG_FMDV1 1218 KSGQGKSFLANVLAQAISTHFTGRIDSVWYCPPDPDHFDGYNQQTVVVMD 1267
POLG_POL1M 1304 DLNQNPDGADMKLFCQMVSTVEFIPPMASLEEKGILFTSNYVLASTN-SS 1352
||.|||||.|.|.|.|||||..|||||||||:||..|.|..::|:|| .|
POLG_FMDV1 1268 DLGQNPDGKDFKYFAQMVSTTGFIPPMASLEDKGKPFNSKVIIATTNLYS 1317
POLG_POL1M 1353 RISPPTVAHSDALARRFAFDMDIQVMNEYSRDGKLNMAMATEMCKNCHQP 1402
..:|.|:...|||.|||.||:|:...:.|..:.||::..|.| ..:.:..
POLG_FMDV1 1318 GFTPRTMVCPDALNRRFHFDIDVSAKDGYKINNKLDIIKALE-DTHTNPV 1366
POLG_POL1M 1403 ANFKRCCPLVCGKAIQL------MDKSSRVRYSIDQITTMIINER----- 1441
|.|:..|.|:.|.|::: |.|......::.|:...:| ||
POLG_FMDV1 1367 AMFQYDCALLNGMAVEMKRLQQDMFKPQPPLQNVYQLVQEVI-ERVELHE 1415
POLG_POL1M 1442 --------------NRRSNIGNCME--------ALFQG-----------P 1458
:::|.:...:| ..|:| |
POLG_FMDV1 1416 KVSSHPIFKQISIPSQKSVLYFLIEKGQHEAAIEFFEGMVHDSVKEELRP 1465
POLG_POL1M 1459 L---------QYKDLKIDIKTSPPPECINDLL------------QAVDSQ 1487
| .:|.||.:.:. ...|:..|. |.:...
POLG_FMDV1 1466 LIQQTSFVKRAFKRLKENFEI--VALCLTLLANIVIMIRETRKRQKMVDD 1513
POLG_POL1M 1488 EVRDYCEKKGWIVNITSQVQT----ERNINRAMTILQAVTTFAAVAGV-- 1531
.|.:|.|: .|||:..:| |:| ...|:.|:..|.
POLG_FMDV1 1514 AVNEYIER----ANITTDDKTLDEAEKN--------PLETSGASTVGFRE 1551
POLG_POL1M 1532 ---------------------------------------VYVMYKLFAGH 1542
:.|..|| ...
POLG_FMDV1 1552 RSLTGQKVRDDVSSEPAQPAEDQPQAEGPYSGPLERQKPLKVRAKL-PQQ 1600
POLG_POL1M 1543 QGAYTG-LPNKKP-----NVPTIRTAKVQGP-GFDYAVAMAKRNI----- 1580
:|.|.| :..:|| ..|.::....:|| ....|:.:..||:
POLG_FMDV1 1601 EGPYAGPMERQKPLKVKVKAPVVKEGPYEGPVKKPVALKVKARNLIVTES 1650
POLG_POL1M 1581 ----------VTATTSKGEFTM----------LGVHDNVAILPTH--ASP 1608
|...|...|..: .||.....::|.| |..
POLG_FMDV1 1651 GAPPTDLQKMVMGNTKPVELNLDGKTVAICCATGVFGTAYLVPRHLFAEK 1700
POLG_POL1M 1609 GESIVIDGKEVEILDAKALEDQ---AGTNL--EITIITLKRNEKFRDIRP 1653
.:.|::||:.:...|.:..|.: .|.:: :..:|.|.|....|||..
POLG_FMDV1 1701 YDKIMLDGRAMTDSDYRVFEFEIKVKGQDMLSDAALIVLHRGNCVRDITK 1750
POLG_POL1M 1654 HI--PTQITETNDGVLIVNTSKYPNMYVPVGAVTEQG-YLNLGGRQTART 1700
|. ..::.:....|.:||.:....:.....|:|.:. .:.:.|......
POLG_FMDV1 1751 HFRDTARMKKGTPVVGVVNNADVGRLIFSGEALTYKDIVVCMDGDTMPGL 1800
POLG_POL1M 1701 LMYNFPTRAGQCGGVITCTGK----VIGMH-VGGNGSHGFAAALKRSYFT 1745
..|...||||.|||.:..... ::|.| .||||. |:.:.:.||...
POLG_FMDV1 1801 FAYKAATRAGYCGGAVLAKDGADTFIVGTHSAGGNGV-GYCSCVSRSMLQ 1849
POLG_POL1M 1746 QSQGEIQWMRPSKEVGYPI--------INAPSKTKLEPSAFHYVFEGVKE 1787
:.:..:. ..|..| |..: ::...||||.|:..:.||.....
POLG_FMDV1 1850 KMKAHVD-PEPHHE-GLIVDTRDVEERVHVMRKTKLAPTVAYGVFNPEFG 1897
POLG_POL1M 1788 PAVLTKNDPRLKTD--FEEAIFSKYVGN-KITEVDE--YMKEAVDHYAGQ 1832
||.|:..||||... .::.||||:.|: |:||.|: :.:.|.| ||.:
POLG_FMDV1 1898 PAALSNKDPRLNEGVVLDDVIFSKHKGDAKMTEEDKALFRRCAAD-YASR 1946
POLG_POL1M 1833 LMS-LDINTEQMCLEDAMYGTDGLEALDLSTSAGYPYVAMGKKKRDILNK 1881
|.| |......:.:.:|:.|.|||:|::..|:.|.|:...||::..:::.
POLG_FMDV1 1947 LHSVLGTANAPLSIYEAIKGVDGLDAMEPDTAPGLPWALQGKRRGALIDF 1996
POLG_POL1M 1882 QT----RDTKEMQKLLDTYGINLPLVTYVKDELRSKTKVEQGKSRLIEAS 1927
:. .:.:...||::.........|::|||:|...||..||:|:::..
POLG_FMDV1 1997 ENGTVGPEVEAALKLMEKREYKFACQTFLKDEIRPMEKVRAGKTRIVDVL 2046
POLG_POL1M 1928 SLNDSVAMRMAFGNLYAAFHKNPGVITGSAVGCDPDLFWSKIPVLMEE-- 1975
.:...:..:|..|...|..|.|.|...||||||:||:.|.:......:
POLG_FMDV1 2047 PVEHILYTKMMIGRFCAQMHSNNGPQIGSAVGCNPDVDWQRFGTHFAQYR 2096
POLG_POL1M 1976 KLFAFDYTGYDASLSPAWFEALKMVLEKI-----GFGDRVDYI-DYLNHS 2019
.::..||:.:||:... :|:.::.|:: ||....::| ..|.::
POLG_FMDV1 2097 NVWDVDYSAFDANHCS---DAMNIMFEEVFRTDFGFHPNAEWILKTLVNT 2143
POLG_POL1M 2020 HHLYKNKTYCVKGGMPSGCSGTSIFNSMINNLIIRTLLLKTYKGIDLDHL 2069
.|.|:||...|:||||||||.|||.|:::||:.:...|.:.|:|::||..
POLG_FMDV1 2144 EHAYENKRITVEGGMPSGCSATSIINTILNNIYVLYALRRHYEGVELDTY 2193
POLG_POL1M 2070 KMIAYGDDVIASYPHEVDASLLAQSGKDYGLTMTPADKS----ATFETVT 2115
.||:||||::.:..:::|...|....|..|.|:|||||| ...:::|
POLG_FMDV1 2194 TMISYGDDIVVASDYDLDFEALKPHFKSLGQTITPADKSDKGFVLGQSIT 2243
POLG_POL1M 2116 WENVTFLKRFFRADEKYPFLIHPVMPMKEIHESIRWTKDPRNT-QDHVRS 2164
:||||||.|..|....| ..|||..|.:...:.:.: |.| |:.:.|
POLG_FMDV1 2244 --DVTFLKRHFHMDYGTGF-YKPVMASKTLEAILSFAR--RGTIQEKLIS 2288
POLG_POL1M 2165 LCLLAWHNGEEEYNKFLAKIRSVPIGRALLLPEYSTLYRRWLDS 2208
:..||.|:|.:||.:... |......:|.|.:||.||:::
POLG_FMDV1 2289 VAGLAVHSGPDEYRRLFE-----PFQGLFEIPSYRSLYLRWVNA 2327
#---------------------------------------
#---------------------------------------
Сразу видно различие в проценте покрытия последовательностей.
| Poliovirus type 1 coverage | Foot-and-mouth disease virus coverage | |
| BLAST | 27,84% | 27,57% |
| water | 99,90% | 91,17% |
То есть локальное выравнивание в water по процентам покрытий может сойти и за глобальное. Но мы будем рассматривать только участок, который был также выровнен и в
BLAST.
Выравнивания в water и в BLAST имеют отличия. Например, индель в water находится в последовательности Poliovirus между аминокислотами Thr1883 - Arg1884. В BLAST же
этот индель находится в позициях Arg1884-Asp1885. Поэтому в water образовалась пара Arg1884 - Pro2003, а в BLAST - Arg1884 - Glu1999.