Работа с EMBOSS
Упражнения по EMBOSS
Необходимо было выбрать 7 упражнений из списка и создать текстовый документ с правильными командами. Файл можно скачать либо по этой ссылке, либо по адресу ~/term3/block2/
Скрипт по задаче
Я выбрал задачу №1 - оценить, сколько в среднем находок с E-value, меньшим 0.1, находит blastn для случайной последовательности в геноме бактерии.
Написанный
скрипт 100 раз ищет находку для случайной последовательности в выбранной базе данных, транслируя выдачу BLAST в таблицу формата .tsv. Затем при анализе таблицы считается
количество находок с удовлетворяющим условиям E-value. После этого суммарное количество находок за 100 повторений цикла делится на 100, и так получаем среднее.
Сначала скрипт запускался с фиксированными базами данных (геномом бактерии
Mycobacterium tuberculosis и длиной
случайной последовательности, равной 100). Такой скрипт можно скачать здесь.
Затем скрипт был усовершенствован, и теперь можно прописать в командной строке длину случайной последовательности, а также геном бактерии, в котором будет
производиться поиск предположительно гомологичных последовательностей. Такой скрипт можно скачать
здесь.
Маленькое полученное значение (меньше 1) говорит о том, что в большом количестве случаев выравниваний с маленьким E-value вообще не было.
P.S.
Почему-то русские буквы при открытии скрипта с сайта становятся непонятной мешаниной (проблема в кодировке, видимо), поэтому на всякий случай прикрепляю модификацию
скрипта с выдачей по-английски.
ссылка на английский скрипт
Поиск гомологов белков в неаннотированном геноме
Для поиска родственных организмов я воспользовался сервисом Lifemap: NCBI version. На нём можно видеть, что наиболее близкими к Amoeboaphelidium protococcarum являются организмы из семейств Кotosphaerida и Ichthyosporea.
Однако в этих таксонах нет ни одного организма с хорошо аннотированными белками (из раздела Reviewed на Uniprot). Большинство белков имеют статус Inferred from homology и Unreviewed,
что является недостаточно высоким статусом для поиска гомологов этих белков среди неаннотированного генома. Поэтому было принято тяжёлое решение - взять
хорошо аннотированные белки менее родственного организма (хотя бы из таксона Opistokonta).
Честно говоря, под такие мягкие критерии родства подошли бы и белки
Homo sapiens при их высокой консервативности, однако мы нашли немного более близкие организмы среди грибов и им подобных.
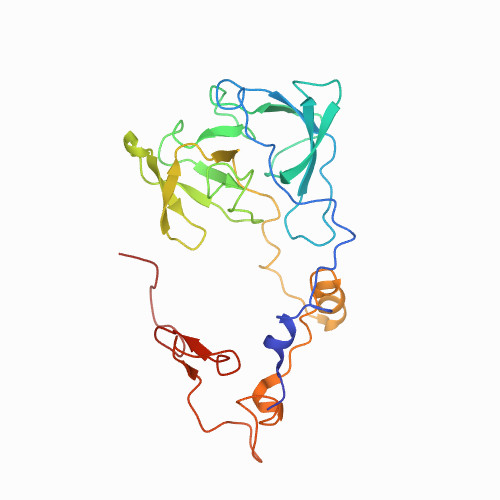
Первым белком стал белок субъединицы цитохрома с - достаточно консервативный белок, который есть у всех эукариот. Белок, гомологи которого мы искали в неаннотированном
геноме, принадлежит хитридиомицету Spizellomyces punctatus.
Белковая последовательность была получена с помощью команды
seqret 'uniprot:Q950S3' cytochrome.fasta.
Скачать последовательность можно
здесь.
Для поиска гомологов использовался tblastn. Команду можно видеть ниже:
tblastn -query 'cytochrome.fasta' -db 'X5.fasta' -out 'output_cyto.tsv'
Забыл сказать, что предварительно была создана локальная база данных:
makeblastdb -in X5.fasta -dbtype 'nucl'
Результаты выдачи BLAST можно посмотреть здесь. Можно говорить о
наличии гомологичного субъединице цитохрома b белка в протеоме у Amoeboaphelidium protococcarum из-за низкого значения E-value у лучшего выравнивания (6e-58), а также из-за большой длины
выравнивания (220 аминокислот при длине белка 360 аминокислот).
Вторым белком стала метиониновая аминопептидаза I - важный фермент, участвующий в процессинге белка (отщепляет метионин на N-конце). Последовательность была взята
у лабораторного организма Saccharomyces cerevisiae (strain ATCC 204508 / S288c).
Поисковый запрос в UniProt:
aminopeptidase 1 saccharomyces AND reviewed:yes
Команда загрузки белковой последовательности:
seqret 'uniprot:Q01662' map1_yeast.fasta[ссылка на последовательность]
Команда поиска гомологов белка:
tblastn -query map1_yeast.fasta -db 'X5.fasta' -out 'output_amino.txt' [ссылка на выдачу BLAST]
Судя по очень низкому значению E-value (1e-112), а также по большому проценту Simimlarity в выравнивании и его длине, примерно равной длине белка аминопептидазы у
Saccharomyces cerevisiae (длина выравнивания примерно 350 аминокислот, длина белка по данным UniProt - 360 аминокислот), можно сказать, что гомологичный
аминопептидазе I белок у Amoeboaphelidium protococcarum с большой вероятностью присутствует.
Третий белок - 60S ribosomal protein L42-A - был взят опять из Saccharomyces cerevisiae. Команды те же:
Поисковый запрос в UniProt:
60S ribosome AND saccharomyces AND reviewed:yes
Команда загрузки белковой последовательности:
seqret 'uniprot:P0CX27 ' ribosome.fasta[ссылка на последовательность]
Команда поиска гомологов белка:
tblastn -query ribosome.fasta -db 'X5.fasta' -out 'output_ribo.txt' [ссылка на выдачу BLAST]
Гомологичный рибосомальному белку L42-A белок с большой вероятностью присутствует у Amoeboaphelidium protococcarum, так как выравнивание произошло по всей длине белка Saccharomyces cerevisiae и E-value
достаточно маленькое (1е-48).