Программа getorf пакета EMBOSS
Из базы данных EMBL была получена запись с идентификатором D89965. В файле содержится последовательность мРНК гена серотонинового рецептора желудка (RSS) rattus norvegicus (серой крысы).
Программа getorf пакета EMBOSS находит открытые рамки считывания в последовательности. Открытой рамкой считывания называется последовательность между двумя соседними стоп-кодонами, либо старт-кодоном и стоп-кодоном. Для определения старт- и стоп-кодонов используются данные таблицы генетического кода.
В последовательности найденной мРНК серотонинового рецептора мыши был произведён поиск открытых рамок считывания между стоп- и страт-кодонами с размером не менее 30 аминокислотных остатков.
В командную строку была введена следующая команда:
getorf -find 1 -minsize 90 -sequence d89965.entret
В результате было найдено 5 белковых последовательностей, соответствующих различным рамкам считывания.
Белок, кодируемый последовательностью открытой рамки считывания с координатами 163-432, полностью совпадает с последовательностью белка, описанной в записи EMBL с идентификатором D89965. Координаты кодирующей последовательности в файле отличаются: 163-435. Видимо, такой вид записи включает координаты строп-кодона.
В записи D89965 можно найти ссылку на запись в базе данных Swiss-Prot с идентификатором P0A7B8. С помощью needle было сделано выравнвиание последовательности из данного файла и последовательностей, полученных с помощью getorf.
Выравнивание без гэпов было построено с пятой последовательностью из файла с результатами работы getorf (координаты (294-1) – обратная цепь).
Белковая последовательность из записи D89965 была выравнена с белком из записи с идентификатором EMBL aaa40618, соответствующей серотониновому рецептору серой крысы. Было найдено крайне мало совпадений. Также можно заменить значительные отличия в количестве аминокислотных остатков. Можно сделать предположение, что исследователи выделили фрагмент мРНК кишечной палочки и приняли его за мРНК гена серотонинового рецептора серой крысы.
Файлы-списки
Для качивания всех доступных в Swiss-Prot записей, содержащих информацию об алкогольдегидрогеназах, была использована следующая команда:
seqret sw:adh*_* adh.fastaПолучен следующий файл: adh.fasta.
С помощью команды:
infoseq adh.fasta -only -usa >> adh.txtбыл получен список универсальных адресов всех белковых последовательностей дегидрогеназ из файла adh.fasta.
C помощью команды:
rep -f list_orgs.txt adh.txt > adh_orgs.txtбыл получен сокращённый список, который содержит USA белков дегидрогеназ следующих организмов: GEOAT, DROMT, RHIME, ENTHI, RHOS4, ORYSJ, STAAS. Файл с USA можно скачать по следующей ссылке: adh_orgs.txt.
Для получения последовательностей дегидрогеназ перечисленных выше организмов в формате FASTA, была использована следующая команда:
seqret @adh_orgs.txt adh_orgs.fastaКонечный файл с последовательностями можно скачать по ссылке: adh_orgs.fasta.
Случайная модель для оценки достоверности выравнивания
Для оценки достоверности выравнивания были взяты последовательности алькогольдегидрогеназ организмов Oryza sativa (идентификатор Swiss-Prot ADH2_ORYSJ) и Entamoeba histolytica штамм 2.4.1 (идентификатор Swiss-Prot ADH2_ENTHI).
С помощью программы water пакета EMBOSS были сделаны парные локальные выравнивания последовательности белка O. Sativa с исходной последовательностью белка R. Sphaeroides и выравнвиание с каждой из 100 перемешанных последовательностей.
С помощью скрипта Python был получен список весов всех выравниваний. Была построена гистограмма распределения весов выравниваний (см рис.1). Вес выравнивания с не перемешанной последовательностью составляет 55.5. Среди выравниваний с перемешанными последовательностями встречаются те, что имеют больший вес. Поэтому нельзя сделать вывод о гомологии последовательностей гидрогеназ O. Sativa и E. histolytica.
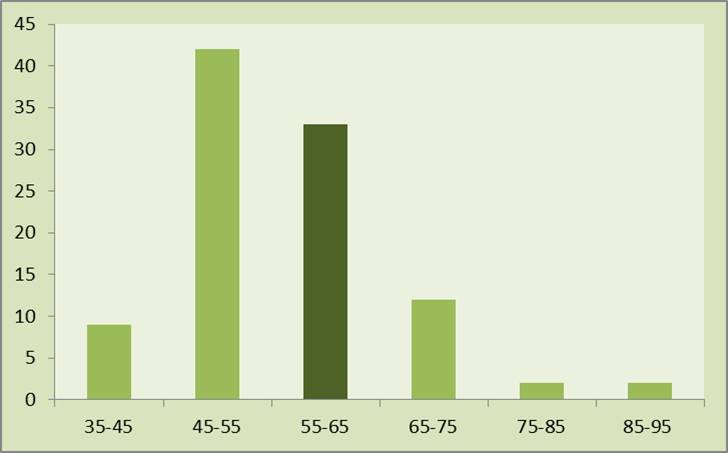
Для оценки достоверности выравнивания также были взяты нуклеотидные последовательности алькогольдегидрогеназ организмов Oryza sativa (идентификатор EMBL AK102799.1) и Entamoeba histolytica (идентификатор EMBL AAA81906.1).
С ними были проделаны те же операции, что и с белковыми последовательностями. Был получен список перемешанных последовательностей. Было получено локальное выравнивание water для нуклеотидной последовательности O. Sativa с изначальной последовательностью E. histolytica, а также выравнивание с перемешанными последовательностями E. histolytica.
С помощью того же скрипта был получен список весов выравниваний. Гистограмма, иллюстрирующая распределение весов выравниваний изображена на рис. 2.
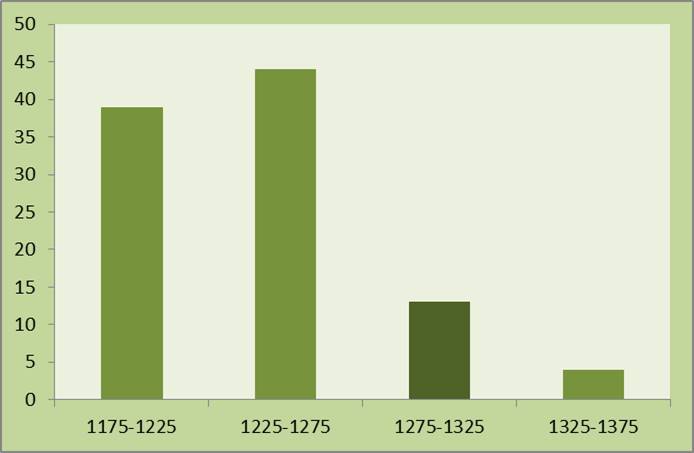
Как видно из рисунка 2, вес выравнивания не перемешанных последовательностей для нуклеотидных последовательностей в большей степени свидетельствует о возможной гомологии, чем вес белкового выравнивания. Полученный результат для данных последовательностей не согласуется с гипотезой о том, что белковое выравнивание должно сильнее отличаться от выравниваний, полученный перемешиванием, чем выравнивание нуклеотидных последовательностей.
Это может свидетельствовать о том, что общий нуклеотидный состав последовательностей похож (речь идёт как и сходстве в связи с функцией, так и в случае, если сходны только относительные содержания нуклеотидов),но произошло достаточное количество мутаций, которые изменили аминокислотный состав белков и сделали их непохожими друг на друга.
На ум приходит мысль, что относительные длины последовательностей сильнее влияют на нуклеотидное выравнивание, чем на белковое (чем больше разница между длинами последовательностей, тем больше гэпов; для белкового выравнвиания гэпов будет меньше, значит меньше сумма штрафов за гэпы).