Предсказание генов у прокариот
Дан контиг микробиома кардиального отдела желудка валлаби. Необходимо проаннотировать этот фрагмент.
Предсказания генов с помощью ORF Finder
C помощью ресурса ORF Finder в данном фрагменте были найдены все возможные рамки считывания. Далее рассматривались последовательности открытых рамок считывания (ОРС) с длиной транскрипта больше 60 а.о. Для поиска был использован бактериальный генетический код. Список всех предсказанных последовательностей ОРС представлен на рис. 1.
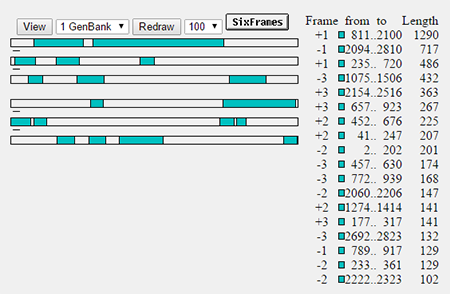
Далее для транскриптов последовательностей ОРС был произведён поиск гомологов в SwissProt с помощью Blastp. Рассматривались находки со степенью перекрывания порядка 80% (этот параметр мог варьировать в зависимости от того, какую часть гена кодирует предсказанная ОРС) и значением evalue меньше 0.001. Список последовательностей открытых рамок считывания, которые прошли фильтр, представлен в таблице 1. Комментарии к таблице приведены ниже.
| Началао | Конец | Длина в а.о. | Цепь | Описание | Идентификатор гомолога | Организм | Вес выравнвиания | Покрытие (%) | E-value | Идентичность (%) |
| 811 | 2100 | 430 (максимум) | + | Неаннотированный белок R802 (предположительно имдозолонпропионаза) | P40896.1 | Saccharomyces cerevisiae S288c | 84.7 | 68 | 5,00*10-17 | 30 |
| 2094 | за пределами контига | >238 | - | Тирозинрекомбиназа | P39776.1 | Bacillus subtilis subsp. subtilis str. 168 | 97.8 | 88 | 1,00*10-22 | 30 |
| 250 | 720 | 156 | + | Неаннотированный белок R802 (предположительно пурин нуклеозид фосфорилаза) | Q5UR60.1 | Acanthamoeba polyphaga mimivirus | 48.9 | 86 | 2,00*-06 | 30 |
| 41 | 247 | 68 | + | гипотетический белок | - | - | - | - | - | - |
В отношении первой ОРС предсказание с помощью blast неоднозначно. С одной стороны наибольший вес и наименьшее значение evalue имеет выравнивание с находкой соответствующей неаннотированному белку. В этом случае последовательности выравниваются с 114 а.о. транскрипта ОРС и с 23 а.о. находки. В области позиции (811+114*3=1153) не найдено старт-кодонов. Если предположить, что начало трансляции смещено на 23 а.о. относительно позиции 114, то в окрестности ((114-23+1)*3+811=1087), а именно в позиции 1078-1080 находится старт-кодон. В большинстве других случаев выравнивание транскрипа ОРС с находками (соотвутсвуют имидозолонпропионазам) начинается примерно с 23-26 а.о., что соответствует кодону с координатами 886-888 . Если же опять предположить, что начальные участи белковых последовательностей просто плохо выравниваются, то в этом случае позиция старт-кодона будет такая же, как и было получено в предсказании с помощью ORF. Поэтому в таблицу была внесена информация о наилучшем выравнивании, но позиция начала гена остаётся под вопросом.
Начало гена для второй предсказанной ОРС вероятно находится за пределами последовательности ОРС, так как большинство достоверных находок выравниваются с транскриптом как минимум после 66-ого аминокислотного остатка. Последовательность ОРС находится на конце контига, поэтому такой вариант возможен. Но, если взять во внимание то, что последовательность ОРС принадлежит обратной цепи, то координате начала в таблице будет соответствовать координата конца кодирующей последовательности.
Аналогичная ситуация наблюдается для третьей предсказанной ОРС (достоверные находки выравнвиаются начиная с некоторой удалённой от начала позиции). Однако, последовательность ОРС не расположена на конце котига. С другой стороны рядом расположено несколько перекрывающихся с ней последовательностей других ОРС. Это делает возможным нахождение начала трансляции за пределами предсказанной области.
Как было сказано выше, для первой рамки считывания не было предсказано старт-кодона в нужном месте. Четвёртая рамка прекрывается с третьей 12 нуклеотидами. Четвёртая рамка не перекрывается с другими рамками, содержазими гомологи. В связи с этим, начало третьей рамки было смещено.
Для остальных последовательностей ОРС нет находок, отвечающих заданным требованиям.
Предсказания генов с помощью GeneMark
Также для предсказания генов был использован ресурс GeneMark. Сначала для поиска генов были выбраны эвристические параметры 1999 года. Файл с результатом можно скачать по следующей ссылке. График кодирующего потенциала можно найти по следующей сслыке. Координаты предсказанных генов можно найти в таблице 2.
| Левый конец | Правый конец | Длина в а.о. | Цепь |
| <2 | 247 | 81 | + |
| 235 | 720 | 161 | + |
| 811 | 2100 | 429 | + |
| 2094 | 2810 | 717 | - |
Сравнение GeneMark и ORF Finder c blastp
С помощью GeneMark был предсказан дополнительный ген в пределах открытой рамки считывания с координатами (<2…247). При этом минимальное значение evalue при поиске гомологов с помощью blastp было равно 0.9 и поэтому находка не рассматривалась. Также в предсказании с помощью ORF Finder соответствующая ОРС имеет координаты 41…247.
GeneMark в отличие от blast не даёт информации о других возможных позициях старт-кодонов. Это делает метод менее чувствительны, так как возможны трудности при точном определении границ генов. С другой стороны, первичная обработка последовательности контига с помощью GeneMark могла бы помочь определить наиболее вероятные участки нахождения генов (их получается в несколько раз меньше, чем просто при предсказании ОРС с помощью ORF Finder) и в результате сделать поиск более продуктивным.
Запуск GeneMark с альтернативными параметрами
Запуск GeneMark с эвристическими параметрами 2010 года не дал результатов, отличных от тех, что были получены выше.