Построение дерева по нуклеотидным последовательностям
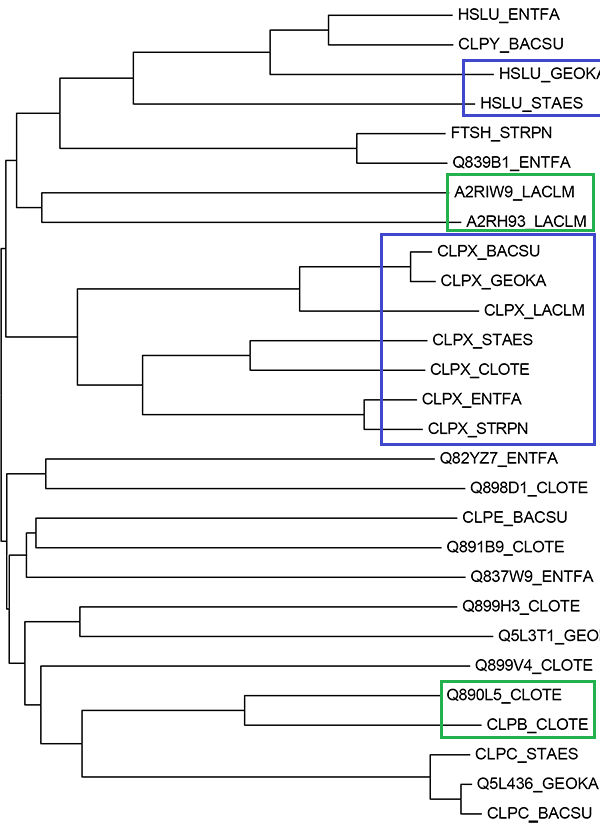
Было построено филогенетическое дерево бактерий отдела Firmicutes по нуклеотидным последовательностям 16S рРНК. Последовательности рРНК были получены из файла с расширением .frn из базы полных геномов NCBI. Последовательности выровнили с помощью алгоритма Muscle и построили дерево методом Neighbor-Joining. Дерево представлено на рис. 1.
Как видно на рис.1, полученное дерево содержит две нетривиальные ветви, которых нет в дереве из первого практикума:
{CLOTE, GEOKA} против {ENTFA, STAES, BASCU, LACLM, STRPN}
{STAES, BASCU} против {ENTFA, CLOTE, GEOKA, LACLM, STRPN}
Качество реконструкции дерева по выравниванию нуклеотидных последовательностей хуже, чем по выравниванию белковых. Так реконструкция дерева по белкам методом максимальной экономии давала дерево, совпадающее с таксономическим (задание защищено на коллоквиуме).
Построение и анализ дерева, содержащего паралоги
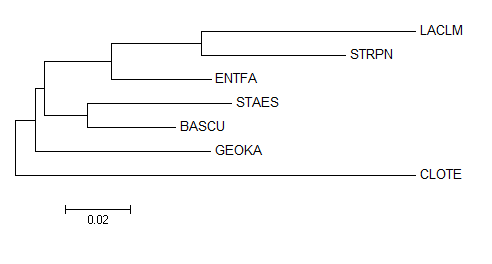
Было построено дерево гомологов белка CLPX_BACSU среди белков изучаемых представителей отдела Firmicutes.
Протеомы всех исследуемых бактерий объединили в один файл, а затем получили гомологов белка CLPX_BACSU с помощью программы blastp. Из выдачи программы извлекли идентификаторы находок и скачали соответсвуеющие им белковые последовательности. Полученные таким образом последовательности выровнили с помощью сервиса Muscle. Дерево, построенное на основании выравнивания методом Neighbor-Joining представлено на рис. 2.