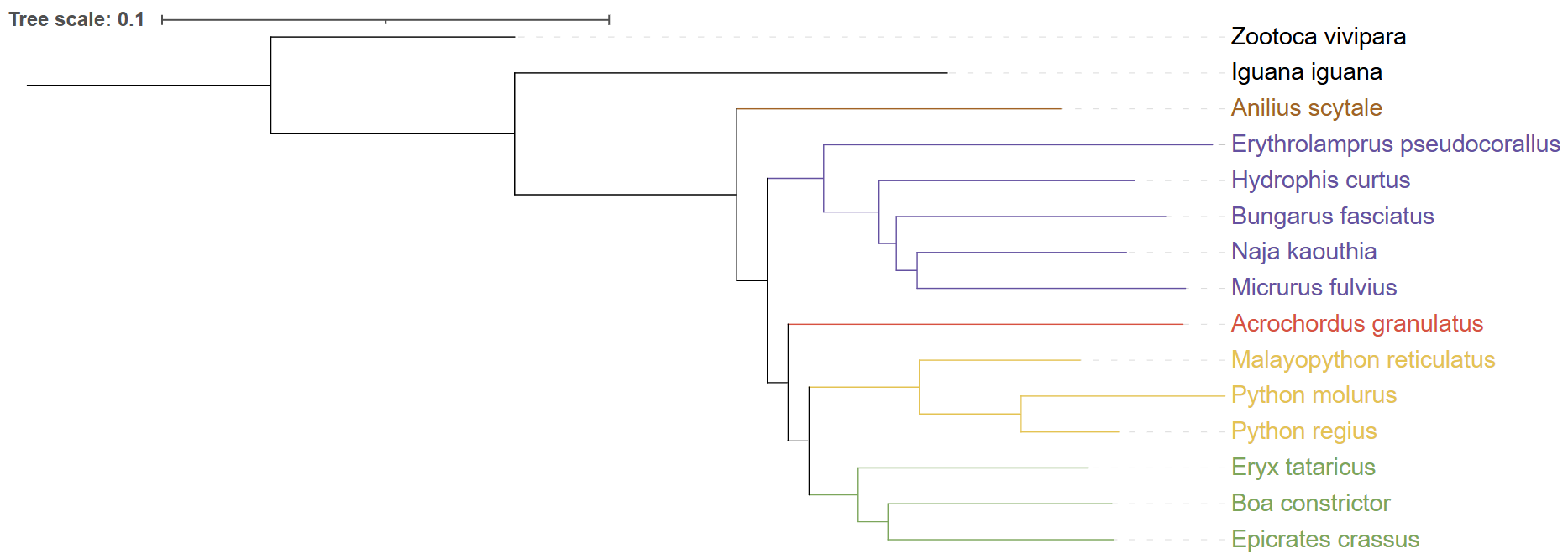
Строил дерево по последовательности 12s rRNA. К сожалению, многие взятые в прошлый раз представители не имели её полностью расшифрованной. Поэтому их пришлось заменить на родственных, за исключением Loxocemus bicolor, которого пришлось поменять на Anilius scytale. Укоренение деревьева производил как и в прошлый раз при помощи аутогруппы - Zootoca vivipara. Из Рис.3 понятно, что NCBI Taxonomy немногое может сказать об отношениях между группами. Если сравнивать дерево FastME по 12s rRNA (Рис.1) и iqTree по цитохромам B(Рис. 2), то дерево iqTree выглядит правдоподобнее так как мы знаем, что ужеобразные отделились не раньше, чем боаобразные и питонообразные. Оба дерева (Рис.1 и Рис.2) согласны с положением Anilius scytale, однако на рис.1 Acrochordus granulatus сближен с питонами и боа, а на рис.2 с ужеобразными.

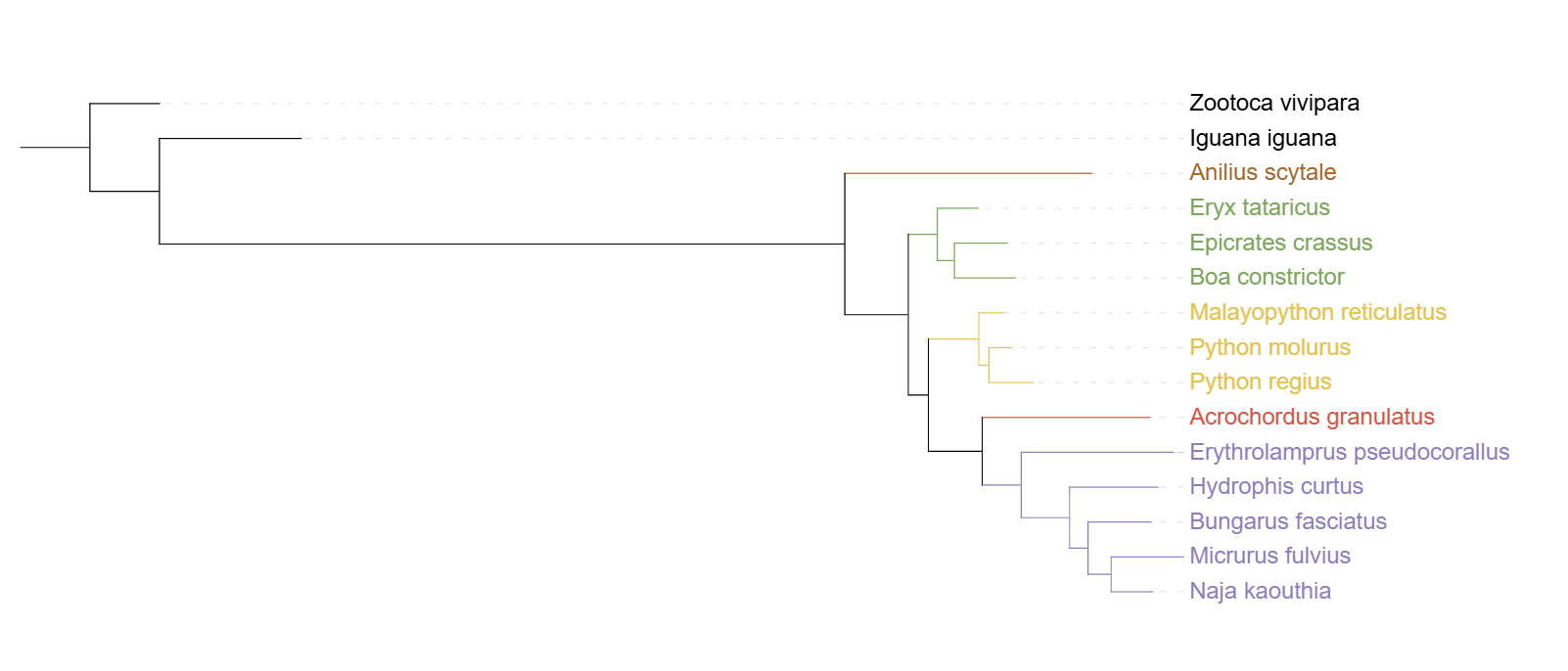
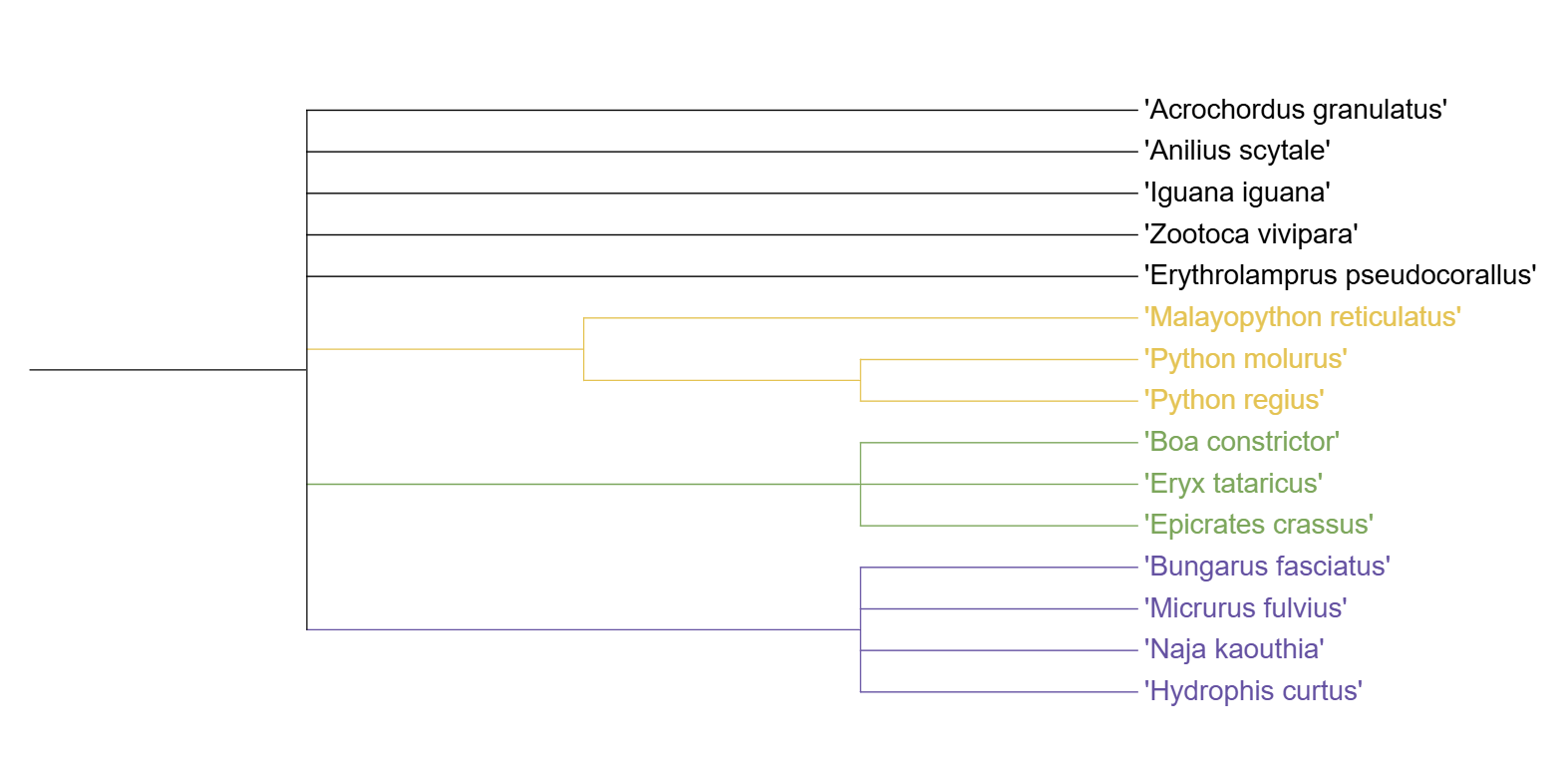
Далее требовалось найти аутогруппу. Она у меня уже была, но задание требовало, поэтому решено было взять Dibamus bourreti, входящий в Diabamidae - таксон, в кооторый не входит ни один представитель из моего дерева и имеющий равный ранг с Bifurcata, к которому принадлежат все другие виды.
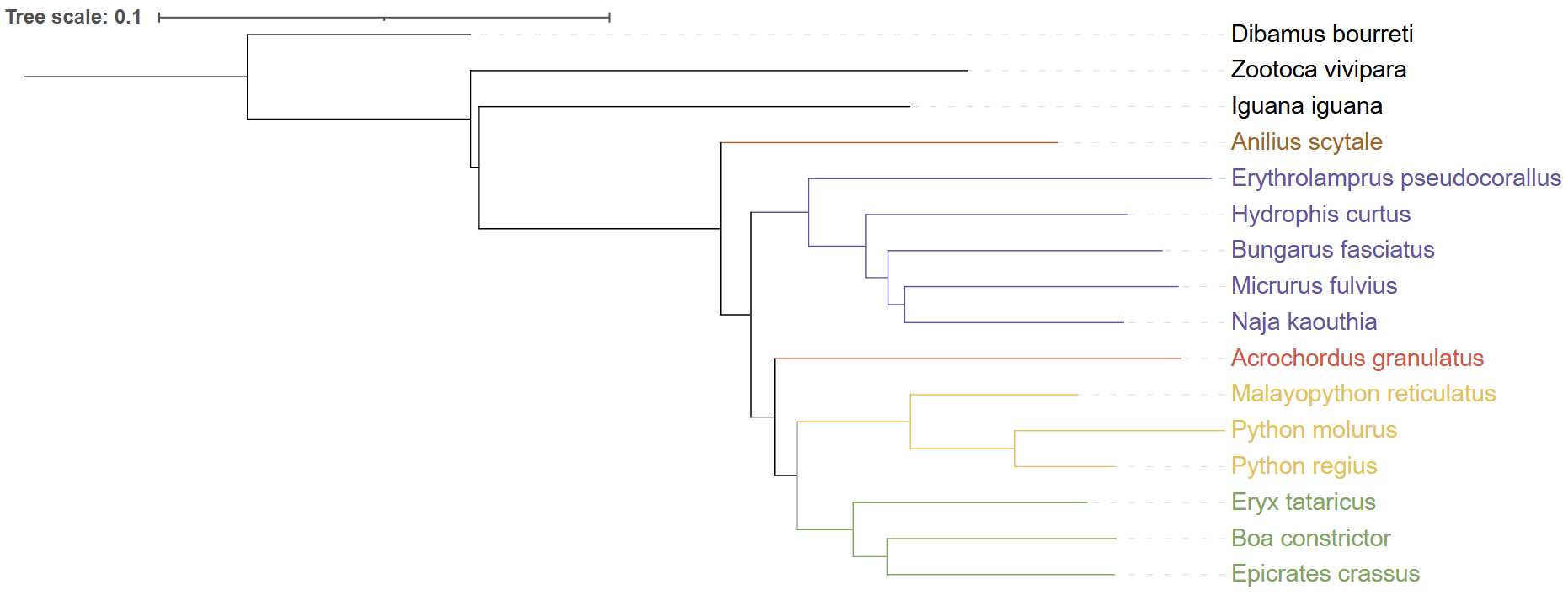
После добавления новой аутогруппы ничего не поменялось, а значит прошлые укоренения вполне справедливы.
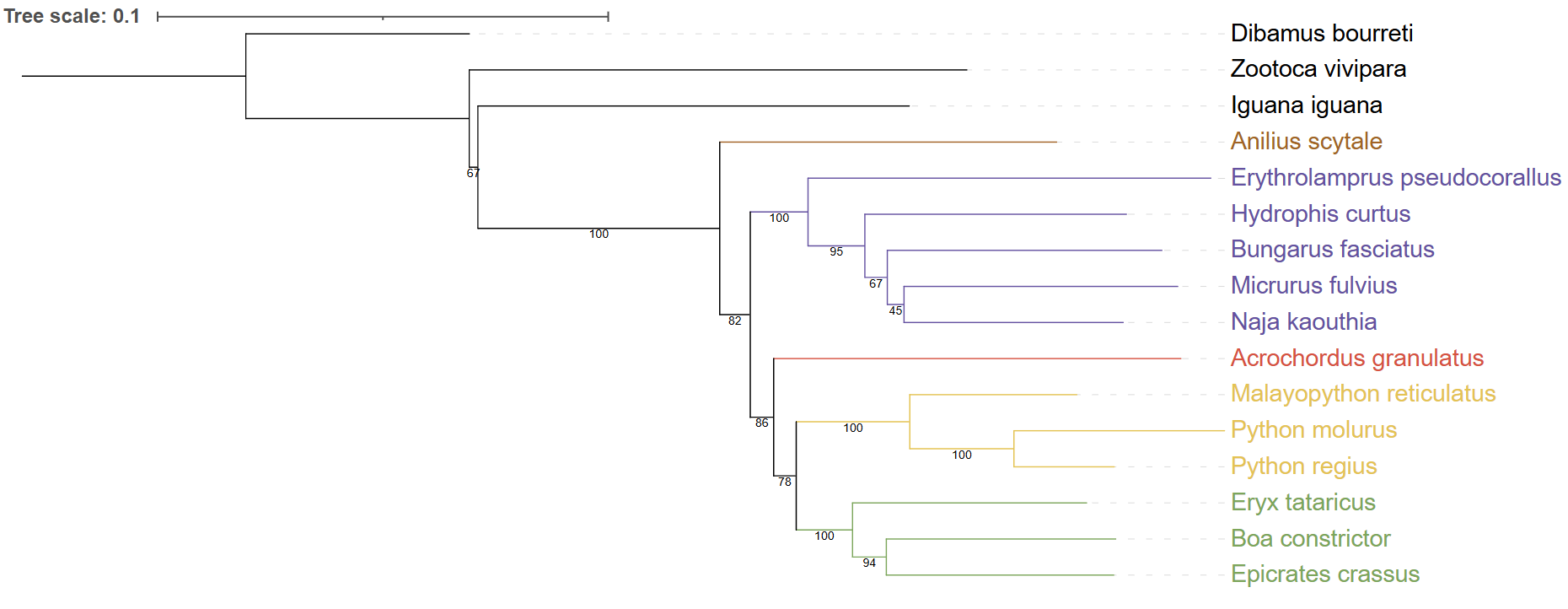
Мы видим, что на ветвях, насчёт которых не все программы прошлого практикума сошлись, действительно стоит низкое значение достоверности. А именно: ветвь, отделяющая Bungarus fasciatus, Micrurus fulvius и Naja kaouthia, и ветвь, отделяющая Micrurus fulvius и Naja kaouthia