
Для этого были взяты геном и аннотация генома из базы данных RefSeq(скачаны с NCBI). Из аннотации извлечены координаты генов, затем получен список межгенных расстояний >=200 пар нуклеотидов для того чтобы хоть как то попытаться избежать генов в опероне. После этого были получены межгенные последовательности, из которых получили промоторных области [-50:-1]. И для контроля взяты области [-150:-100]. Были взяты 100 случайных последовательностей промоторов Ссыла на Google Colab
В этой работе мы будем искать сигнальне последовательности в промоторах хромосомных генов Shigella flexneeri 2a str.301 (плазмидные гены рассматривать не будем). Для E.coli последовательность Shine-Dalgarno выглядит так: AGGAGGU и заканчивается примерно на -7. Запустим MEME с соответствующими параметрами для выборки из 100 последовательностей и для контроля.
meme proms_100.fasta -dna -nmotifs 1 -minw 5 -maxw 8 Выдача
meme control.fasta -dna -nmotifs 1 -minw 5 -maxw 8 Выдача
По итогу никаких стоящих мотивов найдено не было. Хоть p-value на находках и <0.05, но находок всего 5 и e-value=8.4 говорит, что сам "Мотив"(Рис. 1) - просто статистическая случайность. В контроле, ожидаемо, тоже ничего не нашлось evalue=0.61(Рис. 2)


Но все таки хочется найти хоть что-нибудь. Поэтому запустим программу MEME с теми же параметрами, но применим ко всем подхдящим промоторам, а не только к случайным 100 выдача. В итоге получили многообещающий мотив, который нашёлся в 104 последовательностях из 987 с e-value=4.1e-028. Он и внешне напоминает Shine-Dlgarno, и находится почти во всех совпадениях приблизительно от -10 до -7. Однако, прежде чем что-либо утверждать следует провести тестирование при помощи FIMO.
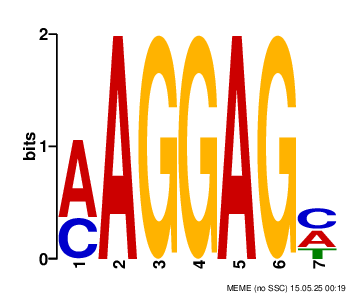
Запустим программу чтобы проверить найденный сигнал:
fimo --oc fimo_proms_multi meme_out_multi/meme.txt proms.fasta Выдача
К сожалению, как видно по q-value=0.104, найденный сигнал не прошёл поправку на множественное тестирование. В этой работе нам не удалось найти сигналов в промоторах генов Shigella flexneri 2a str.301.