|
|
|
|
|
|
|
|
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
|
|
|
|
|
|
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Программа BLASTP
Удалось найти мой белок и в SwissProt, и в nr, найдены а структура белка и в pdb, но в ней отсутствует предпоследня аминокислота Различия в количестве возможных гомологов белка в базах данных и в E-value "худшей находки" объясняются сильным отличием в объёме этих банков данных. В банке "nr" больше всего записей, поэтому больше потенциальных гомологов и самое большое E-value для "худшей находки", а в PDB банке меньше всего записей, значит возможных гомологов меньше. С помощью BLASTP был найден наилучший гомолог FTSH_ECOLI в далеком от E.coli организме. Для поиска были взяты Homo sapiens, Archaea, Actinobacteria, Alteromonadales, Vibrionaceae (приведены в порядке приближения к E.coli). Первый подходящий белок (E-value < 0.001) был найден у Homo sapiens. найболее далекого гомолога по таксонам.
С помощью BLASTP был найден белок, содержащий последовательность, похожую на фрагмент белка FTSH_ECOLI . Затем, была получена последовательность лучшей находки в fasta формате и проведен поиск по AC этого белка.
Лучшей находкой является FTSH_ECO57. Отличие между FTSH_ECOLI и FTSH_RICBR показано на выравнивании Вырнивание, построенное Blastp:
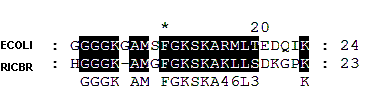 Выделенный фрагмент (отмечен красной рамкой) не совсем совпадает с пробным выравниванием этих последовательностей, проведенных вручную (картинка, расположенная ниже выравния plastp), в выравнивании blastp поседовательности RICBR сдвинута на 1 а.о. влево, засчет этого нарушилось выравнивание. С помощью программ Needle и Water были получены оптимальное глобальное и оптимальное локальное выравнивания, размеры штрафов за гэп были взяты равными стандартным для программы BLASTP [за создание - 11.0, за удлинение - 1.0] Оптимальное локальное выравнивание, построенное с помощью water (Скачать msf) Длина: 600
Оптимальное глобальное выравнивание, построенное с помощью needle (Скачать msf) Длина: 655 Выше на страничке изображено выравнивание этих же белков, построенное с помощью BLASTP В результате три выравнивания (оптимальное глобальное, оптимальное локальное и blastp) разичаются только длинами последовательностей, а счет локального и глобального очень , так же как и идентичность.
|
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
© Замараев Алексей |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
