- Краткое описание структуры в файле 1QU2.pdb
В файле приведены координаты атомов следующих молекул из Staphylococcus aurensis: изолейцил-тРНК и изолейцил-тРНК синтетазы, всего 2 молекулы.
Для исследования была выбрана цепь T, представляющая изолейцил-тРНК со следующей последовательностью:
[1] 5' G G G C U U G U A G C U C A G G U G G U U A G A G C G C A C C C C U G A U A A G G G U G A G G U C G G U G G U U C A A G U C C A C U C A G G C C C A C 3' [74]
1 и 74 - номера первого и последнего нуклеотидов.
Hа 3'-конце последовательности находится триплет CAC, приведены координаты его атомов.
- Исследование вторичной структуры
С помощью программы find_pair пакета 3DNA были определены возможные водородные связи между азотистыми основаниями.
В соответствии с полученными данными
акцепторный стебель состоит из участка 1-7 и комплементарного ему участка 66-72
Т-стебель: 49-53 и 61-65
D-стебель: 10-13 и 22-25
антикодоновый стебель: 38-44 и 26-32
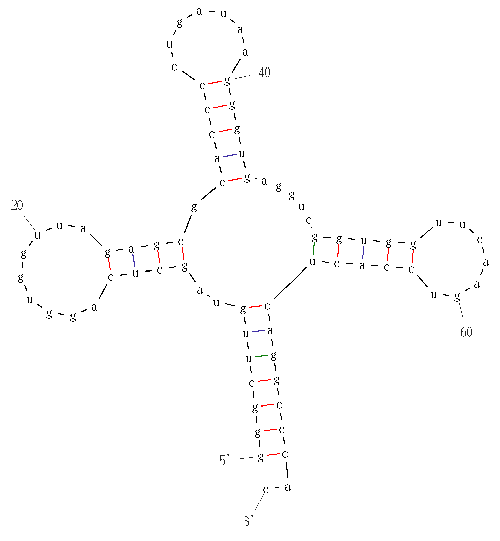
Вторичная структура изолейцил-тРНК из Staphylococcus aurensis.
Акцепторный стебель выделен красным, Т-стебель - зеленым, D-стебель - синим, антикодоновый - оранжевым.
Антикодон в антикодоновой петле составляют остатки 34-36 (GAU), что соответствует AUC кодону мРНК, который кодирует изолейцин. На рисунке нуклеотиды антикодона показаны в шарнирной модели.
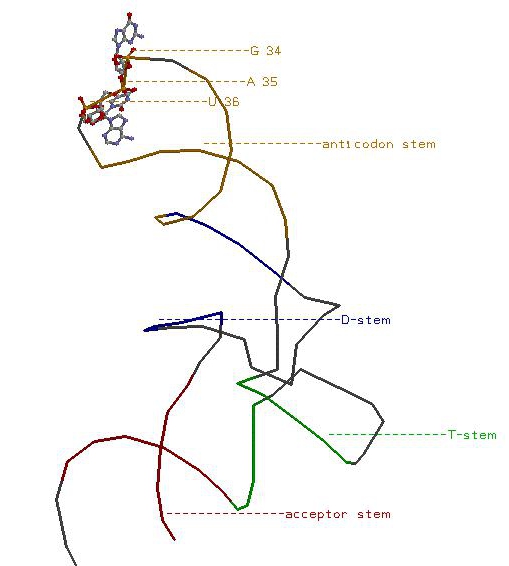
Скрипт для получения изображения:
restrict none refresh background white center rna select all label off color grey backbone 50 select 1-7, 66-72 color red select 3 and *.P label ---------------acceptor stem select 49-53, 61-65 color green select 52 and *.P label ---------------T-stem select 38-44, 26-32 color orange select 41 and *.P label ---------------anticodon stem select 10-13, 22-25 color blue select 12 and *.P label -----------------------D-stem select 34-36 color cpk spacefill 100 wireframe 30 select 34 and *.P label ---------------G 34 select 35 and *.P label ---------------A 35 select 36 and *.P label ---------------U 36 refresh
Структуру тРНК содержит 30 пар оснований, из низ 10 - не Уотсон-Криковские.
Структуру стеблевых дуплексов поддерживают 19 канонических и 4 неканонических пар оснований.
Неканоническая пара G26 - A44
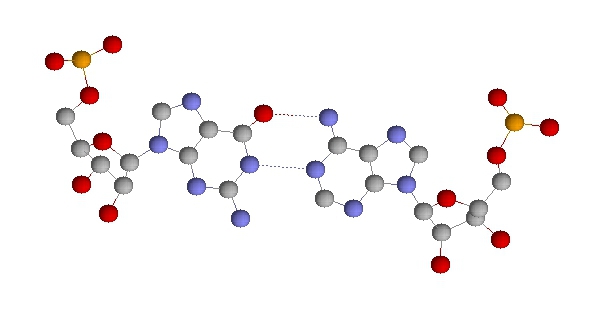
Дополнительные сведения:
Вариабельная петля присутствует (45-48), остаток тимидина в Т-петле и остаток дигидроуридина в D-петле отсутствуют.
- Исследование третичной структуры
Cтекинг-взаимодействия между основаниями конца акцепторно стебля и начала Т-стебля.
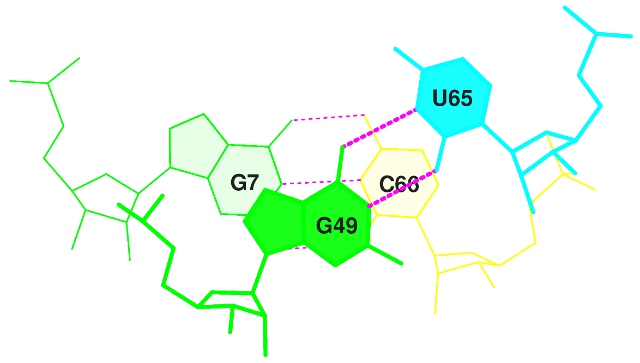
Суммарная площадь перекрвания = 2,87 Å2, однако, площадь перекрывания атомов гетероциклов = 0,32 Å2
Cтекинг-взаимодействия между антикодоновым и D-стеблем.
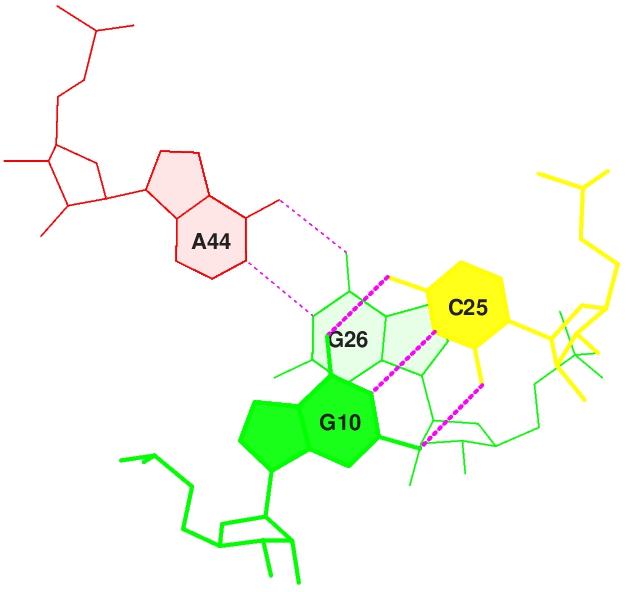
Суммарная площадь перекрвания = 1,56 Å2, однако, площадь перекрывания атомов гетероциклов = 0,11 Å2
Дополнительные водородные связи между основаниями D- и Т-петель.
T-петля: 49-65
D-петля: 10-25
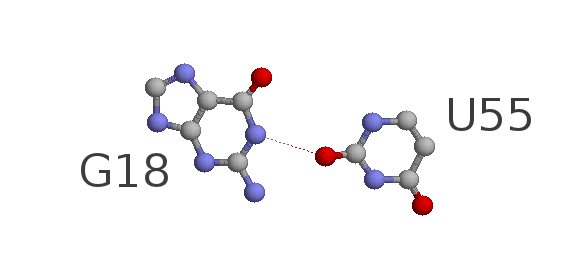
U55 - G18 - неканоническая пара, G19 - C56 - Уотсон-Криковская пара.
- Предсказание вторичной структуры тРНК
Участок структуры |
Позиции в структуре (по результатам find_pair) |
Результаты предсказания |
Результаты предсказания по алгоритму Зукера mfold |
|
Акцепторный стебель |
5' 1-7 3' 5' 66-72 3' Всего 7 пар |
предсказано 7 пар из 7 реальных | предсказано 7 пар из 7 реальных | |
D-стебель |
5' 10-13 3' 5' 22-25 3' Всего 4 пары |
предсказано 0 пар | предсказано 4 пар из 4 реальных |
|
T-стебель |
5' 49-53 3' 5' 61-65 3' Всего 5 пар |
предсказано 0 пар |
предсказано 5 пар из 5 реальных | |
Антикодоновый стебель |
5' 38-44 3' 5' 26-32 3' Всего 7 пар |
предсказано 7 пар из 7 реальных | предсказано 5 пар из 7 реальных | |
Общее число канонических пар нуклеотидов |
19 |
11, предсказаны все канонические пары антикодонового и акцепторных стеблей |
19, предсказаны все канонические пары |
Программа einverted нашла все пары акцепторного стебля только при положительном значении mismatch (gap - 0, min threshold - 0, match - 5, mismatch - 1), все пары антикодонового - при увеличении штрафа за гэп (gap - 12, min threshold - 0, match - 5, mismatch - 1). При подборе параметров я сначала опустила порог, потом пробовала разные варианты со снижением штрафа за гэп и несовпадение оснований(так как в структуре были неканонические пары).
Программа mfold при увеличении параметра P(отличие структуры по энергии от оптимальной) до 15% выдала 7 структур, 2ая из которых представлена ниже. Эта структура предсказывет все пары, кроме двух неканонических пар антикодонового стебля.
Программа mfold лучше приспособлена для данной задачи и не требует пляски с бубном при подборе параметров.