- Филогенетическое дерево
Легенда:
Таксоны:
A - Archaea
B - Bacteria
E - Eucariota
Архитектуры:
1 -

2 -

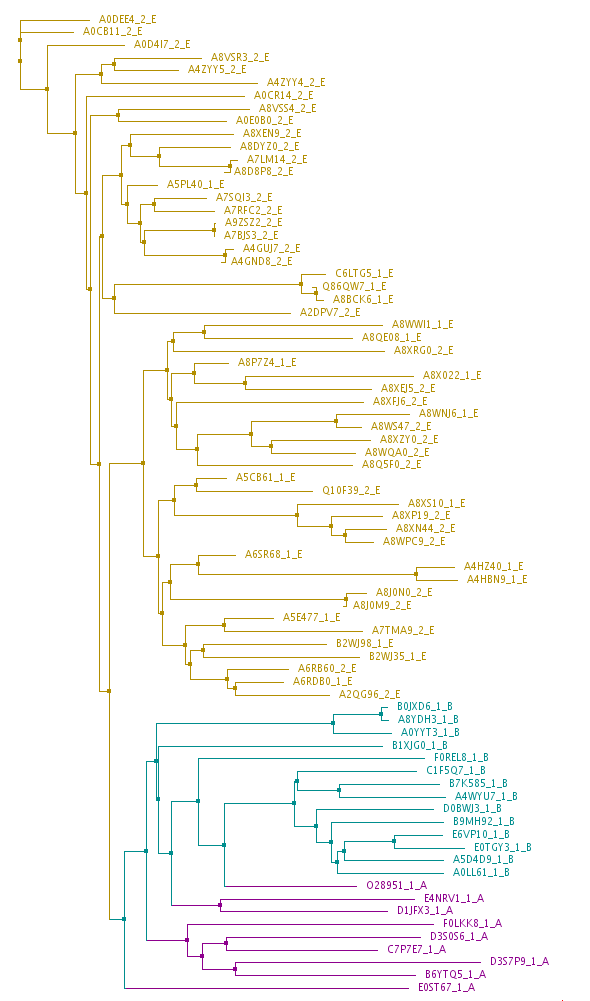
Дерево построено по алгоритму Neibor-Joining, не укоренено и без учета молекулярных часов, так как у бактерий и архей за единицу времени сменяется больше поколений, чем у эукариот.
Выводы:
По имеющимся данным можно предположить, что архитектура 1 является исходной для всех 3х таксонов, а архитектура 2 появляется у эукариот.
Археи и бактерии на дереве слегка перемешаны, что может быть связано с горизонтальным переносом генов или неточностью реконструкции
ссылка на файл со скобочной формулой
- Выбор групп
Учитывая случайное распределение архитектур по эукариотической части дерева и не очень надежную реконструкцию прокариотической между собой, мною были выбраны 2 группы последовательностей: прокариотические (1_B и 1_A) и эукариотические (1_E и 2_E).
- Построение профилей
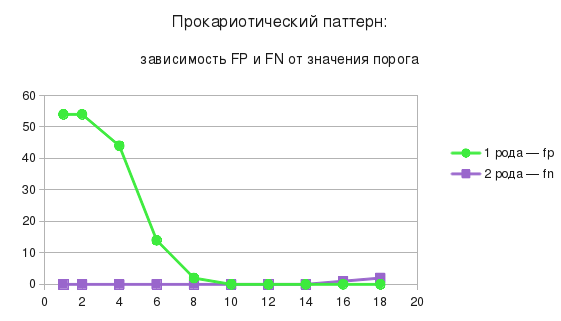
Рекомендуемый порог - 12
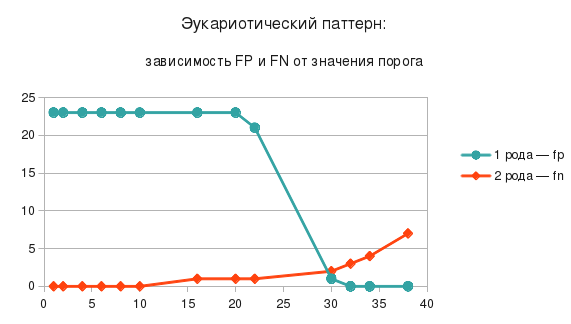
Рекомендуемый порог - 29
- Поиск по Swiss-Prot
Бактериальный профиль
2 находки в Swiss-prot. Архитектура и таксон верные. С высокой вероятностью, это могут быть все белки, так как домен Piwi найден пока только в примерно 40 прокариотических белках, по большей части из трембла. Профиль скорее удачный, чем нет.
Эукариотический профиль
8401 находка при определенном пороге. Среди 20 случайных последовательностей попались как про- так и эукариотические без домена Piwi. Профиль не удался.