Дополнительная информация к практикуму
1. Общее описание скрипта evolve_protein.pl
Этот скрипт написан на языке perl и для запуска требует установки компилятора; на компьютерах в компьютерном классе он уже установлен (как под Windows, так и под Linux, на сервере kodomo). Если Вы хотите запускать его на домашнем компьютере, то можете загрузить и установить компилятор с официального сайта.
Запустить этот скрипт, как и любой другой скрипт perl, можно используя командную строку Linux или же Far: выбирайте любой удобный вариант. Нужно скопировать скрипт в рабочую директорию. Для получения списка параметров скрипта запустите скрипт без параметров: он выдаст весь список, как и те программы, с которыми вы уже работали ранее под Linux.
Обратите внимание пожалуйста, что данный скрипт не установлен на сервере kodomo и в компьютерном классе, и это означает, что его нельзя запускать из любой директории через название, как, например, программы EMBOSS. Перед запуском удостоверьтесь, что вы находитесь в той самой директории, где лежит файл со скриптом - или указывайте полный путь к нему, а не только название.
Собственно, обязательных параметров у скрипта только два: -i для указания файла с последовательностью вашего белка в формате FASTA (только ОДНОЙ последовательности) и -o для указания имени файла с результатом. То есть пример запуска скрипта может быть таким: evolve_protein.pl -i mySeq.fasta -o mySeq_evolution_result.txt
Скрипт старается предусмотреть все возможные ошибки в формате исходного файла и вводимых данных, но не гарантирует этого, поэтому если вы получаете странные результаты - имеет смысл проверить правильность использования входных параметров и написать мне (udavdasha@fbb.msu.ru).
Есть 2 пути изменения параметров скрипта: их можно подавать через командную строку или менять соответствующие им значения по умолчанию в теле скрипта (его можно редактировать как обычный текстовый файл).
2. Простая эволюционная модель, реализованная в скрипте evolve_protein.pl
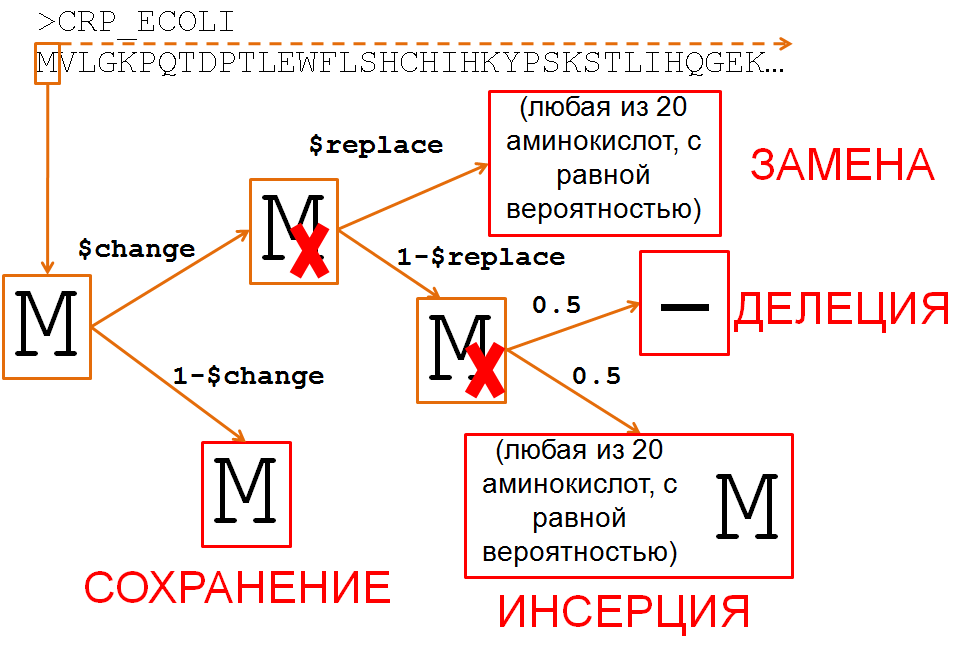
Скрипт моделирует эволюцию последовательности, которую вы задаете ему через опцию -i, без учета естественного отбора: то есть обеспечивает только наследственность и изменчивость. Модель изменения выглядит так (см. Рис. 1): для каждой буквы последовательности есть вероятность, что в данной позиции случится какая-то ошибка. Эта вероятность задается параметром по умолчанию $def_change или опцией -с.
Вероятность тут следует понимать в самом простом смысле: например, как действительное число от 0 до 1, которое при умножении на 100 показывает, в скольких случаях из 100 в среднем произойдет некоторое событие. Например, если вероятность какого-то события равна 0.1, то в 10 случаях из 100, как правило, это событие случится. Все эти "в среднем" и "как правило" говорят о том, что вероятность 0.1 не гарантирует обязательную реализацию события именно каждый 10-й раз, и не гарантирует, что ровно в 10 случаях из 100 событие произойдет, но описывает тенденцию.
То есть если, например, $def_change выставить равным 0.3, то это будет означать, что в среднем в 30% букв будет происходить изменение, а в 70% все останется как было. Изменения могут произойти двух типов: либо аминокислота будет случайно заменена на другую (при этом замена на саму себя возможна с вероятностью 1/20, то есть такой же, как и для всех других вариантов), либо будет удалена из новой последовательности, либо будет сохранена, но перед ней случится вставка еще одной случайной аминокислоты. Вероятность замены описывается параметром по умолчанию $def_replace или задается опцией -r. Если замена не происходит, то далее с вероятностью 0.5 случится делеция, и с вероятностью 0.5 - инсерция (т.е. вставка, но по-ученому; ААл) .
|
Рис. 1. Схема вероятностей разных событий, которые могут произойти с каждым остатком белка, в реализации скрипта evolve_protein.pl. |
Для обязательных заданий вам надо будет изменять только эти опции. В стандартном режиме скрипт после мутирования вырежет из получившейся последовательности случайный кусок длиной 20 аминокислотных остатков, с которым вы и будете работать.
Другие опции:
-f: заставляет скрипт создавать полноразмерные последовательности;
-t: задает количество последовательностей, которые нужно создать;
-g: задает количество "поколений", то есть проходов по исходной последовательности.


 2023
2023 2022
2022 2021
2021 2020
2020 2019
2019 2018
2018
 2017
2017 2016
2016