Блок 3
Задание 1. Сайты рестрикции-модификации в геноме Bacteroides helcogenes.
Был взят геном бактерии Bacteroides helcogenes (CP002352.1.fasta) и набор контигов той же бактерии из метагенома кишечника человека (Bacteroides_helcogenes.fasta), а также список известных сайтов рестрикции-модификации sites.list. С помощью специального сервиса в геноме был проведен поиск сайтов Р-М по методу Карлина. Полученные данные представлены в файле RM_sites.xlsx (геном бактерии – на листе genome, сайты в контигах из метагенома – на листе contigs). В геноме нашлось 27 сайтов Р-М, имеющих контраст меньше 0.78 (выделены фиолетовым), а в контигах из метагенома кишечника всего 6 (выделены салатовым). Скорее всего, эти сайты избегаются при работе системы рестрикции-модификации. Сайты, выделенные в сводной таблице на листе sites синим, избегаются в обоих геномах. В целом заметно, что большое количество сайтов рестрикции в контигах из метагенома не избегается, в отличие от полного генома Bacteroides helcogenes. Это может означать, что штамм из кишечника потерял многие рестриктазы, которые узнавали эти сайты. Кроме того, в обоих штаммах есть сайты GCTAGC и CCTAGG, имеющие контраст выше 1, то есть избыточно представленные в геноме.
Задание 2. Поиск последовательности Шайна-Дальгарно в геноме археи Vulcanisaeta moutnovskia 768-28
Последовательность Шайна-Дальгарно (ШД) – это последовательность нуклеотидов мРНК у прокариот (бактерий и архей), расположенная в upstream-области от старт-кодона и необходимая для связывания мРНК с малой субъединицей рибосомы за счает комплементарности 3'-концу 16S рРНК. Последовательность была впервые описана Дж. Шайном и и Л. Дальгарно в 1974 году. Консенсус последовательности - AGGAGG.
Со страницы археи Vulcanisaeta moutnovskia 768-28 был скачан полный геном хромосомы CP002529 (genome.fasta), а также таблица cо свойствами, которая была обработана в скрипте features2CDs.py. Результат работы скрипта был обработан в Excel: удалены плохо аннотированные гены, а также гены короче 300 пн и длиннее 1200 пн. В результате была получена итоговая Excel-таблица CDs.xlsx по всем кодирующим областям. Всего хороших генов осталось 879.
Так как последовательность ШД довольно короткая, нужно было ограничить области поиска в геноме последовательностей, действительно являющихся ШД. Для этого надо было добавить координаты области поиска в таблицу описания генов. Это было сделано с помощью функции Excel ЕСЛИ с учетом направления гена.
Были взяты области длиной 16 пн перед началом CDs. Итоговый файл с информацией об областях поиска ШД: в файле с расширением txt (для скрипта) fragments.txt . Для получения самих областей поиска был использован скрипт fragments2fasta.py (команда python fragments2fasta.py –i fragments.txt -f genome.fasta –o fragments.fast). Выходной файл с короткими областями поиска: fragments.fast. В нем с помощью сайта МЕМЕ был найден предполагаемый мотив ШД. Параметры поиска показаны на рис. 1. (Поиск мотива от 4 до 10 нуклеотидов длиной, только на данной цепи, поиск максимум 3 мотивов).

Рис. 1. Параметры поиска на странице сервиса MEME suit. Результат поиска – 3 различных мотива предполагаемой последовательности ШД – приведен на рис. 2.
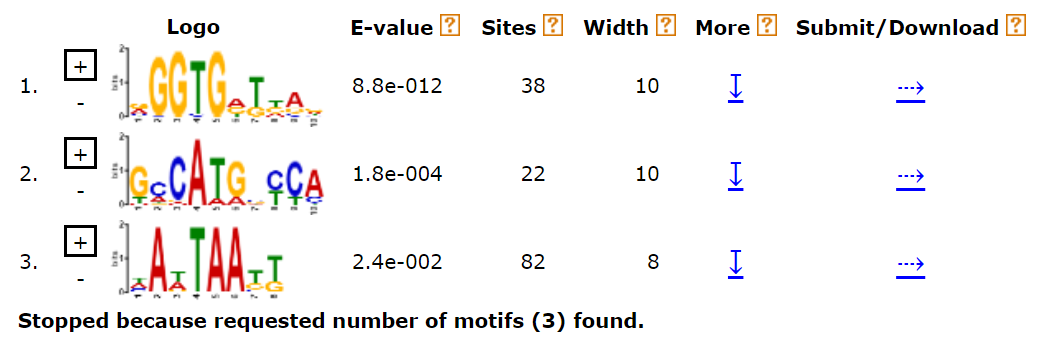
Рис.2. Три найденных сервисом МЕМЕ мотива в области поиска перед началом генов Vulcanisaeta moutnovskia 768-28.
Ссылка на MEME HTML output: http://meme-suite.org/opal-jobs/appMEME_4.11.214740194294781433117892/meme.html. Наилучшим мотивом можно назвать первый, с E-value = 8.8e-012. Его LOGO приведено на рис. 3.

Рис. 3. LOGO лучшего мотива Шайна-Дальгарно, найденного по областям поиска «хороших» генов Vulcanisaeta moutnovskia 768-28.
С помощью PWM, составленной по этому мотиву, был проведен поиск последовательностей ШД с данным мотивом во всем геноме. Для этого предварительно были получены upstream-области не только для «хороших» генов, а для всех кодирующих последовательностей, которых 2320 штук. Области поиска были расширены до 24 нуклеотидов до начала гена, фаста-файл был получен с помощью команды python fragments2fasta.py –i fragments2.txt -f genome.fasta –o fragments2.fasta. Полученный файл, в котором проводился поиск FIMO: fragments2.fasta. Порог поиска по E-value: 0.01.
Ссылка на FIMO HTML output: http://meme-suite.org/opal-jobs/appFIMO_4.11.214742054635281535835063/fimo.html. Итог работы сервиса – в файле fimo.xlsx.
По построенной PWM последовательность Шайна-Дальгарно была найдена всего в 732 upstream-областях генов (примерно 32% от общего кол-ва генов). Это не очень высокий результат, однако стоит учитывать, что организм относится к археям, а именно – к кренархеотам, для которых ранее было показано, что инициация трансляции у представителей этого таксона редко происходит за счет комплементарности последовательности ШД и 3’-конца 16S рРНК, и чаще – с помощью других механизмов инициации.
Задание 3. Определите сайты связывания данного транскрипционного фактора в данном участке хромосомы человека
Имя выданого файла – chipseq_chunk3.fasqc. Первым делом я просмотрел качество ридов, график из FastQC представлен на рис. 1.
Рис. 4. Качество ридов.
Смысла очищать (Trimmomatic) нет, т.к. риды отличного качества.
Далее был взят индексированый геном человека, сборка hg19. Затем риды были картированы командой:
$bwa mem /srv/databases/ngs/hg19/GRCh37.p13.genome.fa chipseq_chunk3.fastq > chipseq_chunk3.sam
Далее были использованы несколько команд:
$ samtools view -bSo chipseq_chunk3.bam chipseq_chunk3.sam | показывает все выраванивания, -b – сообщаем, что выходной файл формата bam; -S – игнорирование предыдущих версий samtools; -o – output file
$ samtools sort chipseq_chunk3.bam -T chip_temp -o chipseq_chunk3.sorted.bam | -T – референс в формате fasta; -o – output file; сортирует по левому концу
$ samtools index chipseq_chunk3.sorted.bam | индексирует файл
$ samtools idxstats chipseq_chunk3.sorted.bam > chipseq_chunk3.idxstats | статистика индекс файла к соответствующему исходному
$ samtools view -c chipseq_chunk3.sorted.bam | -с – вместо печати лишь считать выравнивания
Изначально было 6548 ридов, по 12 хромосоме откартировалось 6172.
Далее была использована macs2, с моделью не прошло, т.к. мало пиков, использовалась команда без модели, название было дано дефолтное NA_peaks:
$ macs2 callpeak –t chipseq_chunk3.sorted.bam –nomodel
Нашлось 6 пиков.
Далее было получено выравнивание. Удалось идентифицировать что-то похожее на консенсус с достаточно неплохим p-value(рис 5):
/srv/databases/ngs/tools/seqtk/seqtk subseq /srv/databases/ngs/hg19/GRCh37.p13.genome.fa NA_peaks.narrowPeak > my_peaks.fa
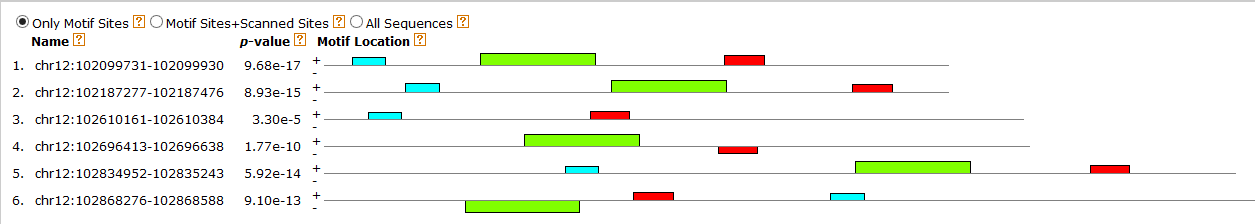
Рис. 5. Мотивы, найденные MEME.

Рис. 6. Лого красного мотива.
Далее в файл формата .narrowPeak были добавлены следующие строки:
track type=narrowPeak visibility=3 db=hg19 name="my_peaks" description="Peaks from chunk X"
browser position chr12:102099730-102869588
Этот файл скормил геномному браузеру. Результат на Рис. 7:
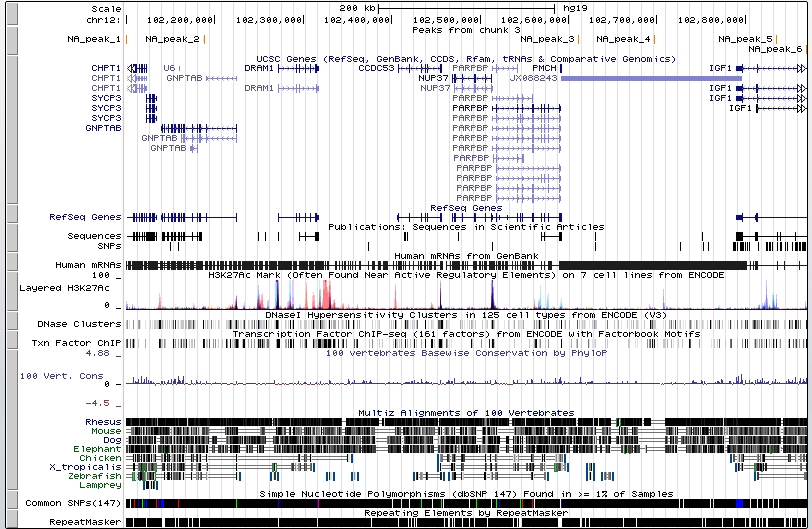
Рис. 7. Пики в геномном браузере.
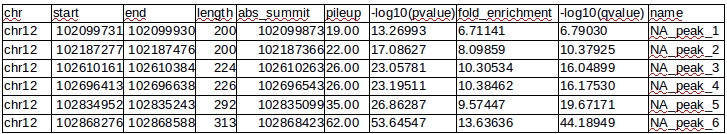
Рис. 8. Информация о пиках.
Чем больше величина -LOG10(pvalue), тем меньше числовое значение pvalue и, следовательно, тем достовернее пик. Самый достоверный пик - шестой.
Пик 1. Лежит в интроне гена CHPT11.
Пик 2. Лежит в интроне гена GNPTAB.
Пик 3. Лежит в экзоне гена GNPTAB.
Пик 4. Лежит в экзоне гена JX088243.
Пик 5. В итроне IGF1. . Пик 6. Тоже в интроне гена IGF1!
Задание 4
TBP является одним из ключевых ДНК-узнающих белков, необходимых для образования на промоторах генов комплекса TFIID и инициации транскрипции с помощью Pol II. Тем не менее, лишь часть промоторов имеет сигнал TATA-box, связываемый TBP. Консенсусная последовательность для связывания TBP - TATAWAAR.
Выбранный эксперимент - ChIP-seq анализ для TBP в стволовых эмбриональных клетках человека H1-hESC с использованием антител кролика.
Было описано три гена, транскрипция которых инициируется с помощью TBP, и один - без сигнала TATA-box в промоторной области.
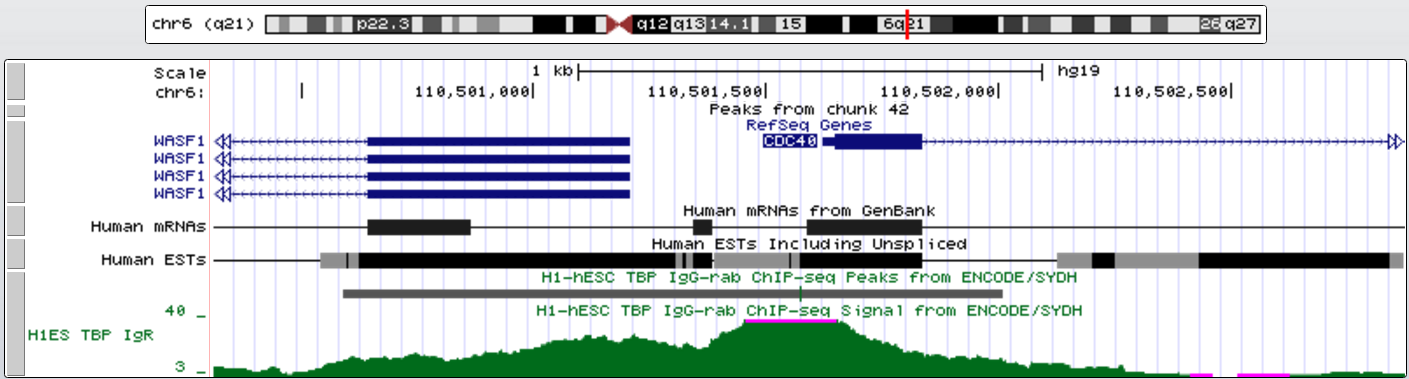
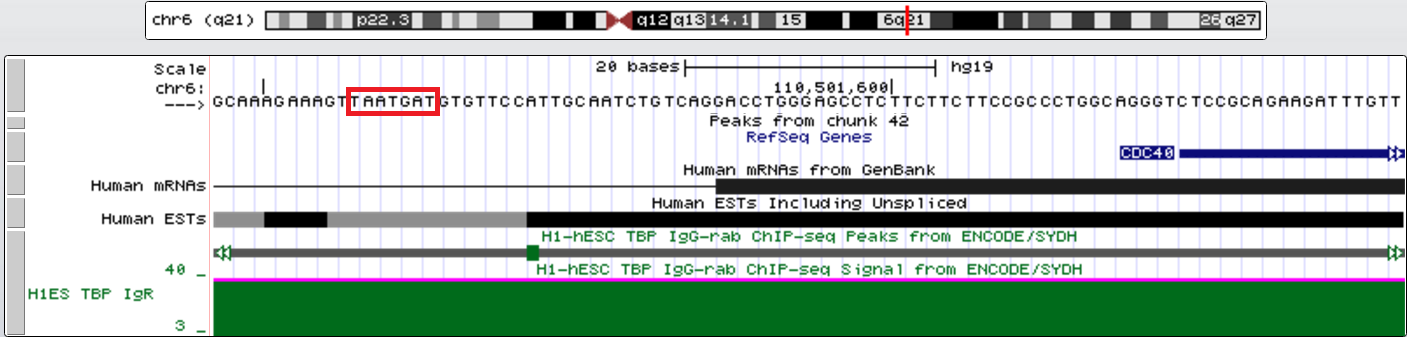
Первый ген с TATA-box - CDC40 (участвует в регуляции клеточного цикла, фактор процессинга пре-мРНК). Его координаты chr6:110501624-110553422, длина 51799, + цепь, 15 экзонов. TATA-box на расстоянии около 60 нт
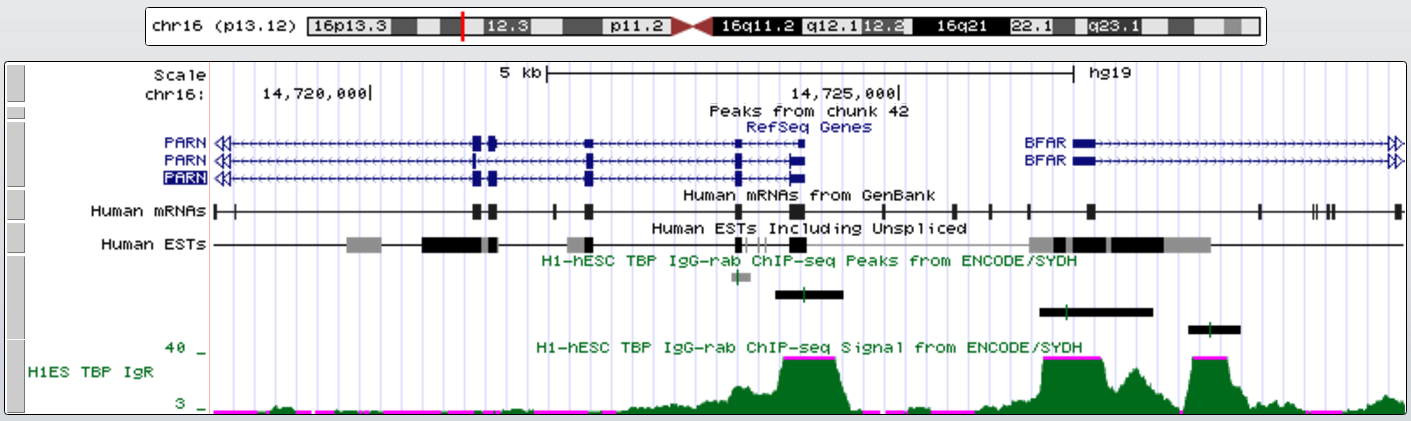
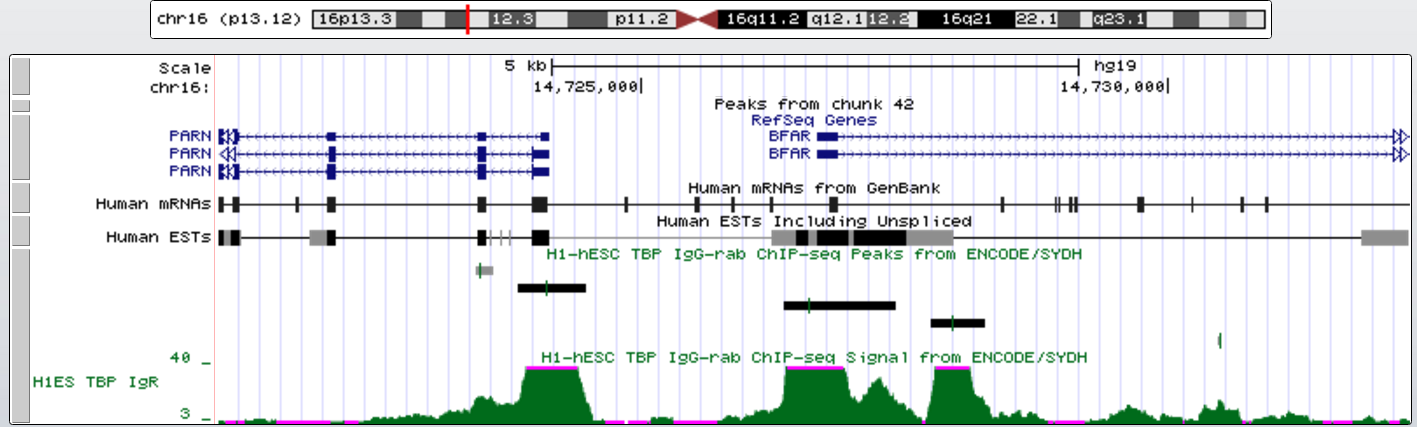
Второй ген с TATA-box - PARN (polyA-специфичная рибонуклеаза). Его координаты chr16:14529557-14724128, длина 194572, - цепь, 24 экзона. TATA-box на расстоянии около 60 нт.
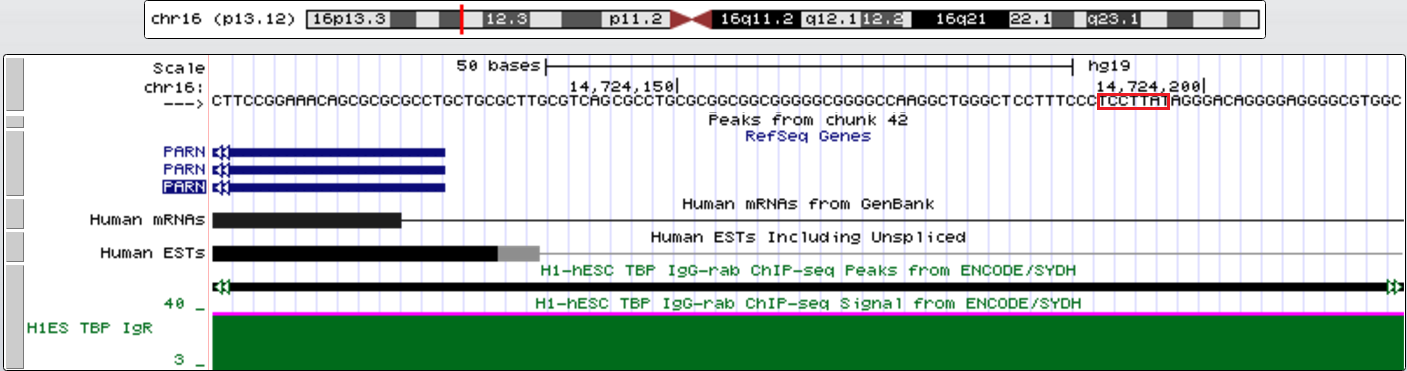
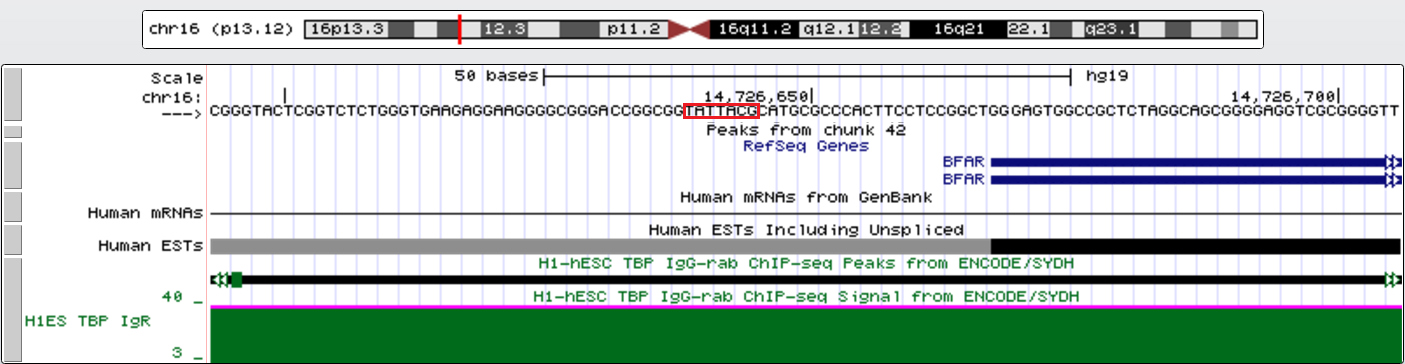
Третий ген с TATA-box - BFAR (бифункциональный регулятор апоптоза). Его координаты chr16:14726668-14763093, длина 36426, + цепь, 8 экзонов. TATA-box на расстоянии около 30 нт.
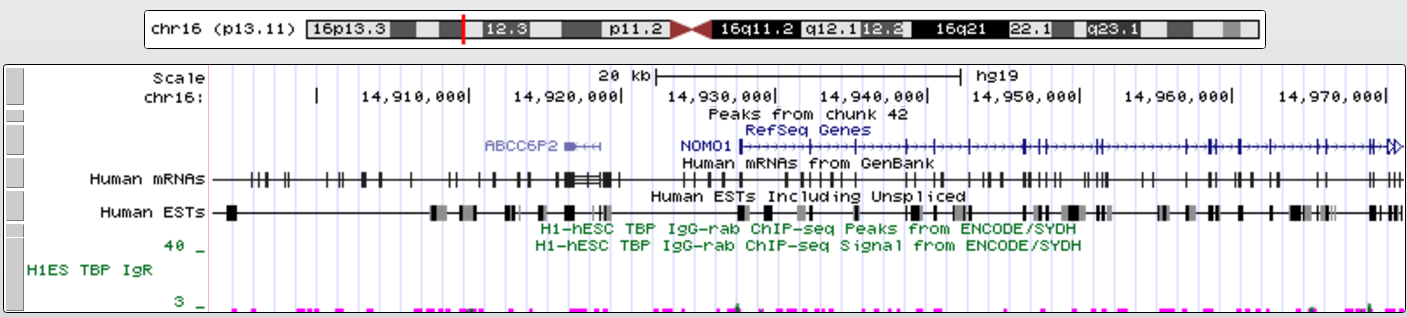
Пример гена без TATA-box - NOMO1 (NODAL modulator 1 - участвует в онтогенезе позвоночных). Его координаты chr16:14927643-14990014, длина 62372, + цепь, 31 экзон.
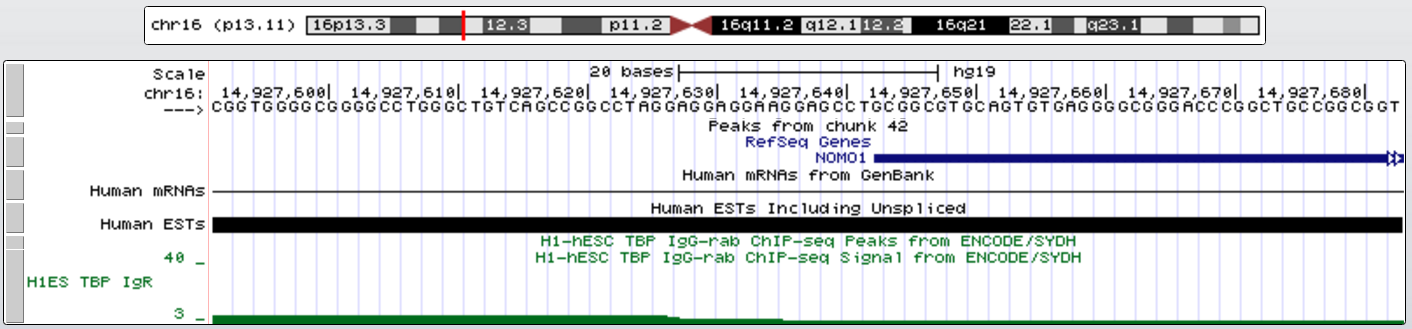
Во всех случаях TATA-box находился перед геном на расстоянии около 30-60 нт до старта транскрипции.
