Исследование структуры тРНК 1IL2
I. Краткое описание структуры в файле 1IL2.pdb
Молекулы: белок (цепи A и В - ASPARTYL-TRNA SYNTHETASE),
две цепи ДНК (С и D - ASPARTYL TRANSFER RNA).
Выбрана C цепь ДНК:
цепь C [901] 5' - UCCGUGAUAGUUUAAUGGUCAGAAUGGGCGCUUGUCGCGUGCCAGAUC
GGGGUUCAAUUCCCCGUCGCGGAGCCA -3' [976]
С учетом модифицированных оснований:
цепь C [901] 5' - uccgugauaguu(psu)aa(h2u)gg(h2u)cagaaugggcgc(psu)
uguc(1mg)cgugcagau(5mc)gggg(5mu)(psu)caauucccguc
gcggagcca - 3' [976]
На 3'-конце присутствует триплет CCA, к которому присоединяется аминокислота.
Измененные основания:
PSU C9 H13 N2 O9 P псевдоуридин-5'-монофосфат
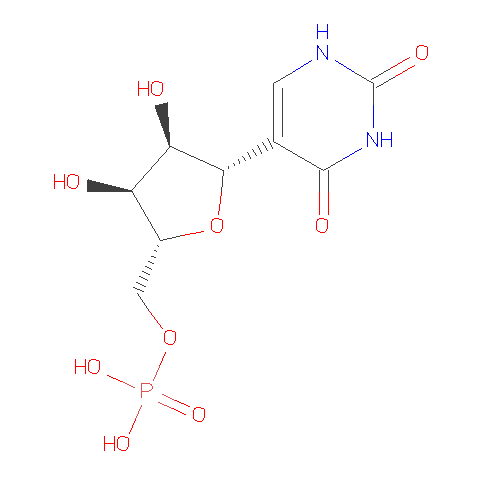
H2U C9 H15 N2 O9 P 5,6-дигидроуридин-5'-монофосфат.
Данная структура - комплекс аспартил-тРНК синтазы E.coli и аспартил тРНК дрожжей.
II. Исследование вторичной структуры.
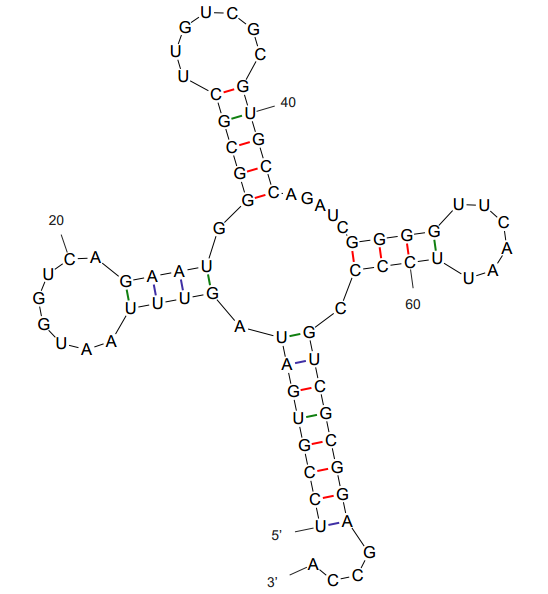
Строение тРНК.
Информация о водородных связях из файла, полученного find_pair и analyze:
----- for WC bp, + for isolated bp, x for helix change)
Strand I Strand II Helix
1 (0.003) C:.901_:[..U]U-----A[..A]:.972_:C (0.005) |
2 (0.004) C:.902_:[..C]C-----G[..G]:.971_:C (0.009) |
3 (0.004) C:.903_:[..C]C-----G[..G]:.970_:C (0.011) |
4 (0.012) C:.904_:[..G]G-----C[..C]:.969_:C (0.004) |
5 (0.008) C:.905_:[..U]U-*---G[..G]:.968_:C (0.010) |
6 (0.006) C:.906_:[..G]G-----C[..C]:.967_:C (0.008) |
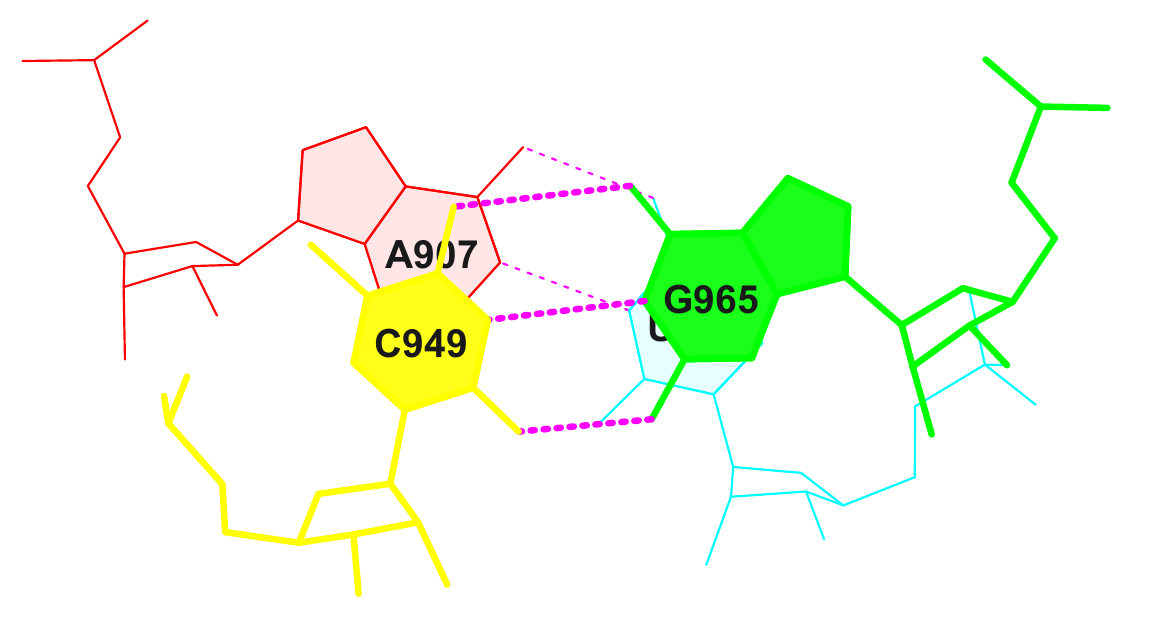
7 (0.006) C:.907_:[..A]Ax----U[..U]:.966_:C (0.004) |
8 (0.017) C:.949_:[5MC]c-----G[..G]:.965_:C (0.008) |
9 (0.005) C:.950_:[..G]G-----C[..C]:.964_:C (0.005) |
10 (0.006) C:.951_:[..G]G-----C[..C]:.963_:C (0.006) |
11 (0.012) C:.952_:[..G]G-----C[..C]:.962_:C (0.005) |
12 (0.009) C:.953_:[..G]G----xC[..C]:.961_:C (0.008) |
13 (0.012) C:.954_:[5MU]u-**-xA[..A]:.958_:C (0.007) |
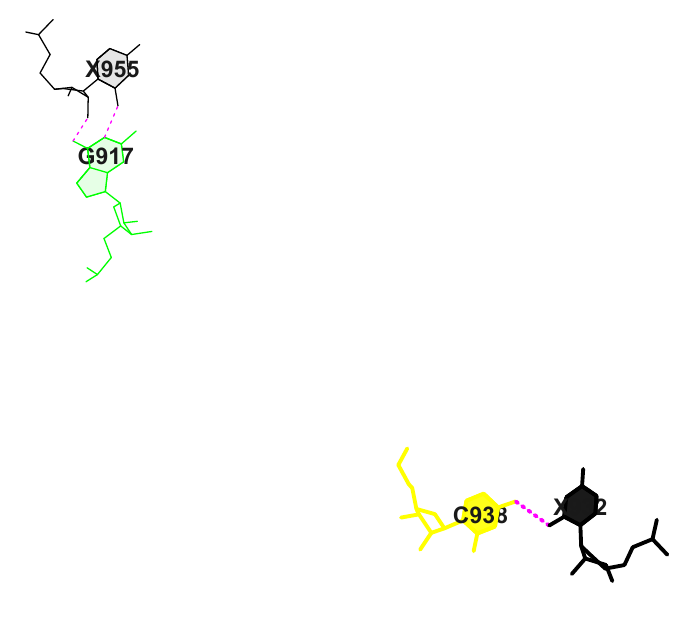
14 (0.043) C:.955_:[PSU]Px**+xG[..G]:.917_:C (0.005) x
15 (0.005) C:.938_:[..C]C-*---P[PSU]:.932_:C (0.044) |
16 (0.006) C:.939_:[..G]G-----C[..C]:.931_:C (0.006) |
17 (0.003) C:.940_:[..U]U-*---G[..G]:.930_:C (0.009) |
18 (0.010) C:.941_:[..G]G-----C[..C]:.929_:C (0.004) |
19 (0.010) C:.942_:[..C]C-----G[..G]:.928_:C (0.004) |
20 (0.003) C:.943_:[..C]C-----G[..G]:.927_:C (0.005) |
21 (0.005) C:.944_:[..A]Ax*---G[..G]:.926_:C (0.005) |
22 (0.009) C:.910_:[..G]G-*---U[..U]:.925_:C (0.004) |
23 (0.003) C:.911_:[..U]U-----A[..A]:.924_:C (0.009) |
24 (0.007) C:.912_:[..U]U-----A[..A]:.923_:C (0.007) |
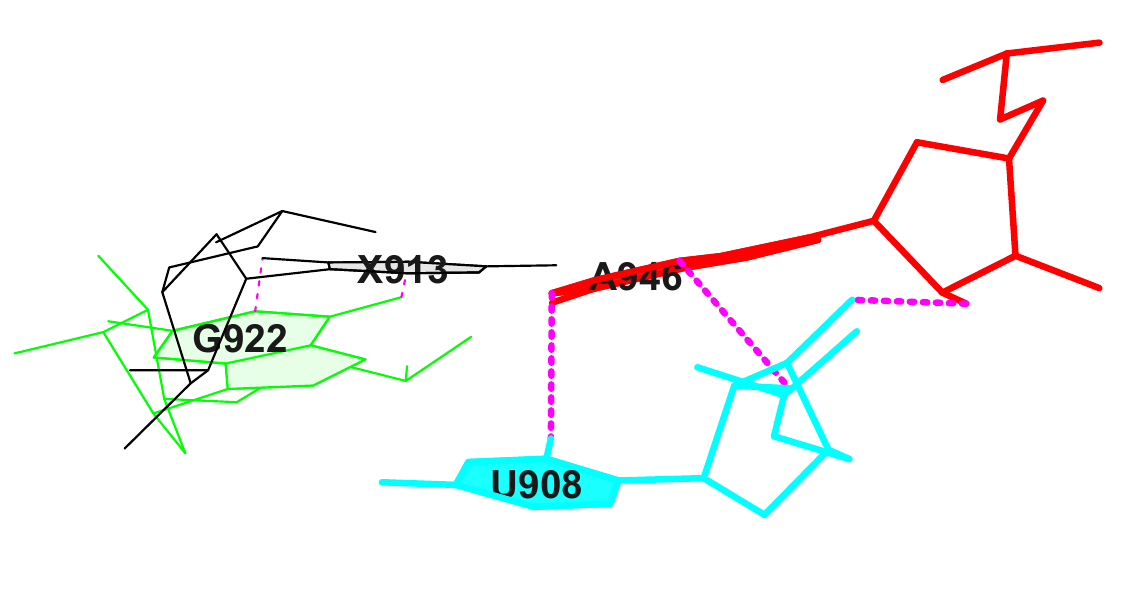
25 (0.044) C:.913_:[PSU]Px*--xG[..G]:.922_:C (0.005) |
26 (0.003) C:.908_:[..U]Ux**-xA[..A]:.946_:C (0.005) |
27 (0.003) C:.914_:[..A]A-*--xA[..A]:.921_:C (0.008) |
28 (0.004) C:.915_:[..A]A-**+xU[..U]:.948_:C (0.003) |
29 (0.115) C:.916_:[H2U]ux**+xU[..U]:.959_:C (0.006) x
30 (0.021) C:.918_:[..G]Gx---xC[..C]:.956_:C (0.007) +
По итогам её анализа формирую таблцу:
| Акцепторный стебель |
T-стебель |
Антикодоновый стебель |
D-стебель |
| 5' - 901 - 907 - 3' |
5' - 949 - 953 - 3' |
5' - 938 - 944 - 3' |
5' - 910 - 913 - 3' |
| 3' - 972 - 966 - 5' |
3' - 965 - 961 - 5' |
3' - 932 - 926 - 5' |
3' - 922 - 925 - 5' |
Выводы:
Наличие вариабельной петли: возможно A944-C948.
Остаток тимидина в T-петле: нет.
Наличие дигидроуридинов в D-петле: есть, PSU913
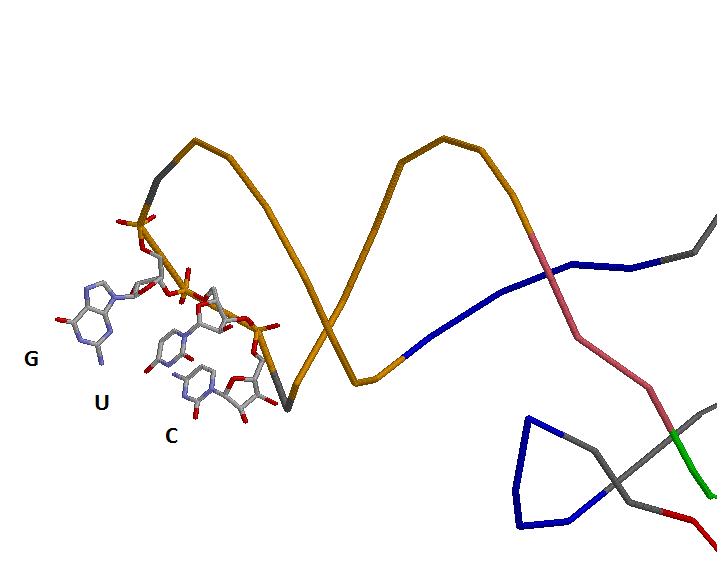
Антикодон: G934 U935 C936.
RasMol изображение тРНК, раскрашеное по петлям:
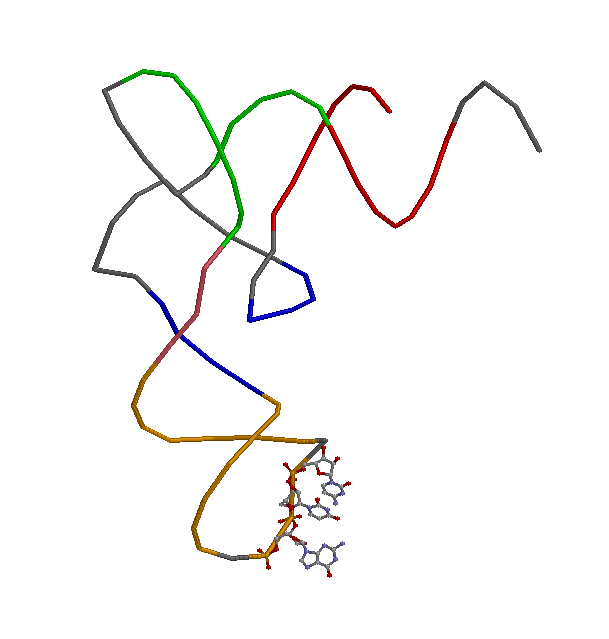
|
load 1IL2.pdb
select 901-907:C or 966-972:C
color red
select 949-955:C or 961-965:C
color green
select 910-913:C or 922-925:C
color blue
select 938-944:C or 926-932:C
color orange
select 944-948:C
color pink
select 934-936:C
cpk 50
wireframe 40
color cpk
|
Поддерживают структуру: 3 канонических и 5 неканонических (выделены цветом ниже) пар оснований.
13 (0.012) C:.954_:[5MU]u-**-xA[..A]:.958_:C (0.007) |
14 (0.043) C:.955_:[PSU]Px**+xG[..G]:.917_:C (0.005) x
26 (0.003) C:.908_:[..U]Ux**-xA[..A]:.946_:C (0.005) |
27 (0.003) C:.914_:[..A]A-*--xA[..A]:.921_:C (0.008) |
28 (0.004) C:.915_:[..A]A-**+xU[..U]:.948_:C (0.003) |
29 (0.115) C:.916_:[H2U]ux**+xU[..U]:.959_:C (0.006) x
30 (0.021) C:.918_:[..G]Gx---xC[..C]:.956_:C (0.007) +
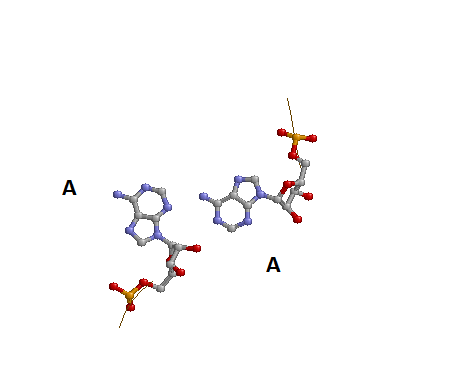
Пример неканонической пары аденин-аденин:
Предположительный анткодон:
III. Исследование третичной структуры.
IV. Предсказание вторичной структуры тРНК с помощью mfold и einverted.
С помощью алгоритма Зукера на сайте http://mobyle.pasteur.fr, получаю такую
картинку, на мой взгляд, наиболее
соотвтетствующую действительности (P=20). Неправильно определен T-стебель и антикодоновый стебель.
Далее, с помощью einverted пакета EMBOSS ищу инвертированные комплементарные участки.
Вообще же, для обоих методов, подобрать условия оказалось нелегко.
mfold постоянно вставлял петли в стеблях, которых не должно быть в тРНК.
einverted не выдавал вообще ничего при стандартных настройках и снижении
Minimum score tрreshold до нуля. Дальнейшие вариации значений штрафов и вознагражений привели к таким результатам.
При gap penalty 10, match score 5, mismatch score -5 (и не только в этом случае)
находился акцепторный стебель. Попытка вытянуть это выравнивание так, чтобы совпал ещё и
антикодоновый стебель, провалилась.
При gap penalty 20, match score 3, mismatch score -4 получилось найти T-стебель почти целиком правильно (см. таблицу).
Таблица сравнения реальной структуры тРНК и предсказанной.
| Участок структуры |
Позиции в структуре (по результатам find_pair) |
Результаты предсказания
с помощью einverted |
Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель |
5'- 901-907 -3'
3'- 972-966 -5'
8 пар |
предсказано 6 пар из 8 реальных |
предсказано 8 пар из 8 реальных |
| D-стебель |
5'- 910-913 -3'
3'- 922-925 -5'
4 пары |
не нашлись условия предсказания |
пресказано 4 пары из 4 реальных |
| T-стебель |
5'- 949-953 -3'
3'- 965-961 -5'
5 пар |
предсказано 4 пары из 5 реальных |
предсказано со сдвигом |
| Антикодоновый стебель |
5'- 938-944 -3'
3'- 932-926 -5'
6 пар |
не нашлись условия предсказаниия |
предсказано со сдвигом |
Вывод: для восстановления вторичной структуры тРНК неудобно использовать поиск инвертированных повторов. Алгоритм Зукера предусматривает некоторые
особенности строения вторичной структуры РНК и, таким образом, упрощает и ограничивает поиск. Тем не менее, алгоритм Зукера может допускать ошибки сдвига найденных последовательностей
в стебле. Такие ошибки не просто быстро найти и исключить из рассмотрения.
Главная страница
© Галицына Александра, 2011