Ознакомление с сервисом TreeTop
Ознакомимся с созданным в НИИФХБ имени А.Н. Белозерского сервисом TreeTop, для чего пошлем на вход программе созданный программой muscle файл с выравниваниями белков EFTS разных бактерий.
На выходе получаем два дерева, полученные разными алгоритмами (топологическим и кластерным), матрицу расстояний и графические изображения деревьев. Далее на картинке представлено графическое изображение дерева, полученного кластерным алгоритмом:
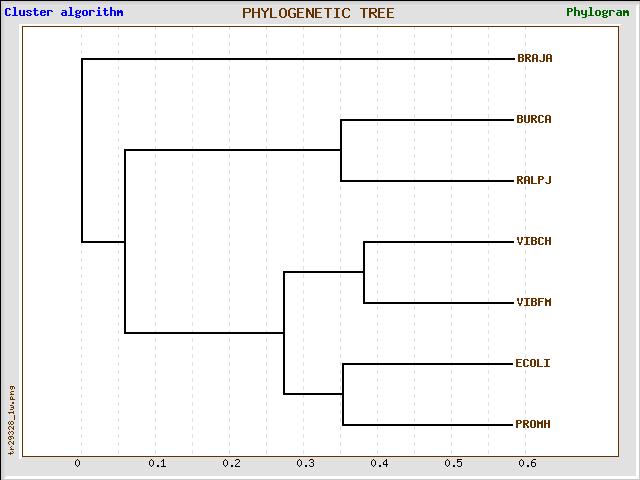
Если сравнивать полученное дерево с деревьями, полученными другими программами (см. Реконструкция и сравнение деревьев. Расстояния между последовательностями.), легко заметить, что они совпадают (и совпадает с правильным филогенетическим деревом). Это говорит в пользу использования этого сервиса. Так выглядит матрица расстояний между бактериями, полученная с помощью сервиса TreeTop:
1 2 3 4 5 6 7
1 BRAJA 0.000 0.588 0.573 0.615 0.605 0.560 0.585
2 BURCA 0.588 0.000 0.234 0.543 0.548 0.504 0.504
3 RALPJ 0.573 0.234 0.000 0.535 0.544 0.514 0.522
4 VIBCH 0.615 0.543 0.535 0.000 0.203 0.299 0.329
5 VIBFM 0.605 0.548 0.544 0.203 0.000 0.301 0.317
6 ECOLI 0.560 0.504 0.514 0.299 0.301 0.000 0.231
7 PROMH 0.585 0.504 0.522 0.329 0.317 0.231 0.000
Эта матрица сильно отличается от матрицы, полученной программой fprotdist, однако значения друг от друга отличаются примерно так же. Например, расстояния между VIBCH и ECOLI и между VIBFM и ECOLI в этой матрице тоже почти равны, то есть эти объекты мало отличаются от ультраметрических.
В целом, сервис TreeTop отлично справился с поставленной задачей, и впечатление от него осталось самое лучшее. Он объединил в себе несколько программ пакета EMBOSS и за короткий срок выдал правильный и доступно оформленный результат.
Назад
