A- и В- формы ДНК. Структура РНК
Задание 1
a) Построила модели структур A-, B- и Z-формы ДНК с помощью инструментов пакета 3DNA.
б) Пакет 3DNA один из популярных пакетов программ для анализа и простейшего моделирования
структур нуклеиновых кислот. Работает под операционной системой LINUX.
в) С помощью программы Рutty, используя протокол ssh, подсоединилась к серверу
kodomo.cmm.msu.ru. Создала и перешла в директорию Term3/Practice2.
г) Ввела следующие команды, что бы указать путь к 3DNA:
export PATH=${PATH}:/home/preps/golovin/progs/X3DNA/bin
export X3DNA=/home/preps/golovin/progs/X3DNA
д) С помощью программы fiber пакета 3DNA построила A-, B- и Z-форму дуплекса ДНК,
последовательность одной из нитей которого представляет собой 5 раз повторенную
последовательность "gatc".
е) Структуру дуплекса в А-форме сохранила в файле gatc-a.pdb,
структуру дуплекса в В-форме в файле gatc-b.pdb,
структуру дуплекса в Z-форме в файле gatc-z.pdb.
Задание 2
Упражнение 1 (Выделение разных атомов и химических группирровок)
Скрипт 1
Скрипт 2
Скрипт 3
Скрипт 4
Скрипт 5
Упражнение 2
Нашла документы на сайте PDB (идентификаторы 1eiy и 1hw2).
Также проверила наличие обеих цепей ДНК.
Документ для 1eiy.
Документ для 1hw2.
Упражнение 3
Разрывов не найдено.
Задание 3
Упражнение 1
1) Рассмотрела структуру и определила большую и малую бороздку.
2) Выбрала пятое заданное азотистое основание (гуанин) в A-форме.
Определила,какие атомы основания явно обращены в сторону большой бороздки, а какие в сторону малой.
С помощью ChemSketch получила изображение основания, выделила красным цветом атомы,
смотрящие в сторону большой бороздки (G5.N7 G5.C8 G5.O4* G5.O2P), синим - в сторону малой (G5.N1 G5.N2 G5.C3* G5.C4*).
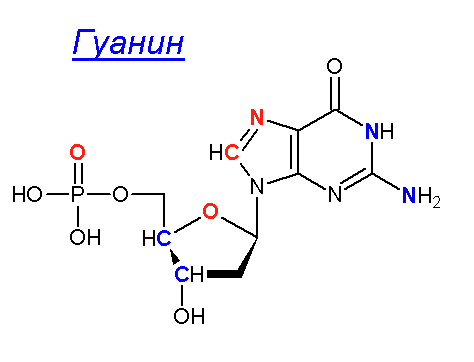
1) Рассмотрела структуру и определила большую и малую бороздку.
2) Выбрала пятое заданное азотистое основание (гуанин) в цепи B-форме.
Определила,какие атомы основания явно обращены в сторону большой бороздки, а какие в сторону малой.
С помощью ChemSketch получила изображение основания, выделила красным цветом атомы,
смотрящие в сторону большой бороздки (G5.O6 G5.N7 G5.O2P), синим - в сторону малой (G5.N2 G5.N3 G5.C5* G5.O3*).
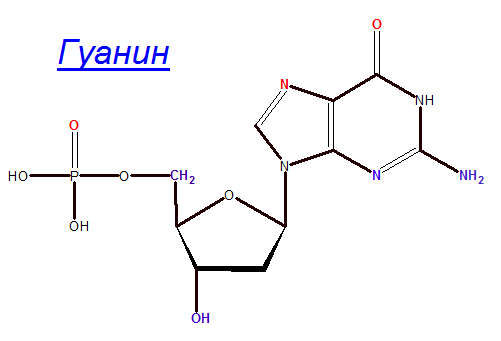
1) Рассмотрела структуру и определила большую и малую бороздку.
2) Выбрала пятое заданное азотистое основание (гуанин) в Z-форме.
Определила,какие атомы основания явно обращены в сторону большой бороздки, а какие в сторону малой.
С помощью ChemSketch получила изображение основания, выделила красным цветом атомы,
смотрящие в сторону большой бороздки (G5.O6 G5.N7 G5.C1*), синим - в сторону малой (G5.O1P G5.O2P G5.O3* G5.N2 G5.N3).
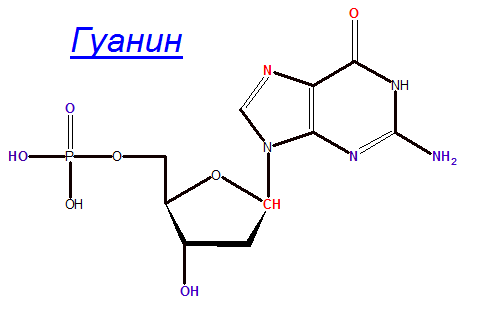
Упражнение 2
|
A-форма |
B-форма |
Z-форма |
Тип спирали (правая или левая) |
правая |
правая |
левая |
Шаг спирали (A) |
28.03 |
33.8 |
43.5 |
Число оснований на виток |
11 |
10 |
12 |
Ширина большой бороздки |
17.12 (G) |
16.60 (A) |
18.58 (C) |
Ширина малой бороздки |
7.98 (A) |
11.69 (G) |
9.86 (G) |
Упражнение 3
| Торсионные углы у гуанина |
| |
α |
β |
γ |
δ |
ε |
ζ |
χ |
А-форма
(приведенная в презентации) |
-62 |
173 |
52 |
88 или 3 |
178 |
-50 |
-160 |
| А-форма |
-51,70 |
174,80 |
41,71 |
79,08 |
-147,08 |
-75,07 |
-157,22 |
B-форма
(приведенная в презентации) |
-63 |
171 |
54 |
123 или 131 |
155 |
-90 |
-117 |
| B-форма |
-29,87 |
136,38 |
31,10 |
143,42 |
105,85 |
-160,52 |
-97,97 |
Задание 4
Упражнение 1
Для анализа структур нуклеиновых кислот использовала программы find_pair и analyze.
find_pair -t gatc-a.pdb stdout | analyze получила файл gatc-a.out
find_pair -t gatc-b.pdb stdout | analyze получила файл gatc-b.out
find_pair -t gatc-z.pdb stdout | analyze получила файл gatc-z.out
В созданных файлах можно найти разную информацию, а также значения всех торсионных углов.
Для a-формы
Для b-формы
Для z-формы
В наибольшей степени отличаются углы ε в в-форме.
Получила файл с информацией для структуры тРНК: 1eiy.out.
Значения всех торсионных углов в файле 1eiy.txt.
Мне кажется, что данная мне структура тРНК больше похожа на А-форму.
Получила файл с информацией для заданной мне ДНК: 1hw2_old.out.
Значения всех торсионных углов в файле 1hw2_old.txt.
Файл Exel со среднем значением каждого из торсионных углов.
Самый "деформированный" нуклеотид - 6G (4 угла отличаются).
На вторм месте - 14А и 15G (3 угла отличаются).
Упражнение 2
Информацию о водородных связях переписали в файл 1eiy(2).txt.
Акцепторный стебель - участoк 1-7 и комплементарный ему участок 72-66
T-стебель - участок 49-53 и комплементарный ему участок 65-61
D-стебель - участок 10-13 и комплементарный ему участок 25-22
Антикодоновый стебель - участок 39-44 и комплементарный ему участок 31-26
Неканонические пары оснований в файле обозначены '*', в данной мне тРНК их 7 штук.
В данной структуре тРНК, для стабилизации третичной структуры, присутствуют многочисленные
дополнительные водородные связи, например:
26 (0.019) C:..18_:[..G]G-**+-U[..U]:..55_:C (0.009) |
27 (0.026) C:..19_:[..G]Gx---xC[..C]:..56_:C (0.014) x
Первая дополнительная водородная связь представляет собой неканоническое взаимодействие,
другая - каноническое.
Упражнение 3
Самое большое перекрывание у пары:
15 GC/GC 7.25( 3.64) 0.00( 0.00) 0.00( 0.00) 4.43( 1.60) 11.68( 5.24)
Из файла stacking.pdb вырезала данные о 15 GC/GС с помощью команды
ex_str -9 stacking.pdb step9.pdb
Затем получила изображение с помощью команды:
stack2img -cdolt step9.pdb step9.ps
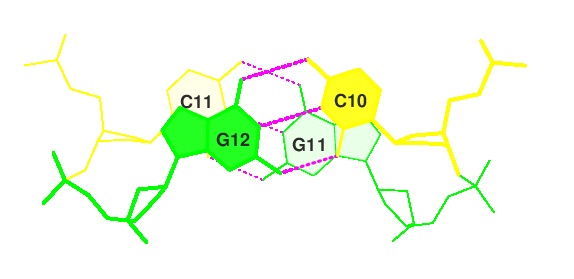
©Melnichuk Anastasia



