| Главная | Семестры | Проекты | Обо мне |
1.Поиск в геноме участков, кодирующих белки, похожие на заданный
Для работы взят белок ENGB_BACSU (P38424) из Bacillus subtilis и искали гомологи в геноме Streptococcus agalactiae.
Команда для создания индексных файлов пакета BLAST+:
>makeblastdb -in sa_genome.fasta -out sa -dbtype nucl
Далее воспользуемся программой TBLASTN для поиска гомологов. Для этого выполним команду:
>tblastn -query engb_bacsu.fasta -db sa -out engb_sa.txt -evalue 0.001
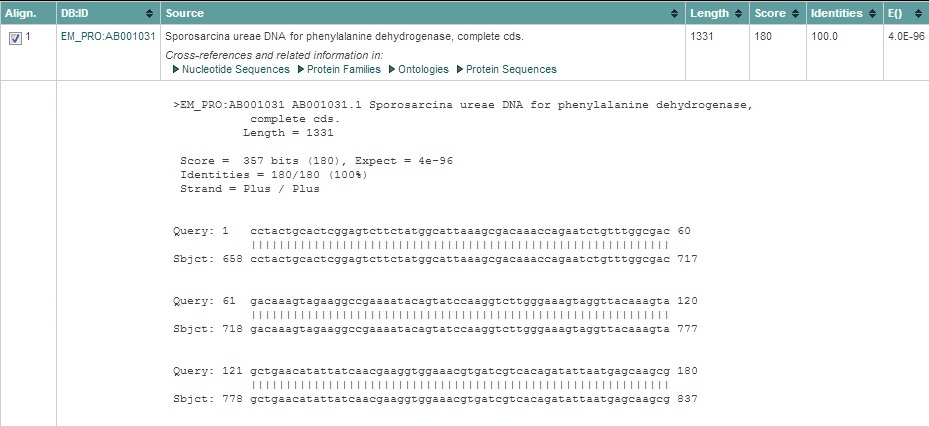
Файл engb_sa.txt.
Поиск гомологов белка ENGB_BACSU (P38424) в геноме Streptococcus agalactiae.
| Число находок с E-value < 0,001 | 2 |
| E-value лучшей находки | 2e-71 |
| Название последовательности с лучшей находкой |
Streptococcus agalactiae NEM316 complete (AL766850) |
| Координаты лучшей находки (от-до) | 118585 - 119151 |
| Доля последовательности вашего белка, вошедшая в выравнивание с лучшей находкой |
189/195≈0,97 |