Молекулярная динамика биологических молекул в GROMACS
Цель данного занятия ознакомится с возможностями моделирования молекулярной динамики.
Моделирование плавления алфа-спирального пептида в формамиде
Подготовка файлов
- Создайте рабочую директорию на диске Н: типа Ivanov/md
- Нам даны файлы:
- Координаты пептида, 2xl1.pdb.
- Файл с ячейкой уравновешеных молекул формамида, fam_em.gro.
- Файл дополнительной топологии для формамида, fam.itp.
- файл праметров для минимизации энергии em.mdp.
- файл праметров для "утряски" воды pr.mdp pr.mdp.
- файл праметров для молекулярной динамики md.mdp.
- Скопируем файлы по WinSCP на 172.16.0.140.
- Построим файл топологии системы в силовом поле amber99sb и файл с координатами в формате Gromacs.
pdb2gmx -f 2xl1.pdb -o pep -p pep -ff amber99sb -water tip3p
- Сделаем небольшой отступ в ячейке от пептида.
editconf -f pep.gro -o pep_ec -d 1.5
- Проведём оптимизацию геометрии системы, что бы удалить "плохие" контакты в молекуле.
grompp -f em -c pep_ec -p pep -o pep_em -maxwarn 1
mdrun -deffnm pep_em -v
- Добавим в ячейку молекулы формамида.
genbox -cp pep_em -p pep -cs fam_em.gro -o pep_s
- Теперь надо изменить в текстовом редакторе файл тополгии pep.top. После строчки:
; Include forcefield parameters
#include "fam.itp"
Добавим количество молекул формамида в запись [ molecules ]
[ molecules ]
; Compound #mols
Protein 1
FAM 2426
Нейтрализуем заряд системы. Это делаем в два шага: строим tpr и запускаем genion.
grompp -f em -p pep -c pep_s
genion -s pep_s -o pep_si -p pep -np X
- Проведём "утряску"
grompp -f pr -c pep_si -p pep -o pep_pr -maxwarn 1
mdrun -deffnm pep_pr -v
Переформатируем pep_pr.gro и pep_si.gro в pdb формат. Как можно видеть, ячейка заполнена молекулами формальдегида и также собственно пептидом.
Запуск вычисления на суперкомпьютере
- Копируем файлы в папку Mintaev на skif-mgu:
scp -r md/* skif:Mintaev/
- Запускаем тестовое моделирование на суперкомпьтере.
grompp -f md -c pep_pr -p pep -o pep_md -maxwarn 1
mpirun -np 16 -maxtime 5 -q test /home/golovin/progs/bin/mdrun_mpi -deffnm pep_md -v
- Ошибок не оказалось, поэтому запускаем основное моделирование.
mpirun -np 16 -maxtime 5 -q test /home/golovin/progs/bin/mdrun_mpi -deffnm pep_md -v
Ориентировочное время счёта 10 часов
Анализ результатов моделирования поведения пептида в формамиде
Пакет программ Gromacs предоставляет много инструментов для анализа траекторий и свойств динамики. Суть любого анализа сводится к пониманию специфики динамики конкретной системы.
Силовое поле используемое при построении топологии топологии: amber99sb
- Заряд система: -10
- Размер и форму ячейки(нм): 5.06500, 4.67000, 4.22100
- Минимизация энергии:
- Алогритм минимизации энергии:
integrator = l-bfgs
- Алгоритм расчёта электростатики и Ван-дер-Ваальсовых взаимодействий:
; Method for doing electrostatics coulombtype = Cut-off
- Модель, которой описывался растворитель
implicit_solvent = No
растворитель: формальдегид
- Утряска растворителя:
- Для биополимеров, укажите параметр который обуславливает неподвижность биополимера.
define = -DPOSRES
- Число шагов: 10**4
- Длина шага: 1 фс
- Алгоритм расчёта электростатики и Ван-дер-Ваальсовых взаимодействий: coulombtype = pme
- Алгоритмы термостата и баростата:
Tcoupl = Berendsen
Pcoupl = no
- Основной расчёт МД:
- Время моделирования: 7 часов 12 минут 42 секунды , количество процессоров=16, эффективность маштабирования: 100%.
- Если моделирование окончилось с ошибкой, указать ошибку.
- Длину траектории: 20 нс
- Число шагов: 10**7
- Длина шага: 2 фс
- Алгоритм интегратора: md
- Алгоритм расчёта электростатики и Ван-дер-Ваальсовых взаимодействий:
coulombtype = pme
vdw-type = Cut-off
- Алгоритмы термостата и баростата :
Tcoupl = v-rescale
Контроль температуры по методу "Velocity rescale". Pcoupl = Berendsen
Визуальный анализ
Переформатируем файлы trr, xtc с координатами после рассчёта в файл PDB.
trjconv -f pep_md.xtc -s pep_md.tpr -o pep_pbc_1.pdb -skip 20 -pbc mol
Получившийся файл pdb.
На рис. изображены некоторые состояния альфа спирали во время моделирования
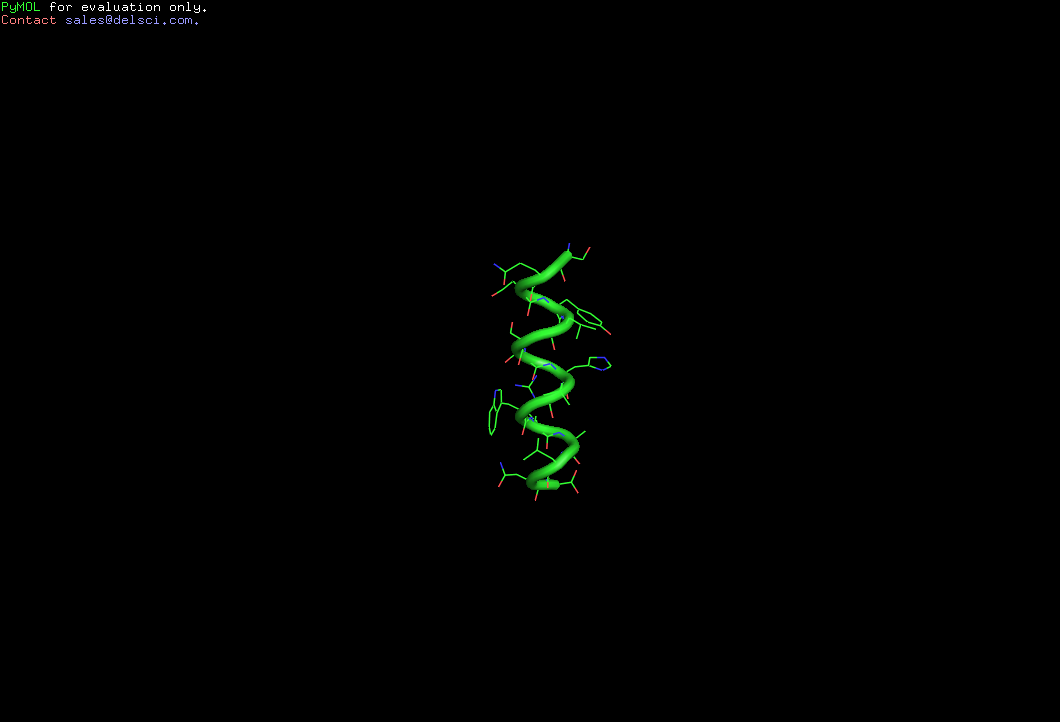
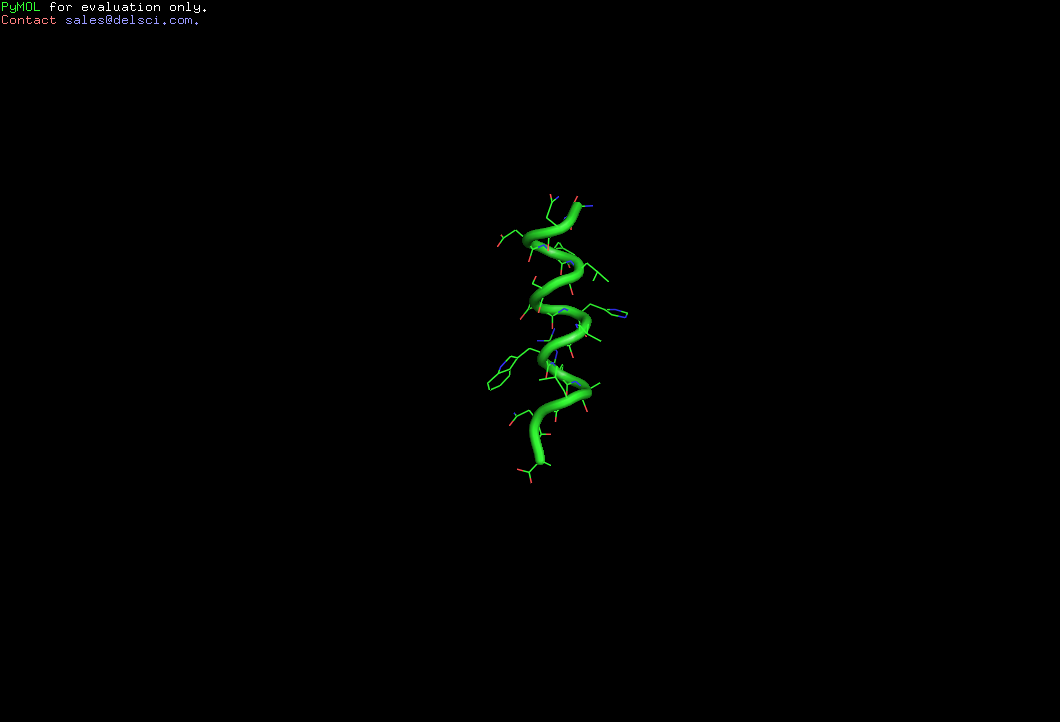
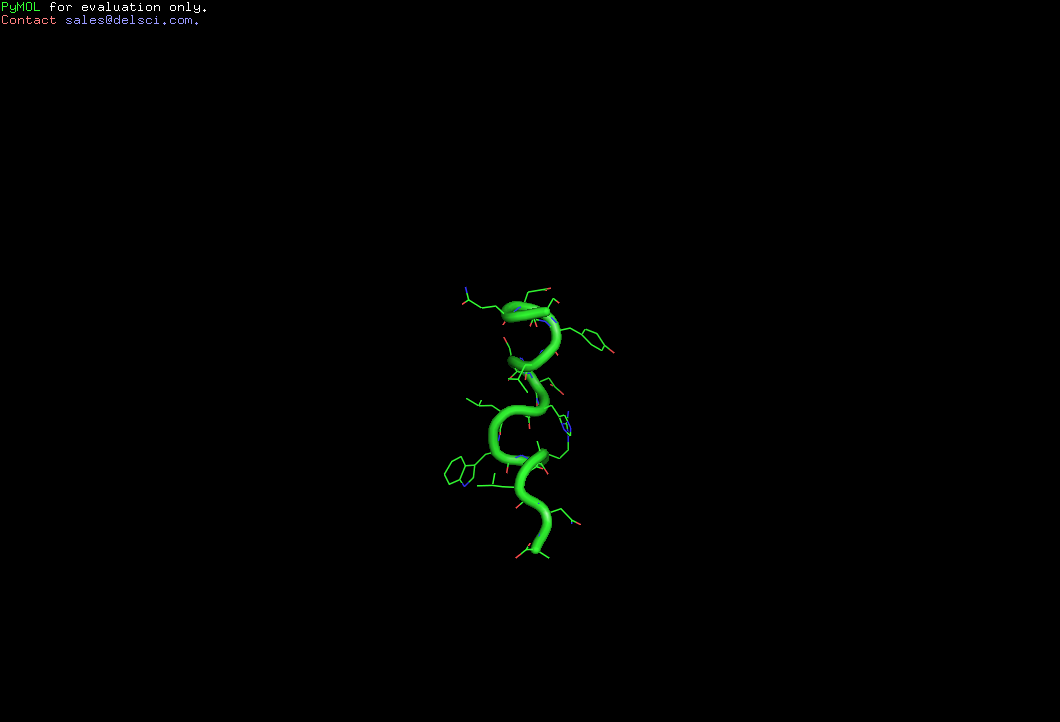
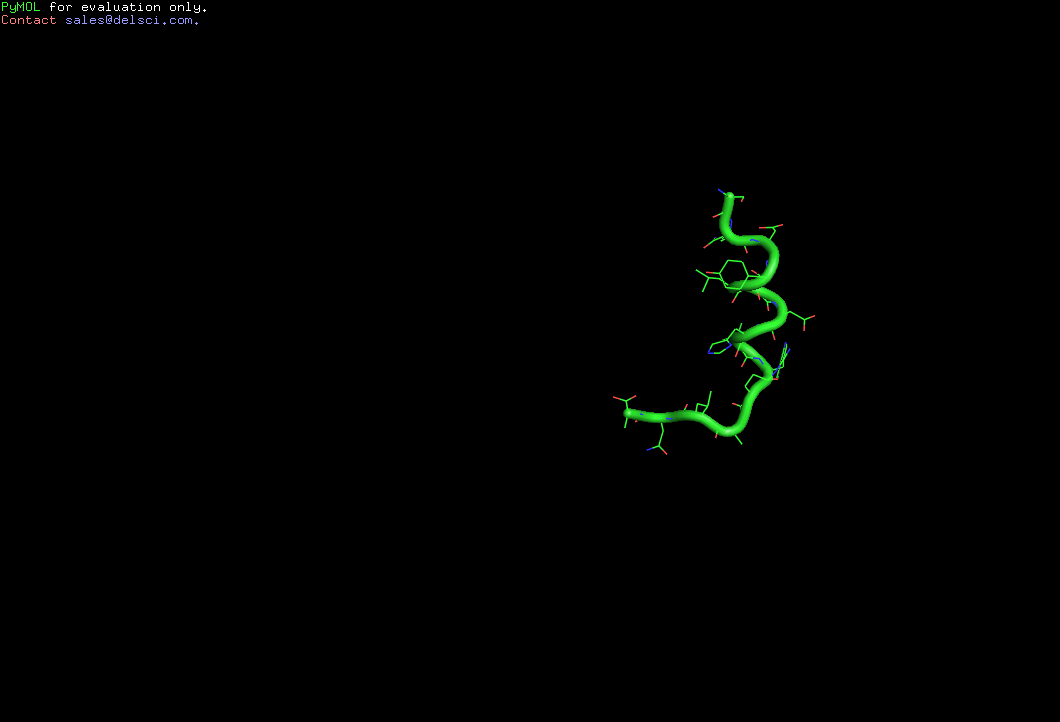
Определим средне-квадратичное отколонение в ходе моделирования
g_rms -f pep_md.xtc -s pep_md.tpr -o rms_1
g_rms -f pep_md.xtc -s pep_md.tpr -o rms_2 -prev 400
- Определим изменение гидрофобной и гидрофильной поверхности в ходе конформационного перехода
g_sas -f pep_md.xtc -s pep_md.tpr -o sas_pep.xvg
Построим в excel зависимость изменения гидрофобной гидрофильной поверхностей доступных растворителю от времени.
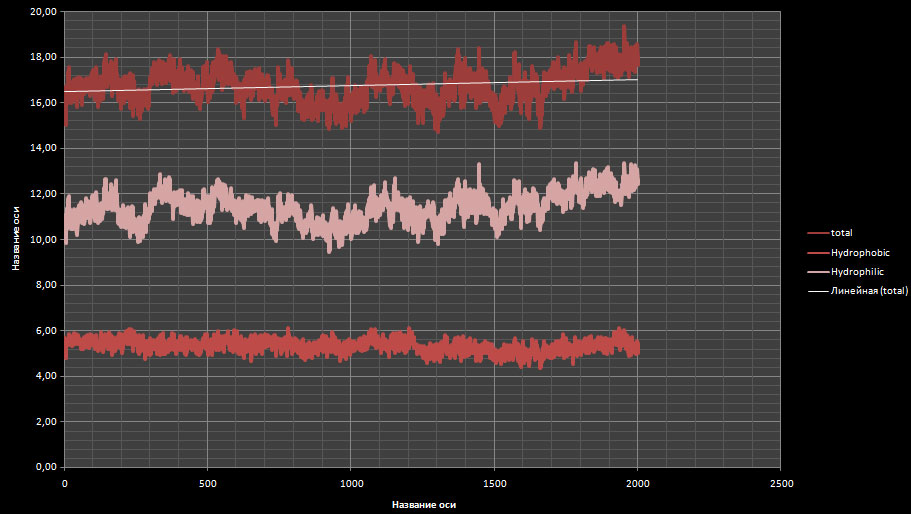
На графике можно видеть увеличение гидрофильной поверхности спирали для для лучшего доступа молекул формальдегида, что соответствует тому, что формальдегид является полярным растворителем.
- Расчёт колчества образуемых водородных связей. между пептидом и пептидом:
g_hbond -f pep_md.xtc -s pep_md.tpr -num hbond_pep
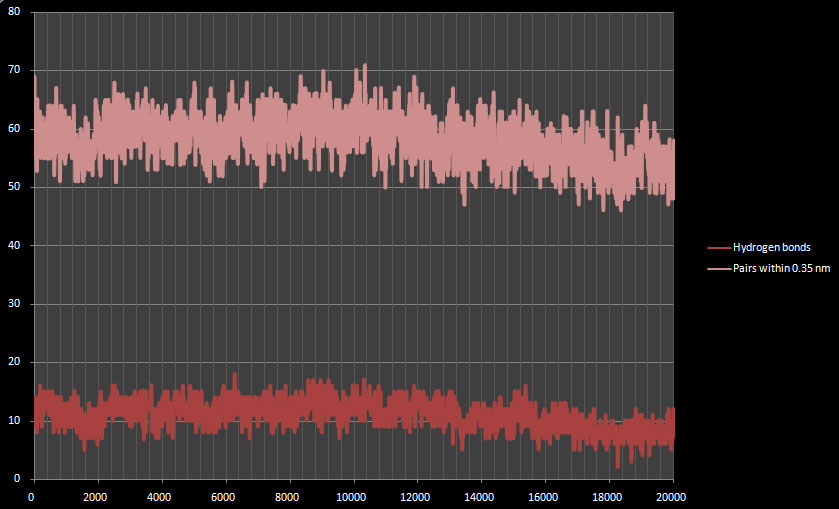
- количество вдородных связей пептид-формамид:
g_hbond -f pep_md.xtc -s pep_md.tpr -num hbond_pep_sl
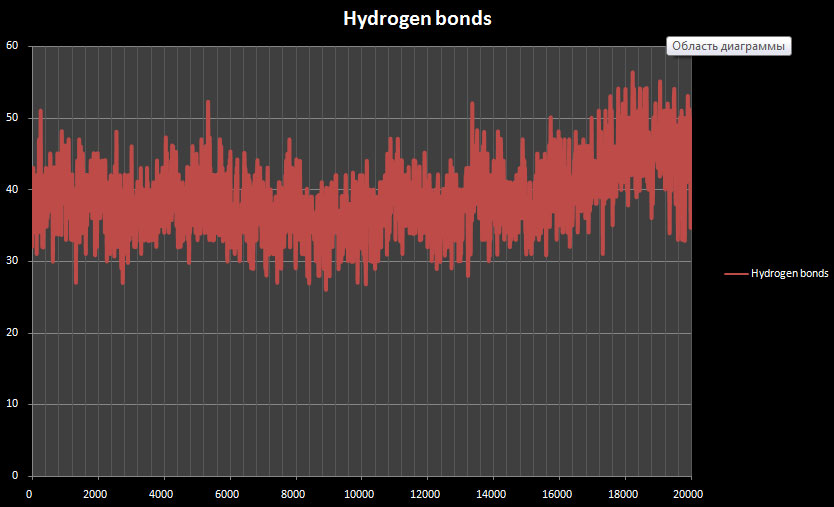
Как видно из графиков, количество водородных связей в альфа спирали падает, при этом увеличивается количество водородных взаимодействий с растворителем, что объясняется тем, что растворитель полярный и может образовывать водородные связи по мере увеличения поверхности доступа альфа спирали.
- Построим зависимость вторичной структуры от времени.
do_dssp -f pep_md.xtc -s pep_md.tpr -o ss
# Для просмотра переведём xpm в eps
xpm2ps -f ss.xpm -o ss.eps -by 10
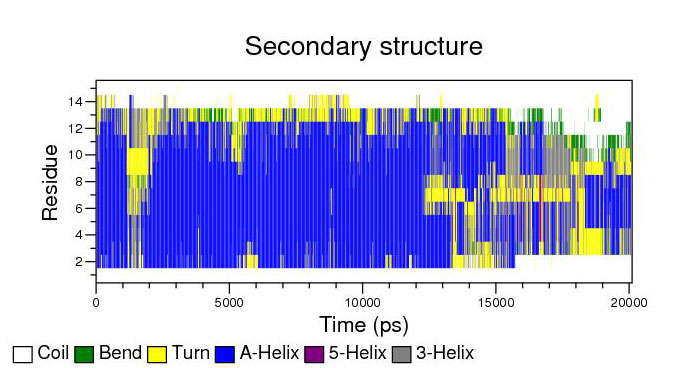
Во время моделирования очень серьезно подверглась изменению вторичная структура, практически она на половину была расплавлена. Причем, разрушился та ее часть, где было меньше экранирующих радикалов и радикалов с полярной составляющей.
