Практикум 11
Задание 1
Получилось 437 находок: 367 принадлежат археям (Formating options -> archaea -> Reformat); 70 - бактериям
Таблица 1. Сравнение находок.
Находка |
Белок |
Организм |
Длина выравнивания |
Bit-score |
Identities |
Positives |
E-value |
Лучшая |
hypothetical protein |
Vulcanisaeta moutnovskia |
422 |
871 |
100% |
100% |
0.0 |
Из середины |
hypothetical protein |
euryarchaeote SCGC AAA261-E04 |
412 |
122 |
28% |
46% |
1e-27 |
Худшая |
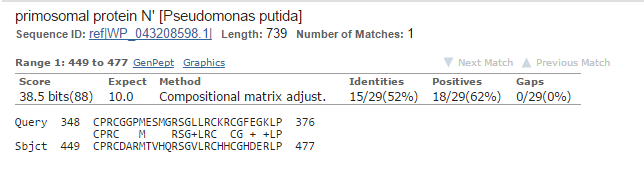
primosomal protein N' |
Pseudomonas putida |
29 |
38.5 |
52% |
62% |
10 |
Сами выравнивания:
Рис 1. Лучшее
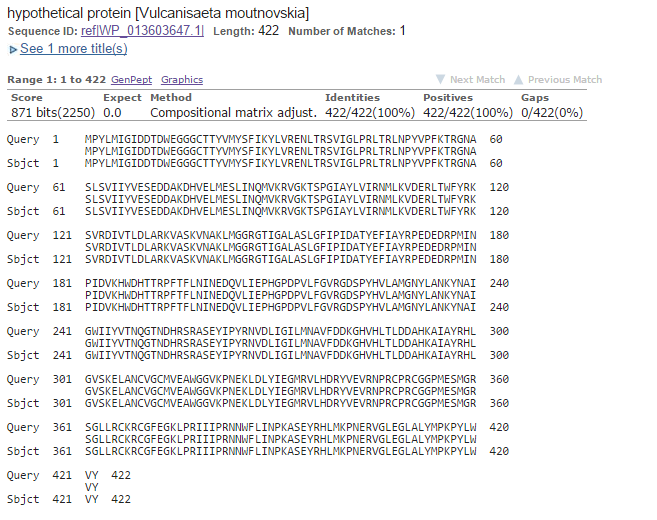
Рис 2. Из середины
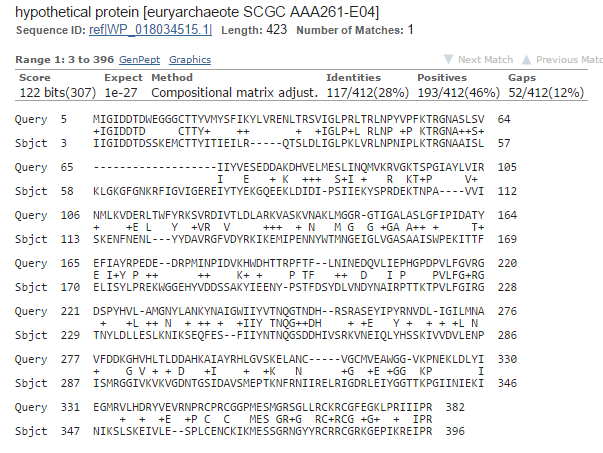
Рис 3. Худшее
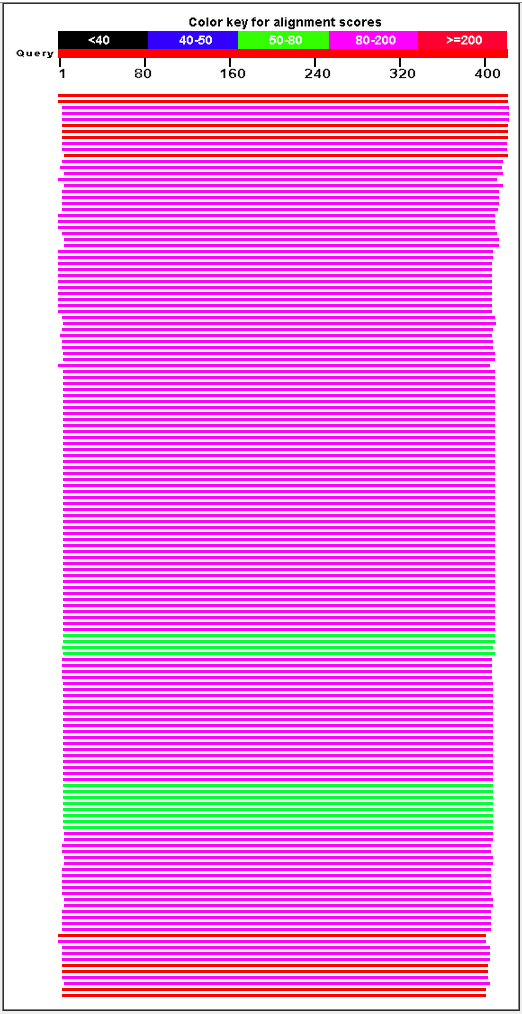
В качестве критерия гомологичности я приму E-value = 0,001 и Query cover = 80%. Тогда белок имеет 261 гомолога. Что интересно, если попросить бласт отсортировать находки по query cover, те что >80% сортируются нормально, а остальные идут вразнобой, поэтому я и взял 80% как условный критерий.
Бласт дает графическое изображение только до 94% QC.
Рис 4. Графическое изображение находок.
Задание 2
Взял hypothetical protein [alpha proteobacterium SCGC AB-629-F11], он бактериальный. Запустил бласт для бактерии. Среди списка белков нашел его же ( по идентификатору WP_018036287.1). Выравнивания совпадают. Score и остальные параметры совпадают. Только E-value отличается(уменьшился с 6e-14 до 4e-14). Объясняется это тем, что второй банк меньше, а значит и случайность находки меньше.
Задание 3
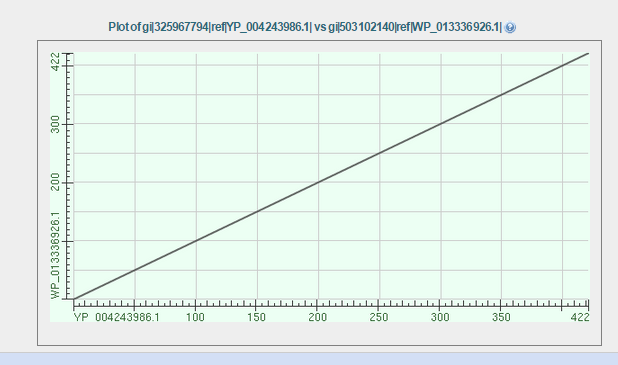
Выбрал последовательность hypothetical protein [Vulcanisaeta distributa]. Query cover 100%
Рис 5. Карта локального сходства
Особенности полученного выравнивания:
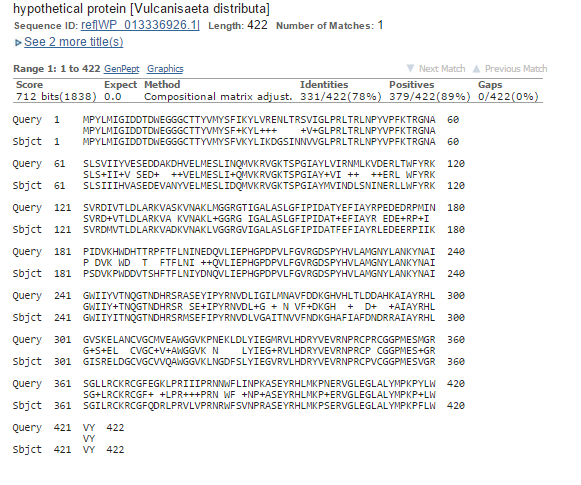
1. Query cover - 100%. То есть последовательности полностью гомологичны, что видно на рисунке 6.
Рис 6. Парное выравнивание
Задание 4
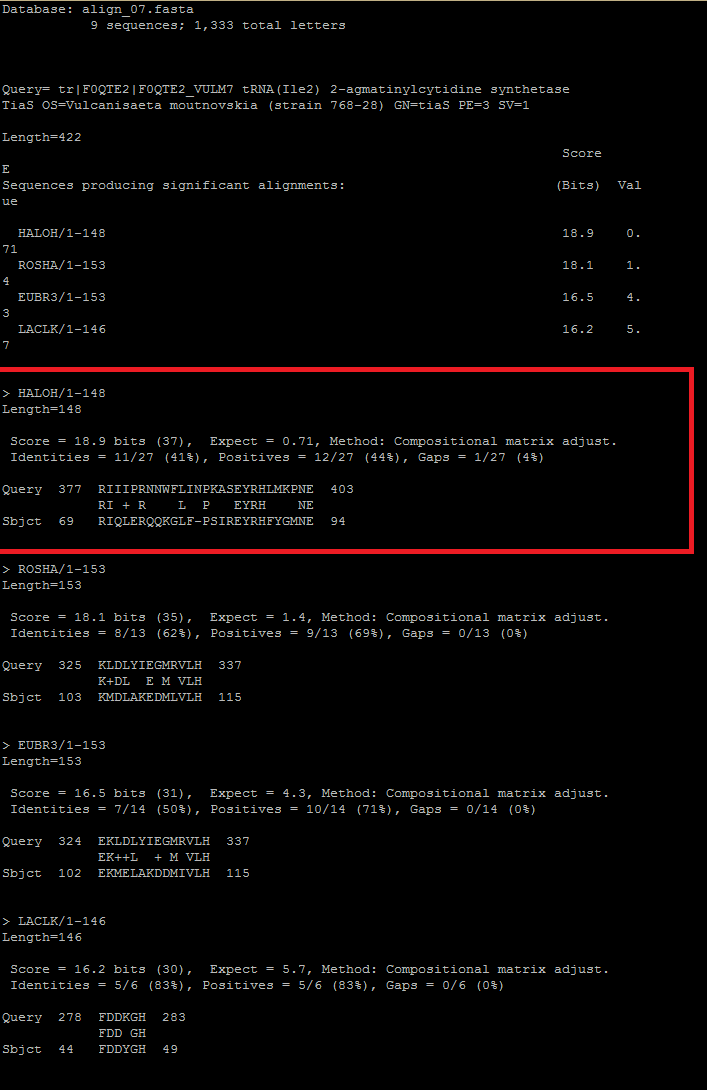
Взял Седьмое выравнивание из 8 практикума, удалил гепы, проиндексировал командой makeblastdb. Затем запустил выравнивания с помощью blastp. Что получилось можно увидеть на рисунке 7 или в отдельном файле. Красным выделено лучшее.
Рис 7. Работа blastp
Находка |
Длина выравнивания |
Bit-score |
Identities |
Positives |
E-value |
HALON |
27 |
18.9 |
41% |
44% |
0.71 |
Хоть и процент идентичных и сходных остатков не меньше чем у тех последовательностей, что я назвал гомологами в п.1, эти гомологами считать нельзя, потомку что длина этих выравниваний в десятки раз меньше того, что выдавал бласт, как, собственно, и Bit score, а E-value при этом очень большой ( хотя при уменьшении базы во 2 задании Е-value уменьшался), а значит полученные совпадения вероятнее всего случайны.
