Практикум 13
Задание 1
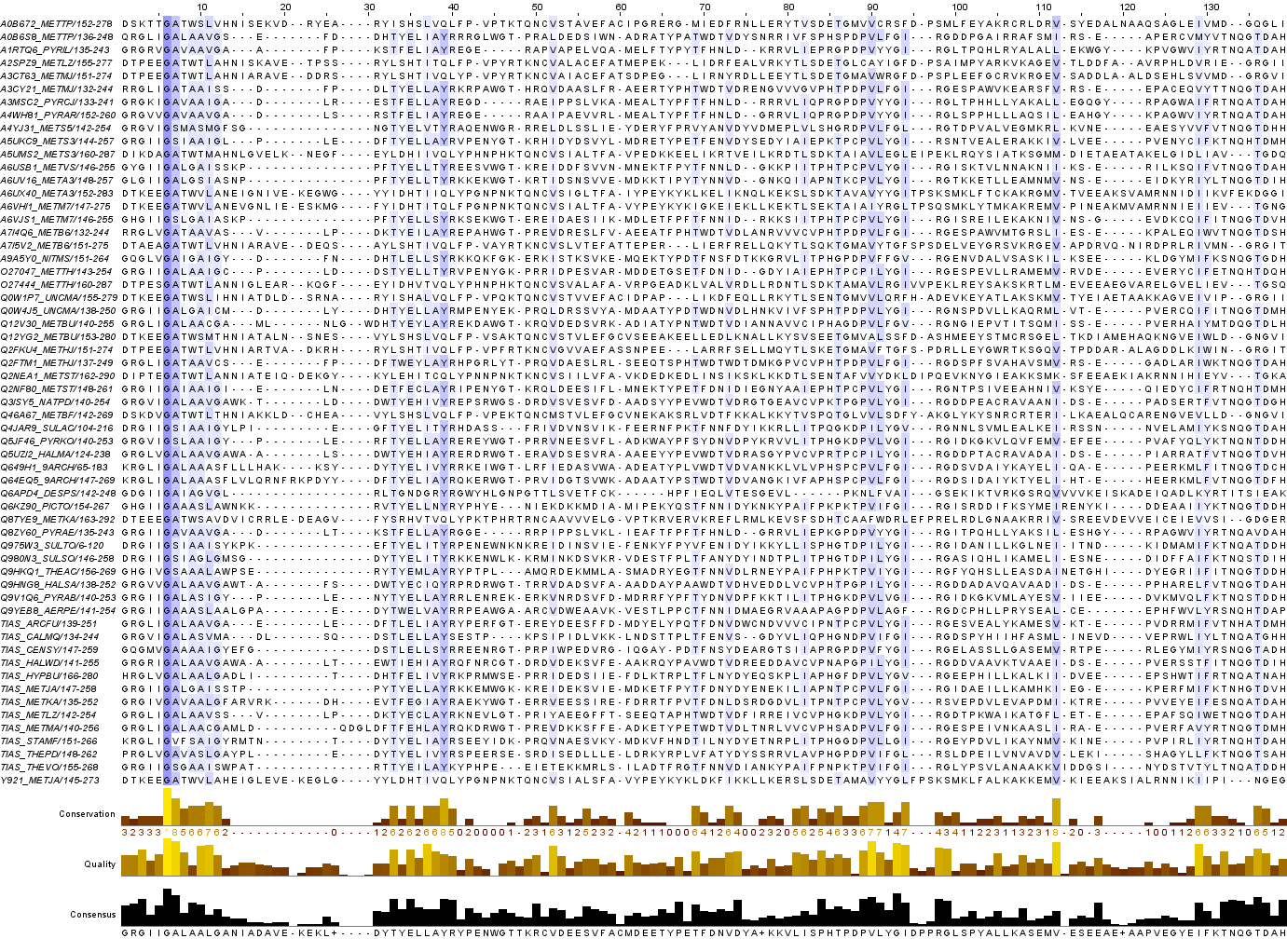
Провел поиск доменов в Pfam a (Pfam b не использовал, т.к. он некурируемы, а следовательно может быть недоств=оверным). В результате получил одно семейство доменов с неизвестной функцией. Координаты домена в моем белке: 145-254; Bit-score = 78; E-value = 4.5e-22. Seed выравнивание
Рис 1. Seed выравнивание.
Задание 2
В моем сиде самый большой блок состоял всего из двух колонок, поэтому мне пришлось сократить количество последовательностей до 24. В итоге удалось выделить блок из шести колонок(рис 2)
Рис 2. Блок из сида

При помощи сервиса Weblogo был получен LOGO данного блока.
Рис 3. LOGO

С помощью jalview была получена консенсусная последовательность данного блока.
Задание 4
Построил слабый и сильный паттерны по LOGO. Они не сильно отличаются друг от друга потому что колонки в моем блоке почти полностью консервативны.
Сильный паттерн - GRG[ILV][IV]G : 47 находок
Слабый паттерн - G[RQ]G[ILVM][IV]G : 117 находок
Мой белок - tRNA(Ile2) 2-agmatinylcytidine synthetase TiaS, а среди белков, которые нашлись что по сильному паттерну, что по слабому белки этой группы составляют очень малую часть. Объясняется это очень просто: есть в моем блоке недостаток - его размер всего 6 колонок, так что каким бы сильным ни был паттерн велика вероятность, что в некой не гомологичной последовательности случайным образом попадется эта последовательность.
