Ресеквенирование. Поиск полиморфизмов у человека (5 хромосома)
Использованные команды:
Команда |
Что делает |
fastqc chr5.fastq |
анализ качества чтений |
java -jar /usr/share/java/trimmomatic.jar SE -phred33 chr5.fastq chr5-trimmed.fastq TRAILING:20 MINLEN:50 |
очиcтка чтений: удаление нуклеотидов с качеством ниже 20, затем удаление чтений длиной меньше 50 |
bwa index chr5.fasta |
индексирование референсной последовательности |
bwa mem chr5.fasta chr5-trimmed.fastq > chr5.sam |
выравнивание очищенных чтений с проиндексированной референсной последовательностью |
samtools view chr5.sam -b -o chr5.bam |
перевод выравнивания в бинарный формат |
samtools sort -T /tmp/chr5_sorted -o chr5_sorted.bam chr5.bam |
сортировка выравнивания по координате начала чтения в референсной последовательности |
samtools index chr5_sorted.bam |
индексирование отсортированного файла |
samtools idxstats chr5_sorted.bam |
для определения, сколько чтений откартировалось |
samtools mpileup -uf chr5.fasta chr5_sorted.bam -o chr5snp.bcf |
создание файла с полиморфизмами |
bcftools call -cv chr5snp.bcf -o chr5snp.vcf |
перевод .bcf файла в .vcf формат (в список отличий) |
perl /nfs/srv/databases/annovar/convert2annovar.pl -format vcf4 chr5snp.vcf -outfile chr5snp.avinput |
создание файла для annovar из .vcf файла |
perl /nfs/srv/databases/annovar/annotate_variation.pl -geneanno -dbtype refGene -buildver hg19 chr5snp.avinput -outfile chr5snp_refgene /nfs/srv/databases/annovar/humandb/ |
аннотация по базе данных refgene (hg19) |
perl /nfs/srv/databases/annovar/annotate_variation.pl -filter -dbtype snp138 -buildver hg19 chr5snp.avinput -outfile chr5snp_snp138 /nfs/srv/databases/annovar/humandb/ |
аннотация по базе данных dbsnp |
perl /nfs/srv/databases/annovar/annotate_variation.pl -filter -dbtype 1000g2014oct_all -buildver hg19 chr5snp.avinput -outfile chr5snp_1000g2014oct /nfs/srv/databases/annovar/humandb/ |
аннотация по базе данных 1000genomes |
perl /nfs/srv/databases/annovar/annotate_variation.pl -regionanno -dbtype gwasCatalog -buildver hg19 chr5snp.avinput -outfile chr5snp_gwas /nfs/srv/databases/annovar/humandb/ |
аннотация по базе данных gwas |
perl /nfs/srv/databases/annovar/annotate_variation.pl -filter -dbtype clinvar_20150629 -buildver hg19 chr5snp.avinput -outfile chr5snp_clinvar /nfs/srv/databases/annovar/humandb/ |
аннотация по базе данных clinvar |
Подготовка чтений
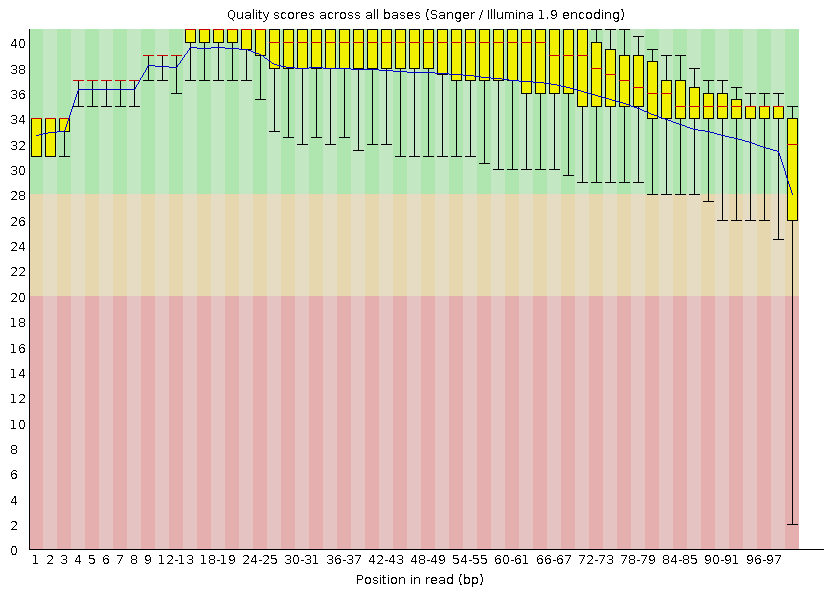
Анализ качества до очистки:
Анализ качества после очистки:
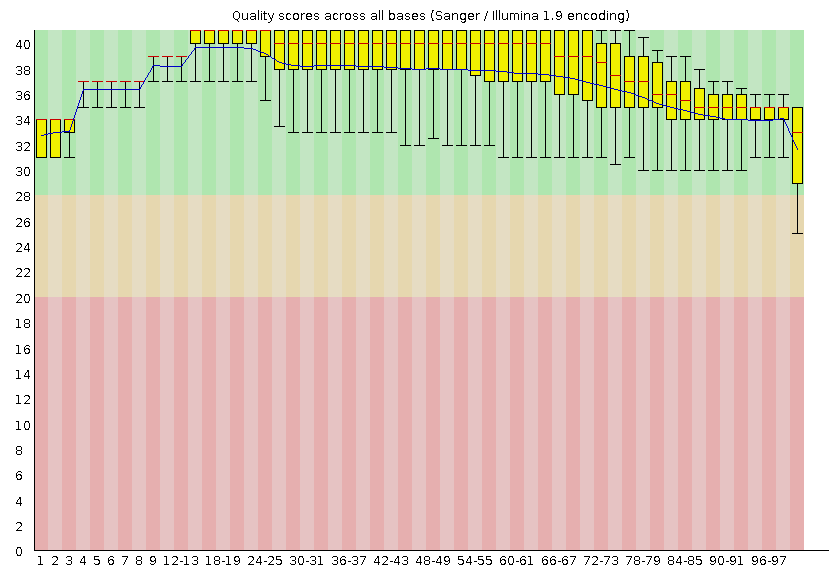
До очистки было 8208 ридов, после очистки осталось 8114: отброшено 94.
Параметр TRAILING:20 позволил отрезатьс конца каждого чтения нуклеотиды с качеством ниже 20, параметр MINLEN:50 позволил удалить чтения длиной меньше 50 нуклеотидов.
Картирование чтений
Команды в таблице.

На хромосому откартировались все риды (значение 0 в четвертом столбце говорит о том, что некартированных ридов не осталось). Странно что количество откартированных ридов на 1 меньше, чем количество оставшихся после чистки.
== Поиск SNP и инделей ==
Найдено 32 полиморфизма: 4 индели и 28 однонуклеотидных замен.
Координата |
Тип полиморфизма |
Референс |
Чтения |
Глубина покрытия |
Качество чтений |
35857177 |
замена |
G |
C |
114 |
221.999 |
35867343 |
замена |
T |
C |
32 |
184.009 |
35857308 |
вставка |
T |
TC |
45 |
178.458 |
Аннотация SNP
Результаты анализа:
Анализ по RefGene выдал 2 файла (variant_function, exonic_variant_function) и разбил SNP на группы:
Позиция SNP - exonic: 5; intronic: 26; 3'UTR: 1
Гомозиготных замен: 17; гетерозиготных: 15
SNP попали в 3 гена: IL7R, CAPSL, HMGCR, причем у всех трех хотя бы один SNP попал в экзон По информации из файла chr5snp_refgene.exonic_variant_function - из 5 экзонных:
не привели к изменению аминокислотной последовательности (synonymous)- 0;
привели - 4 (в генах IL7R, CAPSL); один привел к удалению рамки считывания (frameshift deletion) - в гене HMGCR В продукте гена IL7R из-за SNP произошли изменения: I66T, V138I, T244I В продукте гена CAPSL из-за SNP произошли изменения: R85Q
- Анализ по dbsnp выдал 2 файла:
dropped: имеют rs (аннотированы в базе) - таких SNP 27
filtered: не имеют rs - таких SNP 5
- Анализ по 1000genomes выдал 2 файла:
dropped: аннотированы в базе - таких SNP 24
filtered: не имеют rs - таких SNP 8
- Анализ по gwas выдал файл:
gwasCatalog: аннотированы в базе - таких SNP 4
- Анализ по clinvar выдал 2 файла:
dropped: аннотированы в базе - таких SNP 3
filtered: не аннотированы - таких SNP 29
Клиническая аннотация SNP: некоторые SNP были в ходе GWAS ассоциированы с рассеянным склерозом, диабетом 1 типа, ненормальным уровнем холестерина. В clinvar 3 SNP: 2 патогенны, связаны с возникновением тяжёлого комбинированного иммунодефицита, влияние еще одного неясно, однако указано, что ни один не "протестирован" (и только он один есть в gwas тоже).
Вся полученная информация была сведена в одну таблицу.
Выделенные желтым SNP представляют интерес, так как они экзонные, есть в gwas, (один из них и в clinvar), замены несинонимичные.
