Практикум 4
Задание №1
Для начала. было необходимо получить последовательности 16S рибосомальной РНК для выбранных в предыдущих прктикумах бактерий. Найти их удалось на ftp сервере NCBI в файлах .frn.
Проект jalview(общий для всего практикума)
Конкретно, последовательности были получены из следующих организмов:
- Agrobacterium tumifaciens AGRRK*
- Burkholderia cenocepacia BURCA
- Pseudomonas aeruginosa PSEAE
- Erwinia tasmaniensis ERWT9
- Yersinia pestis YERPE
- Vibrio cholerae VIBCH
- Haemophilus influenzae HAEIN
- Proteus mirabilis PROMH
Последовательности помеченые звездочкой были заменены аналогичные из других видов, из-за отутствия на сервере.
Далее последовательности были выровнены, а по выравниванию было построено методом Neighbor Joining дерево.
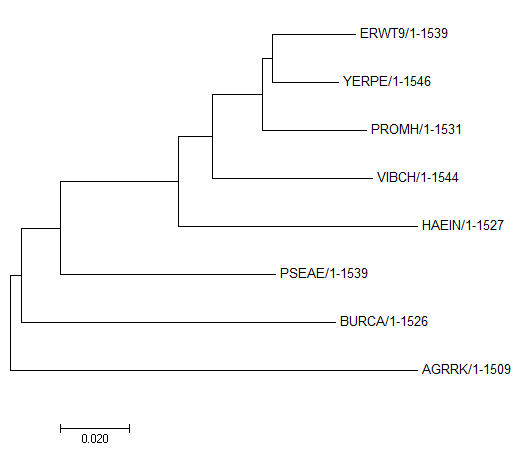
Дерево по белкам:
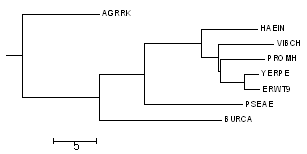
Дерево по РНК и дерево по белкам, совпадают, это говорит о том, что эволюция 16S рРНК и белков шла одинаково в организмах.
Задание 2
Здесь нам потребовалось найти гомологов белка CLPX_ECOLI, во все тех же организмах. Гомологи были выровнены, с помощью полученного выравнивания затем было остроено дерево методом Neighbor Joining.

На рисунке отмечены: Зеленым - дупликация Желтым - видообразование Фиолетовым - группы ортологичных белков Оранжевым - паралоги
