Задание 1
Cсылка на проект в формате .jvpС помощью JalView было построено дерево (Average Distance Using BLOSUM62)
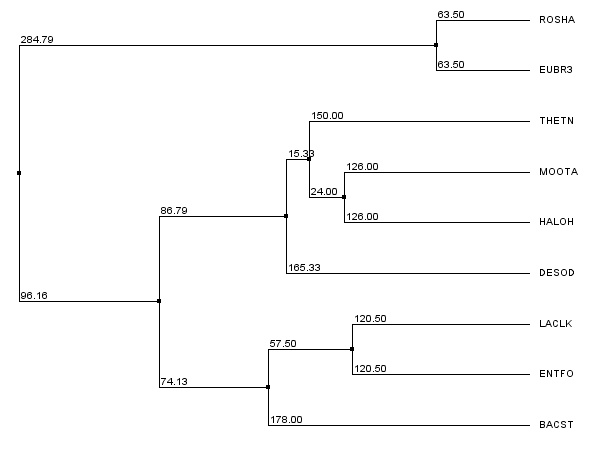
Рисунок1.Дерево
По данному дереву было отсортировано выравнивание
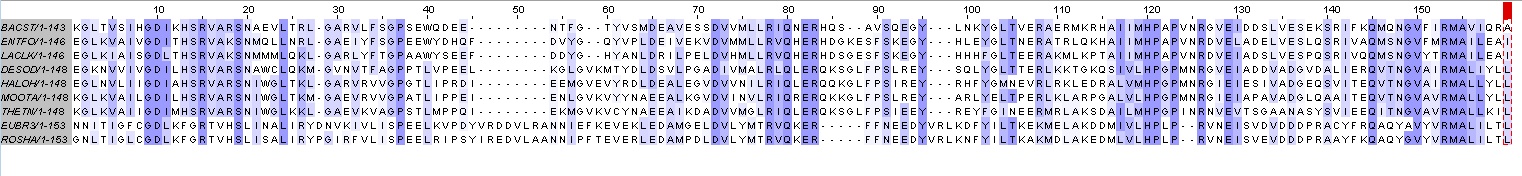
Рисунок2.Выравнивание (окраска BLOSUM62, порог консервативности 30)
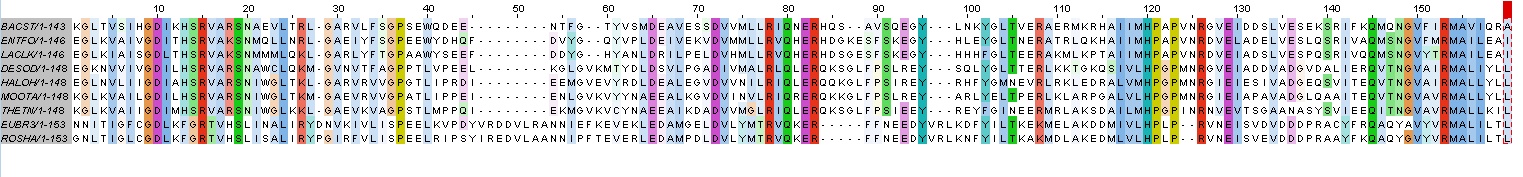
Рисунок3.Выравнивание (окраска ClustalX)
Задание 2
Cамый большой участой выравнивания, не входящий в кластеры и блоки, состоит из 23 позиций
Из них 11 содержат хотя бы 1 гэп, что составляет (11/23)*100% = 47,8%.
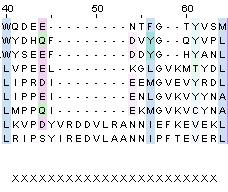
Рисунок 4.Самый большой участок, не входящий в кластеры и блоки
На рисунке 5 представлен кластер, состоящий из 2 блоков и участка из 2 позиций между ними.
Он удовлетворяет критерию: все гэпы имеют длину, равную расстоянию между блоками.
Первый блок состоит из 7 позиций.
Он содержит 6 абсолютно функционально консервативных позиций и 4 абсолютно консервативных позиций,
Второй блок состоит из 5 позиций
Он содержит 1 абсолютно функционально консервативную позицию, 1 функционально консервативную на более чем 70% и 2 абсолютно консервативные позиции

Рисунок 5.Кластер
Две последовательности ROSHA/1-153 и EUBR3/1-153 объединены в одну группу на основе высокого сходства этих последовательностей между собой при отсутствии сходства с другими последовательностями.
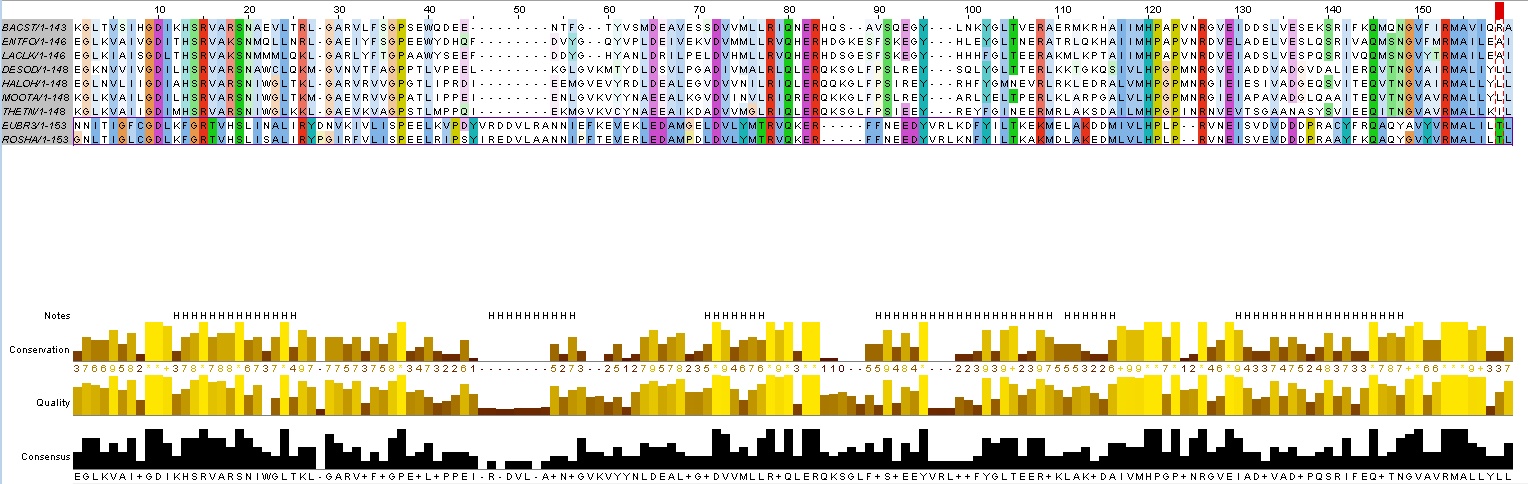
Рисунок 6.Группа
Задание 3
Рассмотрим блок, состоящий из 12 позиций, представленный на рисунке 6 Он содержит
Рисунок 7.Случайно выбранный блок
Задание 4
Добавленная последовательность – sequence_07, CALS8/1-147 была вписана в выравнивание вручную.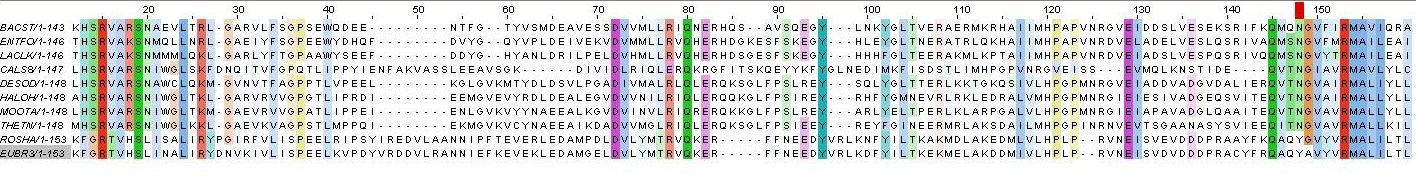
Рисунок 8.В выравнивание вручную вписана последовательность
Задание 5
Далее я добавила в выравнивание заведомо негомологичную последовательность.
Выравнивание получилось неудачным - найдено 5 совпадений в абсолютно функционально консервативных позициях, что составляет 3,125 % от общего числа позиций.

Рисунок 9.В выравнивание добавлена заведомо негомологичная последовательность.
Задание 6
Также я построила выравнивание заведомо негомологичных последовательностей. Для этого я использовала 7 белков из списка белков, с которыми работают мои однокурсники. AC выбранных белков в базе данных Uniprot: B7JW79; K9R0L9 ;Q6L1N7 ;R4T2E1; Q5SIL5 ;O67781; A3PS35. Выравнивание было построено с помощью команды "Muscle with Default". Результат представлен на рисунке. Совпадающих позиций очень мало, так как последовательности негомологичны друг другу. Самые лучшие блоки (те, в которых аминокислотные остатки гомологичны в половине или более последовательностей) представлены отдельно на рисунках
Рисунок 10.Фрагмент выравнивания негомологичных последовательностей. Окраска ClustalX

Рисунок 10.1 Лучший блок №1 (содержит 2 абсолютно функционально консервативные позиции (33,3%))

Рисунок 10.2 Лучший блок №2 (содержит 2 абсолютно функционально консервативные позиции (28,6%)
Выбранные последовательности негомологичны, что четко отражает "выравнивание": консервативных позиций крайне мало, найти блок, включающий в себя все последовательности, не представилось возможным.
Cсылка на проект в формате .jvp