Задание 1 Аннотирование последовательности и сравнение её с аннотацией генов в записи GenBank.
Для выполнения данного задания я взяла контиг unplaced-449 длиной 36412 bp.
Он был предварительно отобран при помощи команды infoseq с опциями -only -name -snucleotide1 -length на сервере kodomo
Последовательность выбранного контига я подала на вход в blastx. С выдачей можно ознакомиться на рисунке 1
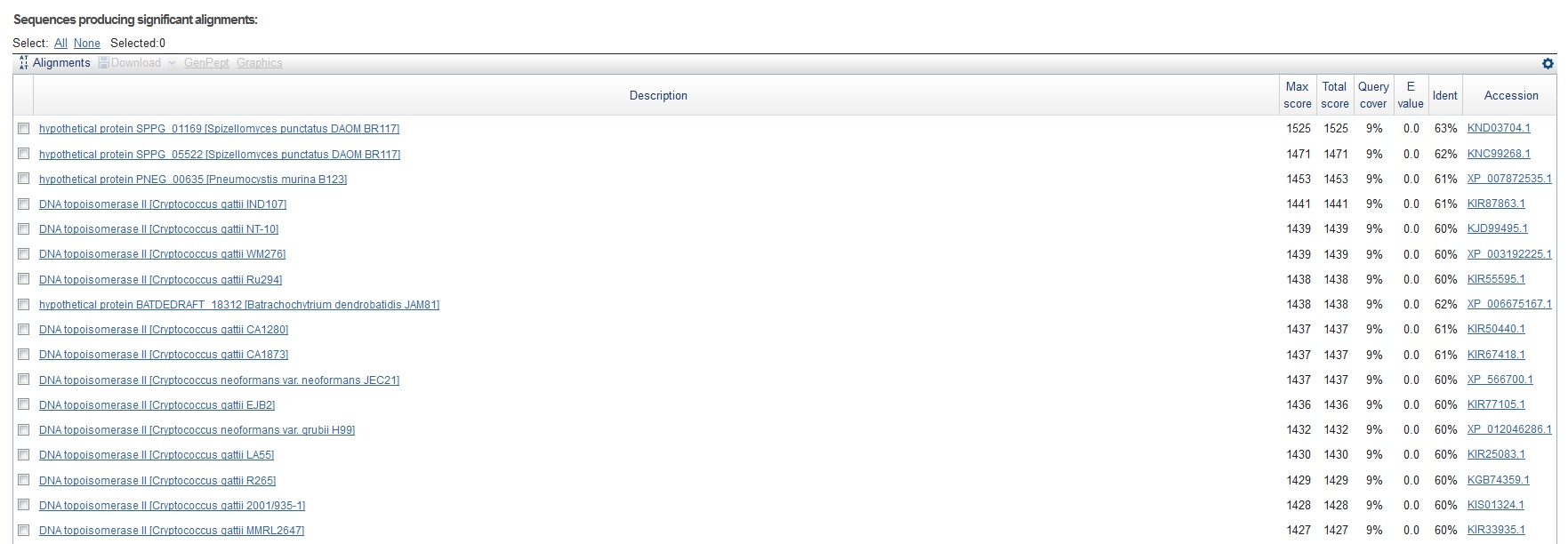
Рисунок 1 Выдача blastx
Далее я перешла на сайт augustus.
Наиболее часто в выдаче blastx фигурировал род Сryptococcus, так что поиск производился по таксону Сryptococcus neoformans
Программа выдала архив, содержащий следующий набор файлов:
Проверку предсказания я осуществила с помощью BLAST. Из файла augustus.aa я взяла аминокислотные последовательности и запустила blastp.
Область поиска ограничена таксоном Fungi
Находки для гена g93.t1
Возможная функция гена g93.t1 - Xaa-Pro aminopeptidase 2
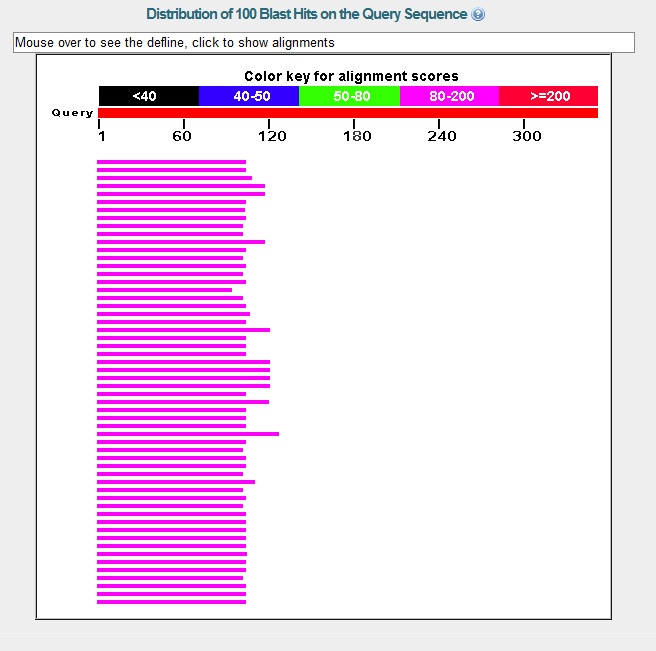
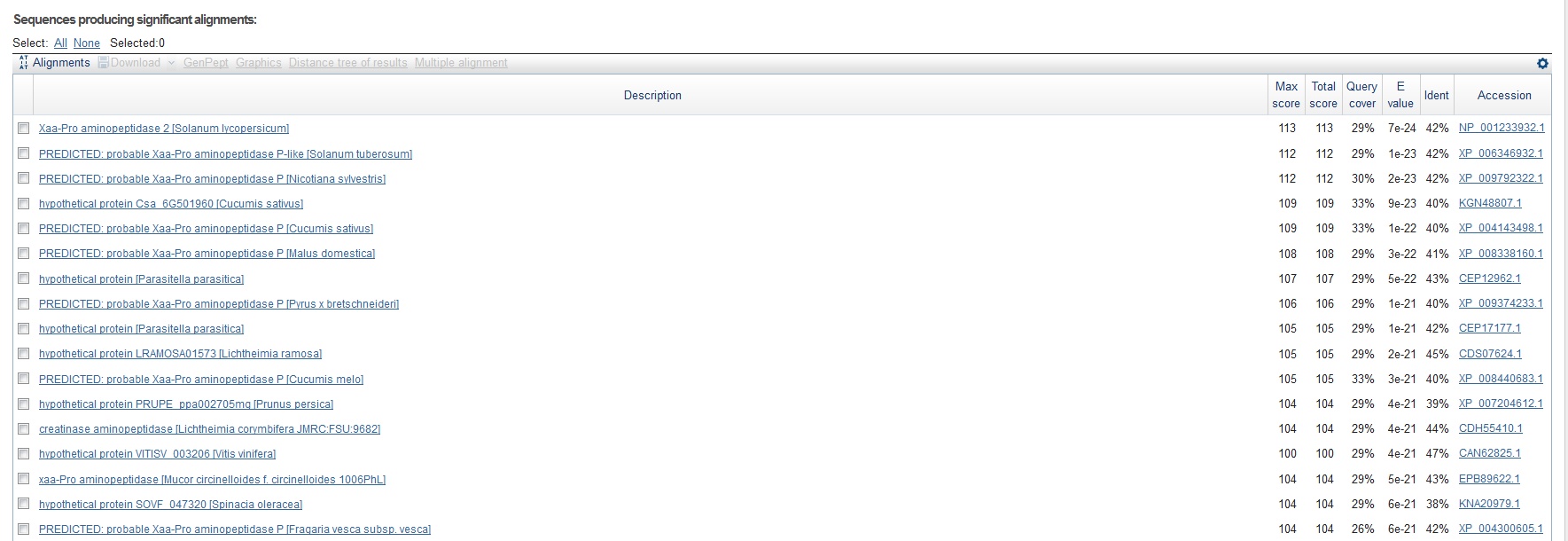
Рисунок 2 Выдача blastp для гена g93.t1
Находки для гена g92.t1
Выдача содержит довольно хорошие находки
Судя по ним, можно сделать вывод, что границы генов и их экзон-интронная структура 8 гена предсказана правильно.
Возможная функция гена - DNA topoisomerase II
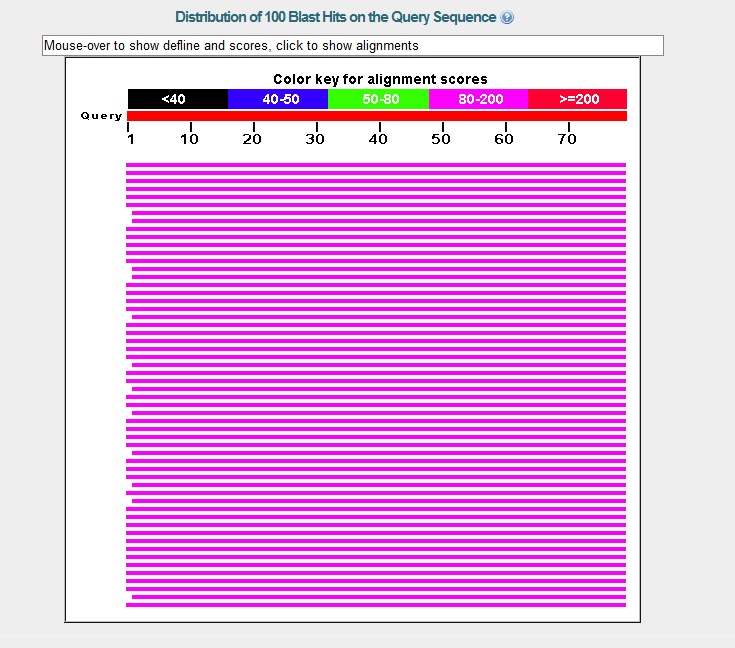
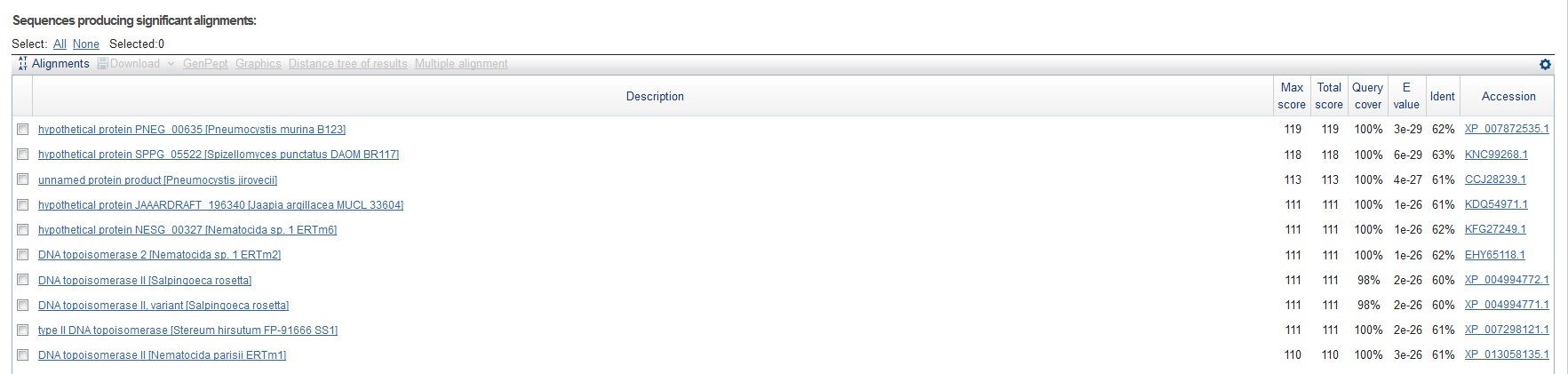
Рисунок 3 Выдача blastp для гена g92.t1
Находки для гена g97.t1
Процент идентичности находок низкий, находок мало, две трети с гипотетическими белками, что говорит о том, что достоверность данной предсказанной находки довольно низкая.
Сделать более подробные выводы об правильности экзон-интронной структуры и функции белка я не могу на основании полученных данных.
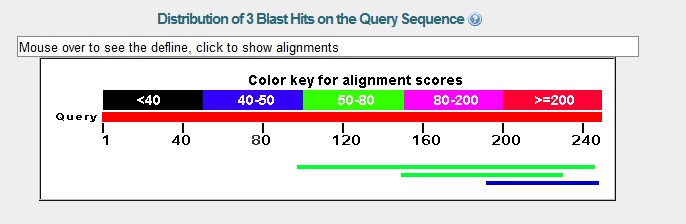
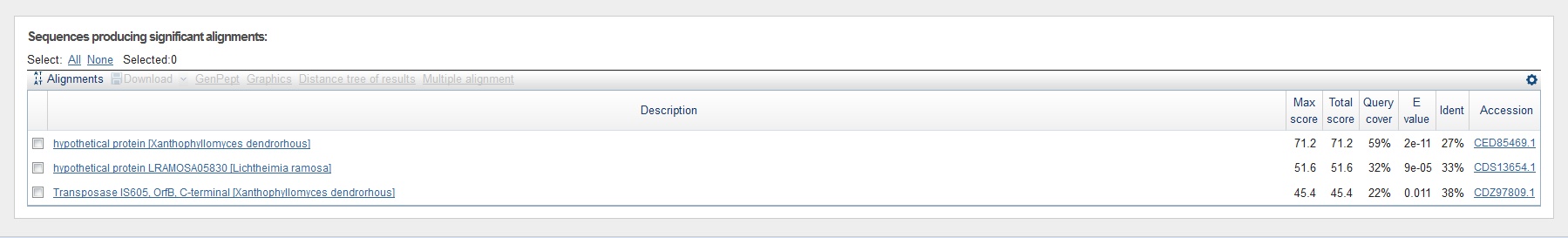
Рисунок 4 Выдача blastp для гена g97.t1
Находки для гена g16.t1
Процент идентичности находок средний, но функцию белка можно предположить (isocitrate dehydrogenase)
Экзон-интронная структура, на мой взгляд, предсказана правильно.
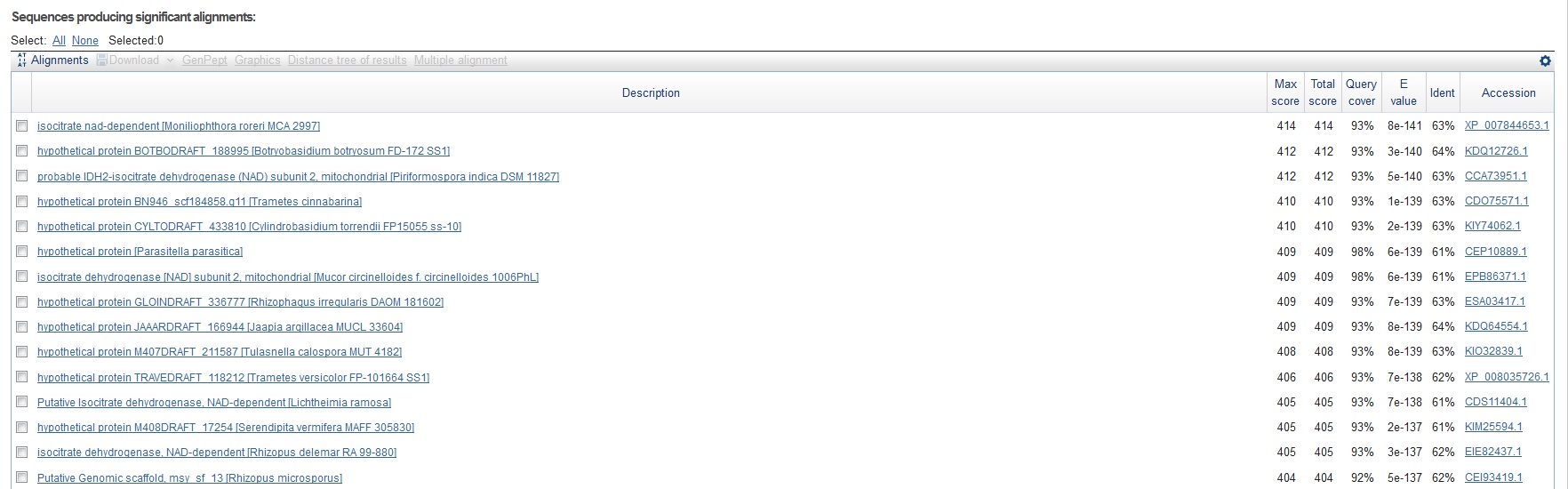
Рисунок 5 Выдача blastp для гена g16.t1
Находки для гена g104.t1
Выдача содержит очень хорошие находки
Процент идетничности довольно высокий. Судя по ним, можно сделать вывод, что границы генов и их экзон-интронная структура 8 гена предсказана правильно.
Возможная функция белка - heat shock protein
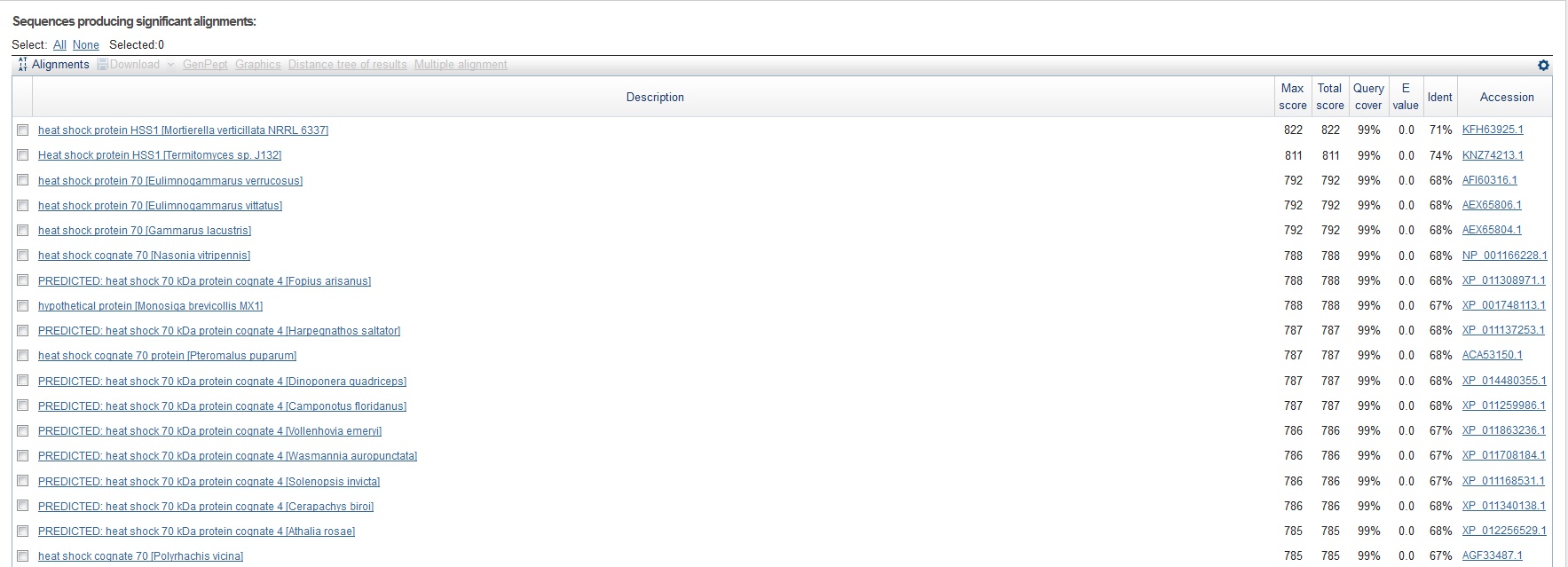
Рисунок 6 Выдача blastp для гена g104.t1
Сравние аннотации Refseq и AUGUSTUS одного гена человека
Для выполнения данного задания я использовала UCSC Genome Browser, в которой выбрала последнюю сборку генома hg38.
Белок - vascular endothelial growth factor A идентификатор - NM_001171630
Хромосома chr6: 43 770 820 - 43 784 135
Были оставлены только три трэка: base position, Refseq и AUGUSTUS. Поиск по двум последним производился отдельно.
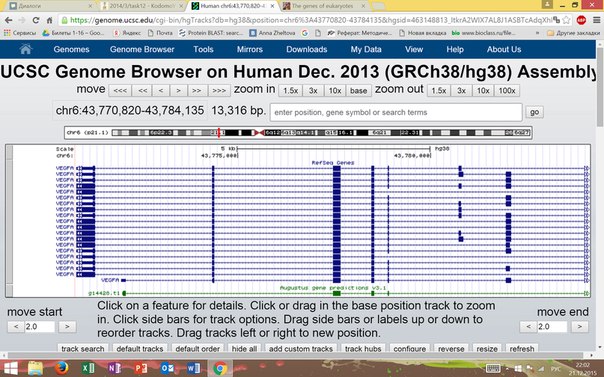
Рисунок 7 Выдача для RefSeq

Рисунок 8 Параметры поиска для Refseq
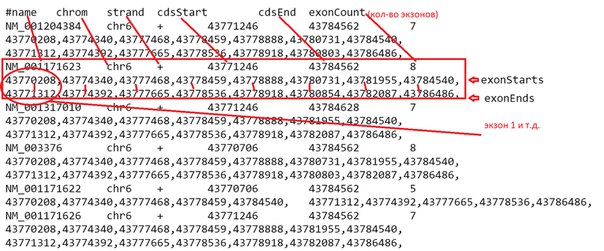
Рисунок 9 Таблица экзонов для Refseq. Все - кодирующие
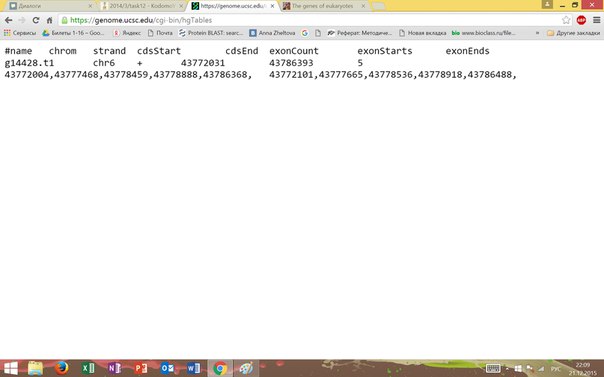
Рисунок 10 Таблица экзонов для Augustus. Все - кодирующие
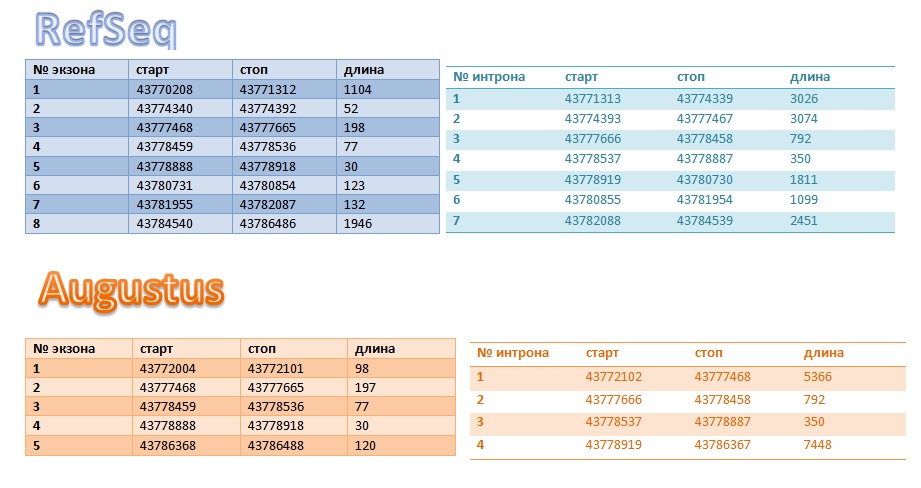
Рисунок 11 Сводная таблица
В аннотации Refseq указаны экзоны, который не определил AUGUSTUS
Аннотации довольно существенно различаются
Ссылки
© Козлова Анастасия, 2015