Задание 1 Ферменты и метаболические пути. База данных KEGG
В данном задании требовалось проверить, являются ли члены разных ортологических рядов KEGG гомологичными белками, и проанализировать их филогенетические отношения.
Для выполнения задания я взяла реакцию (R)-2-Hydroxybutane-1,2,4-tricarboxylate <=> (Z)-But-1-ene-1,2,4-tricarboxylate + H2O из пути биосинтеза лизина (выделена на рисунке синим)
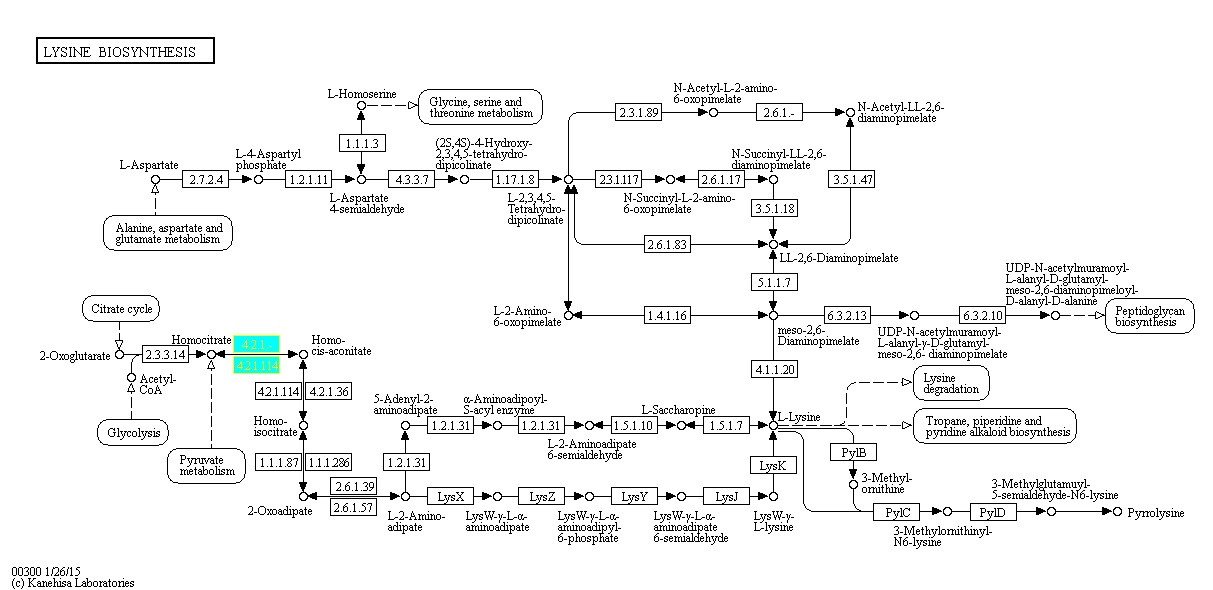
Рис. 1. Метаболическая карта биосинтеза лизина с выделенной реакцией (EC: 42.1.114) (БД KEGG PATHWAY).
Данную реакцию катализируют три ортологических ряда
| Идентификатор в БД KEGG ORTHOLOGY | Количество белков в ряду | Ссылка на запись в БД KEGG ORTHOLOGY |
| K16792 | 94 | KEGG ORTHOLOGY |
| K16793 | 106 | KEGG ORTHOLOGY |
| K17450 | 94 | KEGG ORTHOLOGY |
Затем были послучены последовательности белков с помощью сервиса "Retrieve/ID mapping" из БД Uniprot. Идентификаторы преобразованы следующим образом: ХХХХХХ_ХХХХХ|K*****, где К***** - идентификатор ортологического ряда в БД KEGG ORTHOLOGY.
Полученные последовательности были выровнены с помощью программы Muscle (выравнивание в формате fasta).
Было выполнено множественное выравнивание последовательностей Выравнивание было открыто в JalView (ссылка на проект).
На мой взгляд, гомология наблюдается между ортологическими рядами K17450 и K16792. Для дальнейшей работы пришлось разбить последовательности на группы и сформировать два отдельных выравнивания.
Далее из каждого выравнивания были удалены короткие последовательности. Блок с белками, принадлежащими ортологическому ряду K16793 был плохо выровнян с остальными, и его пришлось удалить.
Дерево разделяется на клады, соответствующие отдельным ортологическим рядам.
У ветви, отделяющей две клады друг от друга бутстрэп-поддержка равна 99% - очевидно, что ветвь достоверна.
Дерево