Задание 1
Для работы я выбрала следующие протеобактерии
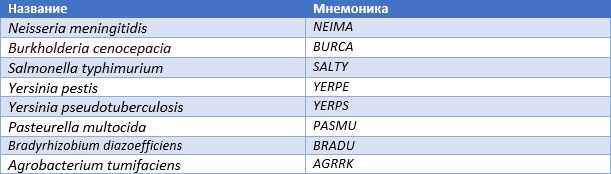
Таблица 1 Выбранные мною протеобактерии
Скобочная формула дерева ((BRADU, AGRRK), ((BURCA, NEIMA), (PASMU, (SALTY, (YERPE, YERPS)))));
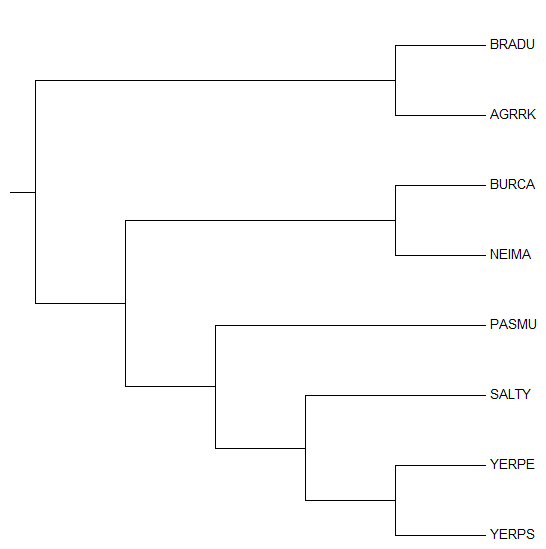
Рисунок 1 Дерево, построенное программой MEGA
Дерево содержит 5 нетривиальных ветвей
Реконструкция филогении
Данные получены с помощью таксономического сервиса NCBI
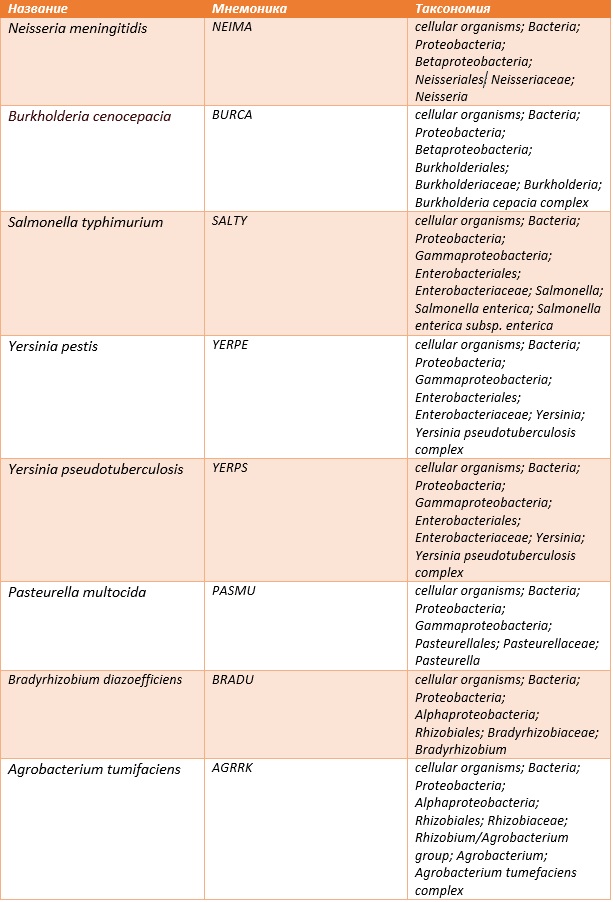
Таблица 2 Таксономия выбранных протеобактерий
Список нетривиальных ветвей, выделяющих некоторые таксоны:
Укоренение и бутстрэп
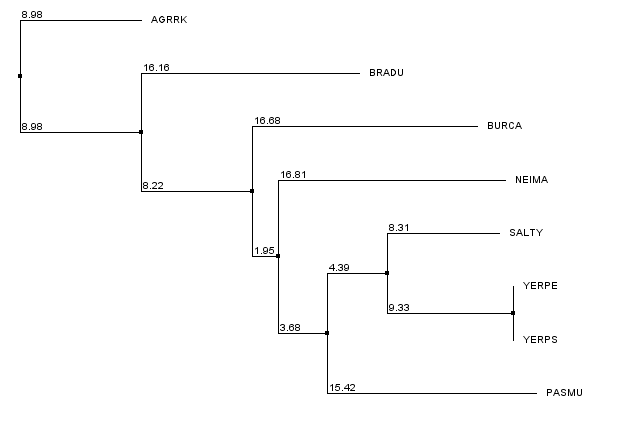
Рисунок 3 Филогенетическое дерево выбранных организмов, реконструированное по последовательностям белка пептидил-тРНК гидролазы методом "Neighbor Joining Using % Identity" в программе JalView.
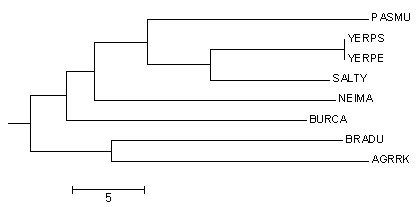
Рисунок 4 Филогенетическое дерево выбранных организмов, реконструированное методом "Neighbor Joining Using % Identity" и укорененное в среднюю точку с помощью программы retree пакета PHYLIP.