Задание 2 Сравнение списков находок, полученных 3-я разными алгоритмами blast: blastn, megablast и discontiguous megablast.
Для выполнения задания возьмем последовательность из практикума 7
Требуется зависимости от находок blastn ограничить область поиска подходящим таксоном, в моем случае таксоном Loxosomella
Итак, я запустила поиск по данной последовательности и с данным ограничением в blastn, megablast и discontiguous megablast.
Оценим результаты
Рисунок 1 Результаты поиска в blastn
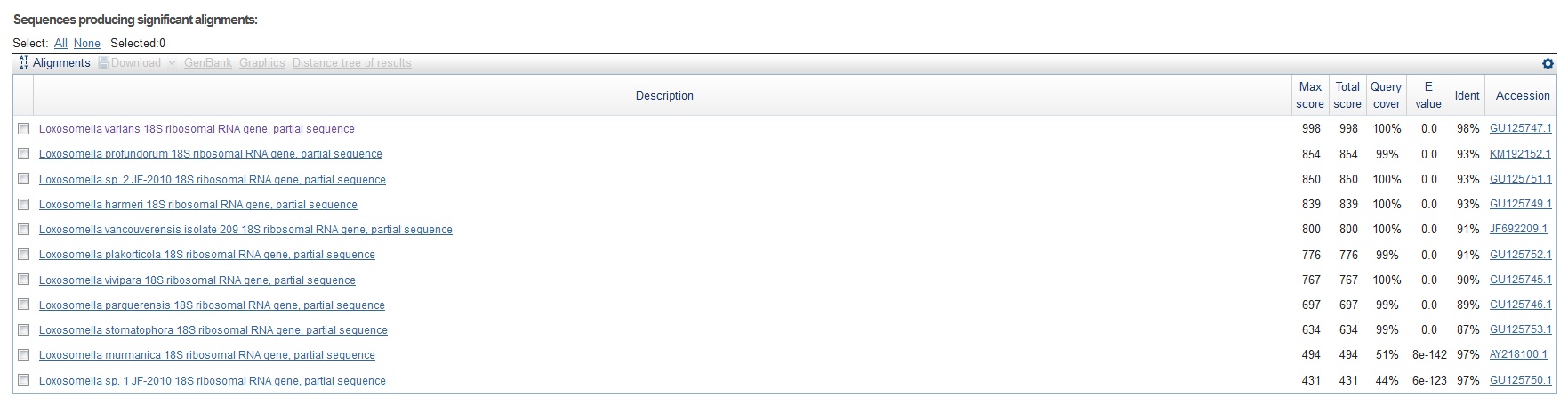
Рисунок 2 Результаты поиска в megablast
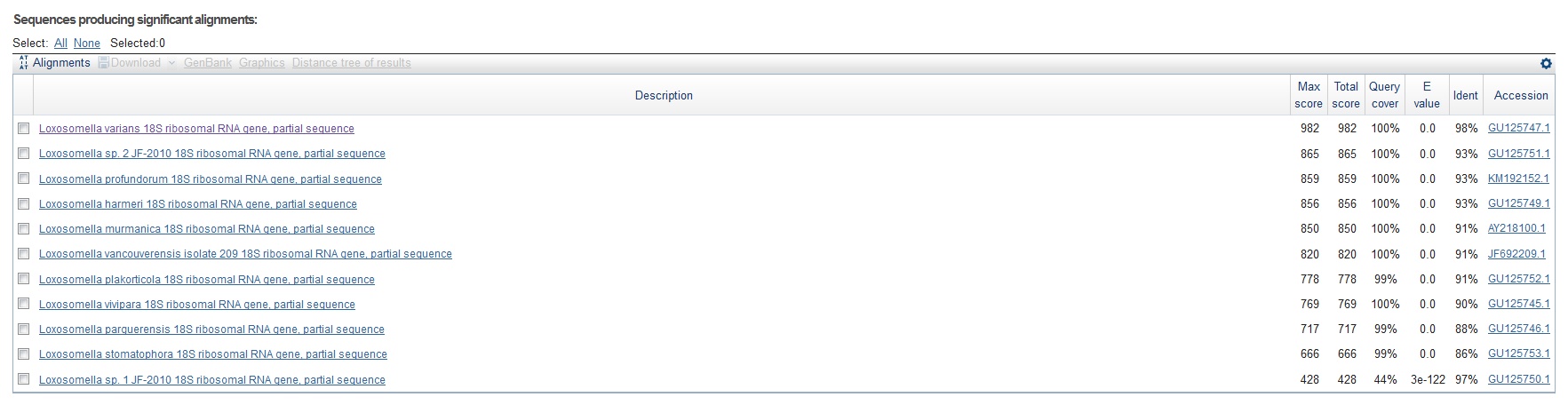
Рисунок 3 Результаты поиска в discontiguous megablast

Рисунок 4 Таблица
Blastn нашел 10 последовательностей с E-value 0.0 и Query cover >75%. Три оставшиеся находки ввиду низкого значения Qury cover интереса не представляют
Megablast выдал 9 последовательностей, отвечающих подобным требованиям, Discontiguous megablast - 10.
Из таблицы видно, что megablast и discontiguous megablast отрезают находки со слишком высоким E-value, при этом порог для megablast значительно выше
Задание 3 Проверка наличия гомологов пяти белков в геноме одного из организмов.
Требовалось взять организм из задания 1 практикума 7 - Plasmodium falciparum - и проверить наличие у него гомологов следующих пяти белков: HSP7C_HUMAN, TERT_HUMAN, CISY_HUMAN, RPB1_HUMAN, PABP2_HUMAN.
Для выполнения задачи использовался tblastn, то есть поиск белка в базе транслированных в 6-ти рамках нуклеотидных последовательностей. Область посика была ограничена организмом Plasmodium falciparum
Вначале для каждого белка была найдена запись в Uniprot, сохранены их AC и fasta-последовательности. Затем проведен поиск с помощью tblastn. На вход blast подавался AC белка.
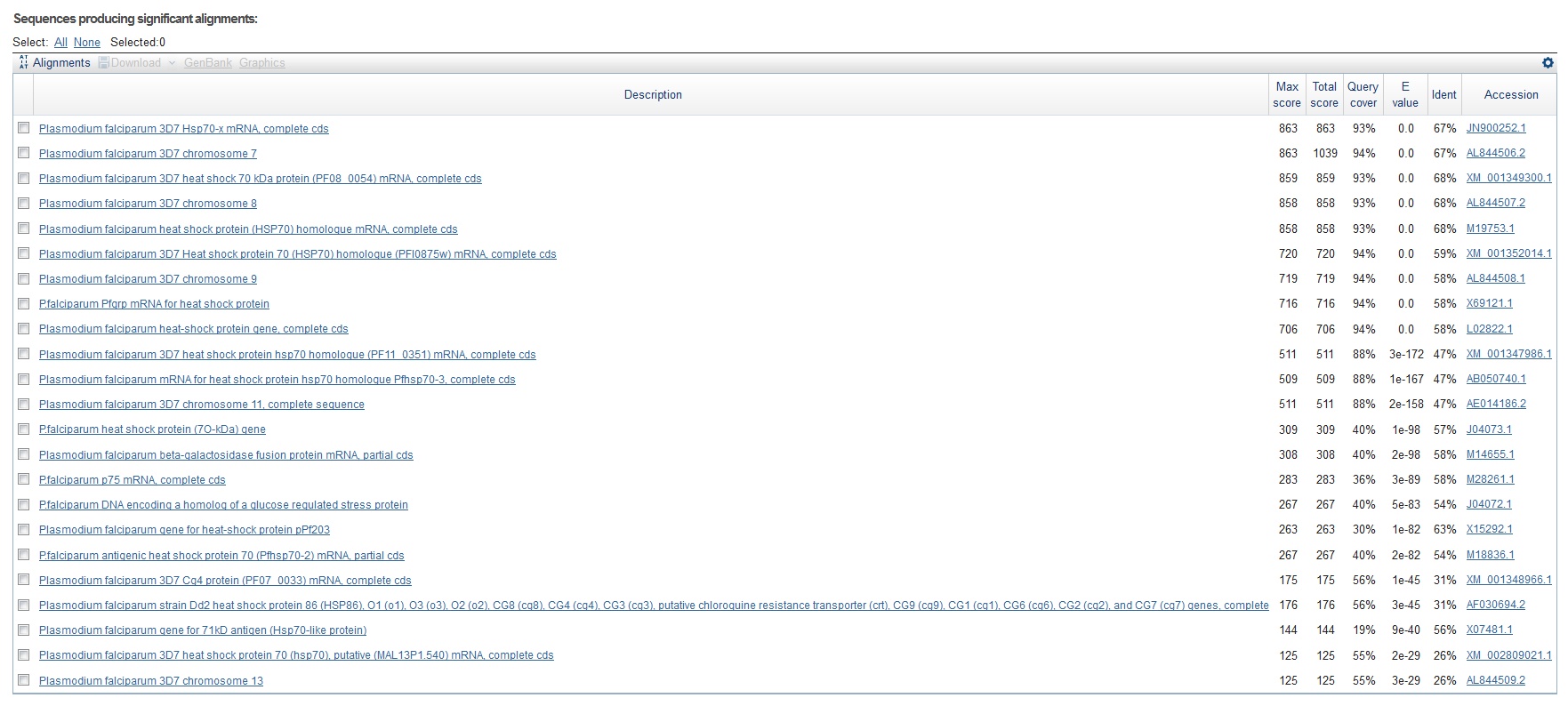
Рисунок 5 Находки из Plasmodium falciparum по запросу P11142
По данному запросу выдано 23 находки, из которых хорошими можно считать 12 (с хорошими показатели E-value и Query cover)
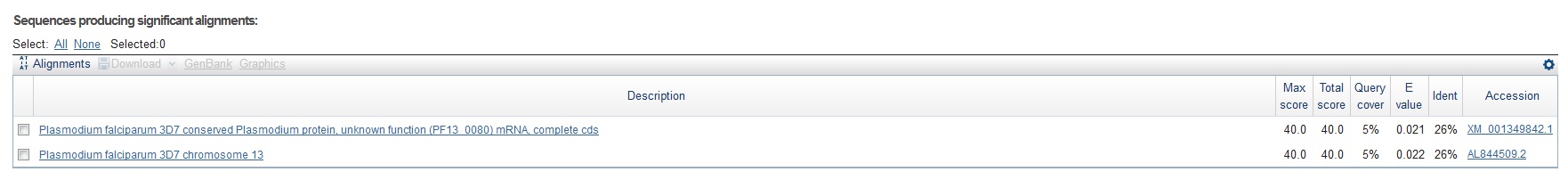
Рисунок 6 Находки из Plasmodium falciparum по запросу O14746
По данному запросу обнаружено всего 2 находки, каждая из которых имеет низкий показатель Query cover
На мой взгляд, о наличии гомологии ничего не свидетельствует.
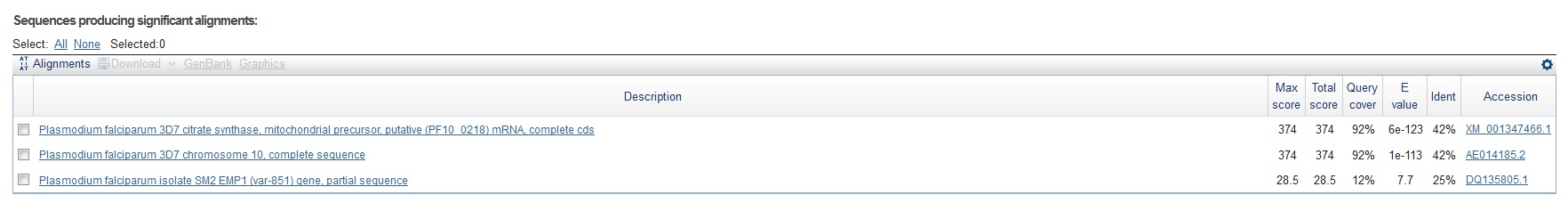
Рисунок 7 Находки из Plasmodium falciparum по запросу O75390
Найдено три записи, из которых 2 являются хорошими.
Исходя из названия, первая находка - гомолог нашего белка.
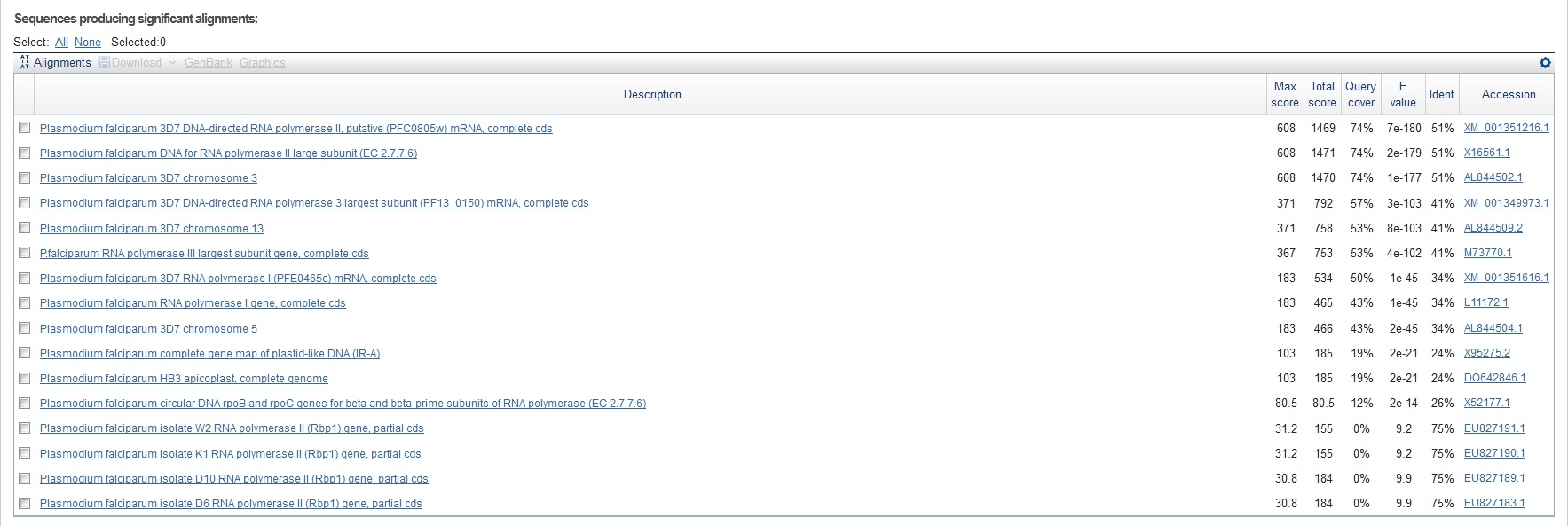
Рисунок 8 Находки из Plasmodium falciparum по запросу P24928
Хороших находок 3. Гомологами являются те, которые кодируют субъединицы РНК-полимераз 1,2 и 3, так как РНК-полимеразы всех трех типов похожи, имеют приблизительно одинаковые задачи и механизмы работы и явно гомологичны
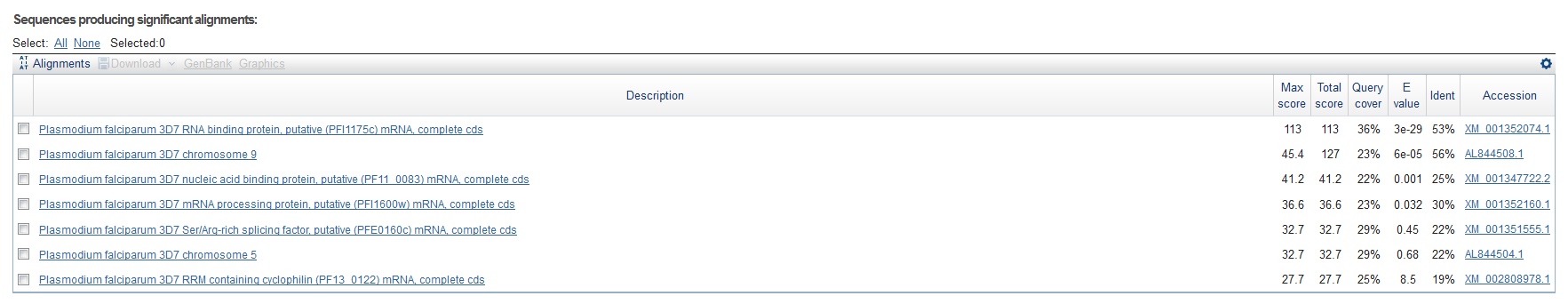
Рисунок 9 Находки из Plasmodium falciparum по запросу Q86U42
Всего 7 находок, среди которых особо хороших нет ввиду очень низкого показателя Query cover. Судя по названию, первая находка может являться гомологом нашего белка
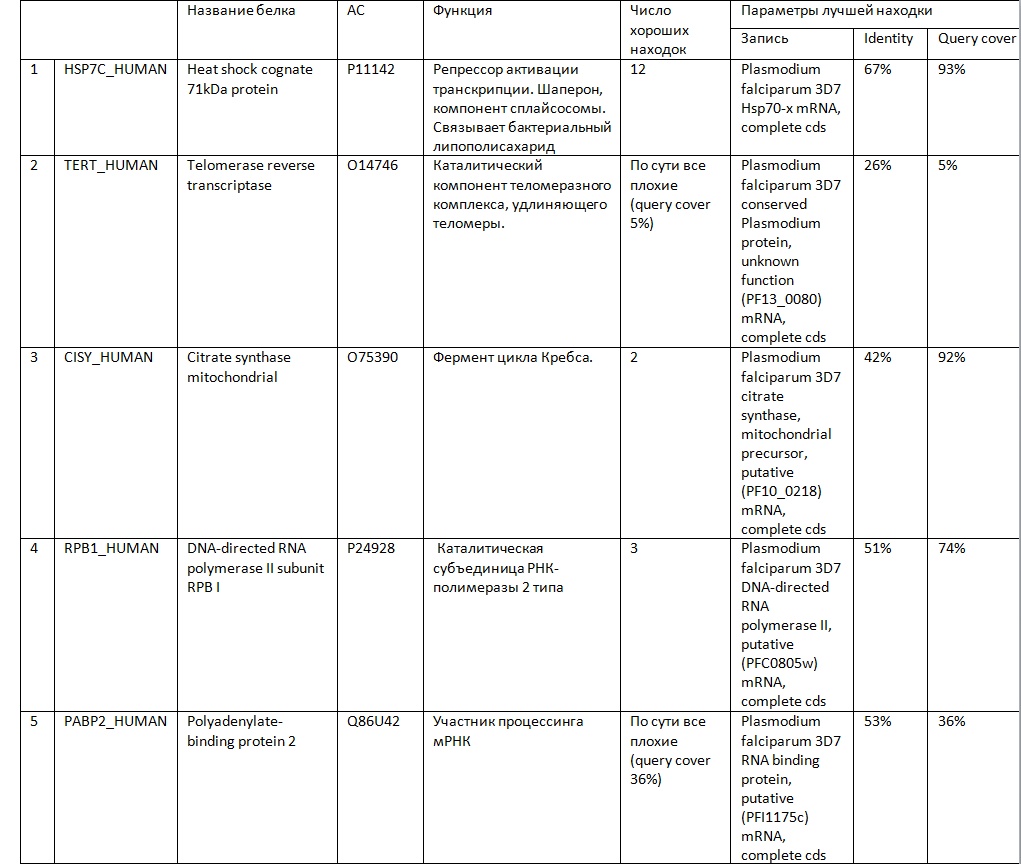
Рисунок 10 Таблица
Задание 4 Оценка сходства геномов вирусов.
Для выполнения задания я выбрала вирус пятнистости орхидей Orchid fleck virus, с которым мне довелось работать в первом семестре.
Далее были сохранены геномы данного вируса Orchid fleck virus (Идентификатор в INSDC AB244417), а также 5 родственных ему, а именно:
Все эти вирусы - двухцепочечные РНК-содержащие. Я брала для сравнения только одну цепь РНК (RNA1 segment)
При помощи команды seqret я объединила файлы с геномами вирусов в один файл viruses.fasta
Далее я сделала базу индексов blast для созданного fasta файла: makeblastdb -in viruses.fasta -dbtype nucl
Потом запустила tblastx, подав на вход тот же самый fasta файл: tblastx -query viruses.fasta -db viruses.fasta -out blast.out -outfmt 7. В итоге таблица с результатами была записана в файл blast.out.
С помощью python-скрипта были удалены неинформативные и слабо сходные находки из полученной таблицы. Были установлены параметры для значение E-value, score и т.д

Рисунок 11 Создание итоговой таблицы
Далее была произведена сортировка по убыванию значения identity. Оказалось,что первые 17 находок с наибольшим процентом идентичных нуклеотидов представляют собой сходные участки двух одних и тех же вирусов: AY618417 и NC_011542,
(Maize fine streak virus и Iranian maize mosaic nucleorhabdovirus), что позволяет предположить, что именно геномы этих двух вирусов наиболее схожи
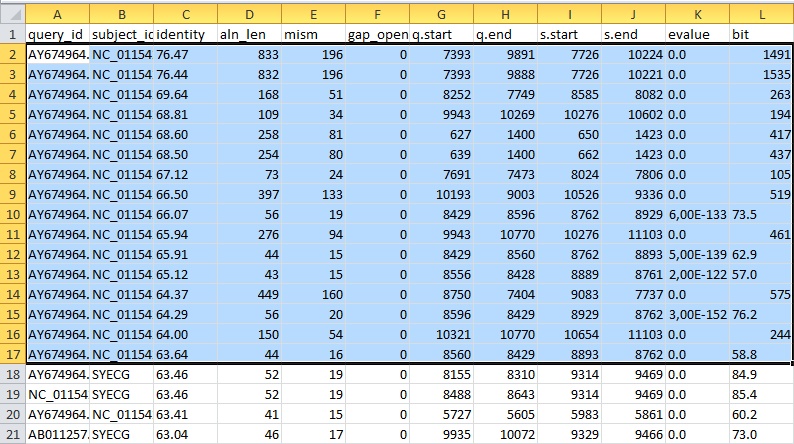
Рисунок 12 Сортировка по убыванию значения identity
Далее сортировки по длине выравнивания, его счёту и E-value показали, что лучшие находки по этим параметрам также представляют собой выравнивания данных вирусов.
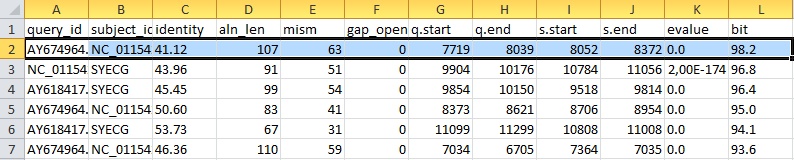
Рисунок 13 Сортировка по убыванию значения bit score
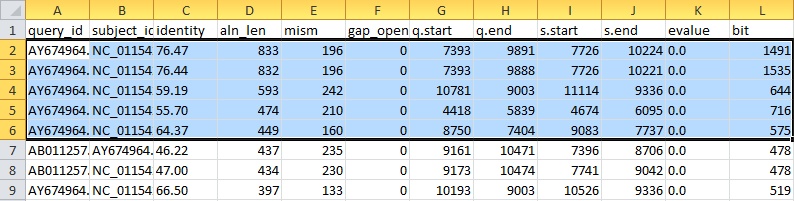
Рисунок 14 Сортировка по убыванию значения aln_len
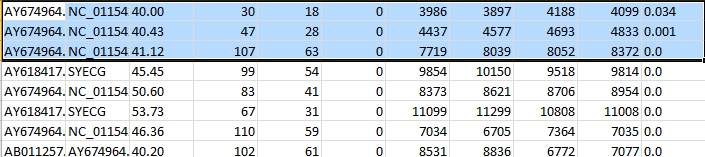
Рисунок 15 Сортировка по возрастанию значения evalue (правая колонка таблицы)
Таким образом, если оценивать сходство двух геномов вирусов по максимальной длине, максимальному скору, максимальному проценту идентичности и минимальному E-value находок, тогда можно признать, что из данных вирусов наиболее сходные геномы имеют Maize fine streak virus и Iranian maize mosaic nucleorhabdovirus Ссылки
© Козлова Анастасия, 2015