
|
Учебный сайт Алены Корягиной |

|
Главная |
Oбо мне |
Cеместры |
Cсылки |
PSI-BLAST. Построение семейства гомологов белка
Алгоритм PSI-BLAST (Position-Specific Iterated BLAST) на сайте NCBI предназначен для поиска удаленных гомологов белков и строится на основе уже найденных белков в отличии от BLASTP, который, как упоминалось ранее, находит все последовательности белков сходные с входной последовательностью.
Для освоения работы с алгоритмом PSI-BLAST случайным образом был выбран белок с АС: Q9QCL4.
Ниже представлена некоторая информация по выбранному белку, полученная с помощью базы данных Uniprot:
1) Идентификатор белка (ID) — Q9QCL4_BDV;
2) Белок состоит из 63 аминокислотных остатков;
3) Белок экспертом не проверен, относится к разделу UniProtKB/TrEMBL;
4) Уровень доказательства существования белка — 4: Predicted;
5) Белок получен из вируса, вызывающего болезнь Борна (Borna disease virus [BDV]).
Данный вирус классифицируется как ssRNA negative-strand viruses, т.е. носителем информации является
одноцепочечная РНК отрицательной полярности, вирус относится к отряду Mononegavirales, семейству Bornaviridae и роду Bornavirus.
Далее был проведен поиск удаленных гомологов с помощью вышеуказанного алгоритма по базе данных Refseq, но результатом стал всего один белок с идентификатором YP_009031.1 и E-value 4e-34, следовательно, будем осуществлять поиск по базе данных nr. Результатом первой итерации стало 55 последовательностей, E-value которых меньше 0.005. Все найденные последовательности белков получены из вирусов семейства Bornaviridae. После второй итеррации добавился еще один белок с идентификатором BAM72615.1 и E-value 0.002. После третьей итерации новых последовательностей не добавилось, следовательно, можно считать, что семейство гомологов выбранного белка сформировано. В таблице 1 вы можете найти информацию о худших и лучших находках каждой итерации.
Таблица 1. Протокол итераций PSI-BLAST для белка с АС: Q9QCL4
| Номер итерации | Число находок выше порога (0,005) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
| 1 | 55 | ADU05396.1 | 0.003 | CAB52091.1 | 6e-38 |
| 2 | 56 | BAM72615.1 | 0.002 | CAB52091.1 | 8e-30 |
| 3 | 56 | BAM72615.1 | 7e-09 | CAC70648.1 | 2e-25 |
Следующим действием было построено множественное выравнивание последовательностей созданного семейства гомологов (рис.1.). Вы можете скачать выравнивание в формате fasta. Полученное выравнивание очень консервативное, сильно консервативные широкие блоки расположены на концах последовательностей, а в центрах консервативные блоки уже и их меньше. Посмотрим на позиции с 7 по 12 и с 14 по 18, если не рассматривать последние три последовательности, то почти все вышеуказанные позиции будут абсолютно консервативными. Можно сделать вывод, что, возможно, выбранные блоки являются важными для белка и их отсутствие может означать потерю каких-либо функций белка. Так же стоит обратить внимание на длины последовательностей: в основном длины последовательностей варьируются от 63 до 89 аминокислотных остатков, но есть последовательность длиной 95 а.о. (p10-FLAG, АС: AAC09175) и последовательность длиной 217 а.о. (OFR1, АС: 1702430A). Было решено проверить, не содержит ли C-концевой участок этой аномально длинной последовательности какого-то белкового домена с известной функцией. С помощью базы данных Pfam предположение было подтверждено: белок OFR1 включает в себя два домена BDV_p10 и BDV_p24, первый из которых соответствует исходной последовательности. Для построенного выравнивания в Jalveiw была выбрана раскраска BLOSUM62 и консервативность >70%. Проект в формате jar вы можете скачать здесь.
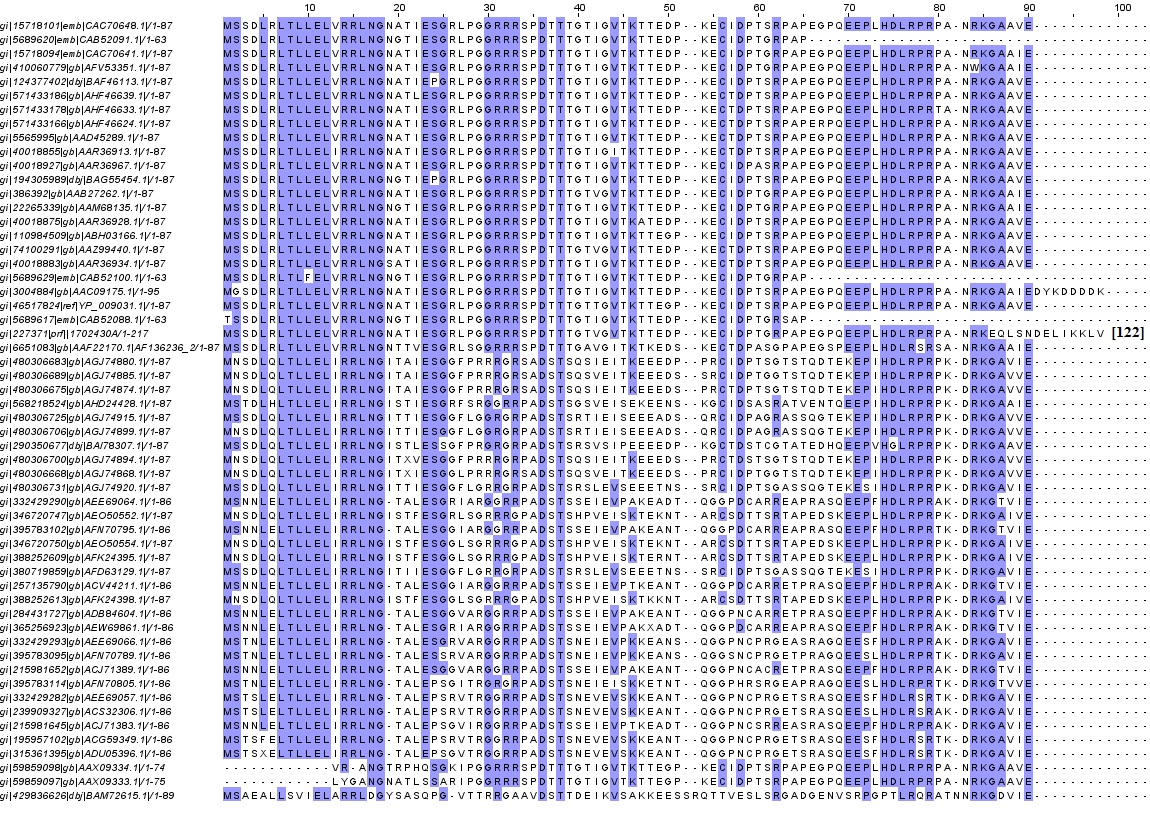
|
Рис.1. Множественное выравнивание последовательностей семейства гомологов для белка с АС: Q9QCL4. Рисунок получен с помощью Jalview. |
Дата последнего изменения: 10.05.2014