
|
Учебный сайт Алены Корягиной |

|
Главная |
Oбо мне |
Cеместры |
Cсылки |
Работа со структурами нуклеиновых кислот с помощью средств программы Jmol
Благодаря возможностям программы Jmol были выделены следующие элементы структуры А-формы дуплекса ДНК:
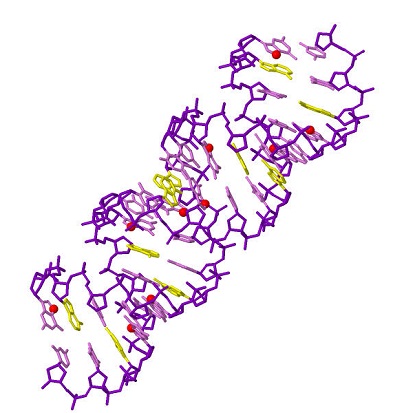
- сахарофосфатный остов ДНК (рис.1);
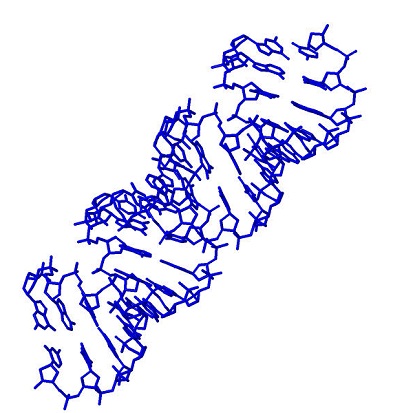
- все нуклеотиды (рис.2);
- все аденины (рис.1);
- атом N7 во всех гуанинах последовательности (рис.1).
|
|
Рис.1. Структура А-формы дуплекса ДНК. Сахарофосфатный остов выделен фиолетовым цветом, светло-розовым выделены азотистые основания, кроме аденина, он выделен желтым, красным — все атомы N7 из всех гуанинов. Рисунок получен с помощью программы Jmol. |
Рис.2. Структура А-формы дуплекса ДНК. Структура А-формы дуплекса ДНК.
Все нуклеотиды выделены синим цветом. Рисунок получен с помощью программы Jmol. |
Вы можете в точности воспроизвести данные визуализации. Для этого необходимо скачать файл .pdb со структурой А-формы дуплекса ДНК. После открыть Jmol, и запустить один из скриптов: скрипт №1 для визуализации первого рисунка, скрипт №2 — для второго.
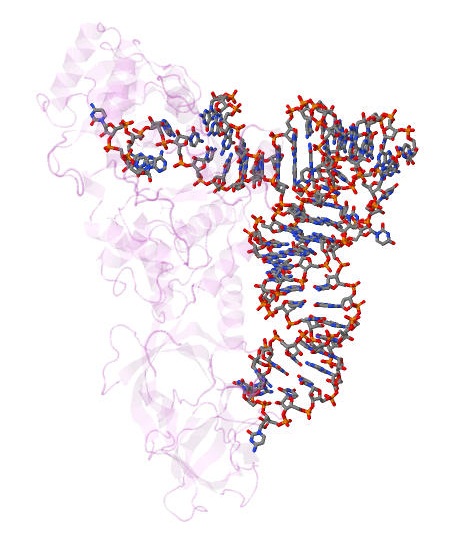
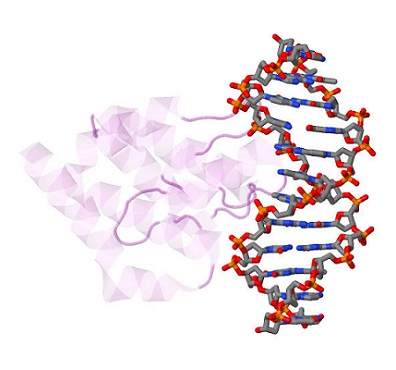
Далее структуры фермента глутаминил-тРНК-синтетазы в комплексе с тРНК (идентификатор записи в базе данных PDB — 1QTQ) и белка SEQA в комплексе с полуметилированной ДНК (1LRR) были исследованы на наличие разрывов в структурах НК. Таковых не было обнаружено (рис.3 и 4). Также были сохранены координаты атомов только ДНК и РНК.
|
|
Рис.3. Глутаминил-тРНК-синтетаза в комплексе с тРНК (идентификатор записи в базе данных PDB — 1QTQ). Раскраска атомов в структуре тРНК соответствует их химической природе: атомы углерода серые, азота голубые, кислорода красные, фосфора оранжевые. Рисунок получен с помощью программы Jmol. |
Рис.4. Белок SEQA в комплексе с полуметилированной ДНК (идентификатор записи в базе данных PDB — 1LRR). Раскраска атомов в структуре ДНК соответствует их химической природе: атомы углерода серые, азота голубые, кислорода красные, фосфора оранжевые. Рисунок получен с помощью программы Jmol. |
Для воспроизведения этих визуализаций с сайта PDB скачайте файл «PDB File (Text)» для структуры с идентификатором 1QTQ и файл «Biological Assembly 1 (gz)» для структуры с идентификатором 1LRR, затем запустите в программе Jmol скрипт №1 и скрипт №2, соответственно, для каждой структуры.
Смотрите также другие работы:
Дата последнего изменения: 23.09.2014