
|
Учебный сайт Алены Корягиной |

|
Главная |
Oбо мне |
Cеместры |
Cсылки |
Определение параметров структур нуклеиновых кислот с помощью программ пакета 3DNA
Как уже говорилось, 3DNA — пакет программ для анализа и простейшего моделирования структур нуклеиновых кислот (НК). По мимо прочих в пакет входит программа fiber, которую мы использовали для моделирования А-, В- и Z-форм дуплекса ДНК (см. здесь), а также программы find_pair и analyze, которые применяются для анализа параметров структуры НК.
Далее мы работали с файлами, полученными здесь, и содержащими последовательности только ДНК и тРНК. Поскольку данные программы работуют только со старым форматом PDB, то для начала эти файлы были переведены в нужных формат, для этого были введены следующие команды:
remediator --old ''1LRR_dna.pdb'' > ''1LRR_dna_old.pdb''
remediator --old ''1QTQ_rna.pdb'' > ''1QTQ_rna_old.pdb''
Теперь, используя полученные файлы, были запущены программы find_pair и analyze. Были введены следующие команды:
export PATH=${PATH}:/home/preps/golovin/progs/X3DNA/bin
export X3DNA=/home/preps/golovin/progs/X3DNA
find_pair -t 1LRR_dna_old.pdb stdout | analyze
find_pair -t 1QTQ_rna_old.pdb stdout | analyze
В результате работы данных программ с файлом, содержащим ДНК, возникли проблемы, так как данная структура ДНК содержит нетипичное азотистое основание метилированный по N3 аденин. Для устранения этой проблемы вручную в файле 1LRR_dna_old.pdb были заменены а.о. с названием 6МА на просто А. После произведенной замены программы снова были запущены. В результате мы получили ряд файлов с описанием разных параметров структур ДНК и тРНК, среди которых есть файлы с форматом .out. В этих файлах содержиться информация о водородных связях, значениях всех торсионных углов, ширине малой и большой бороздки.
Сначала были проанализированны только значения торсионных углов в ДНК и тРНК. Далее с помощью Excel было рассчитанано среднее значение каждого из торсионных углов, причем в расчете не учитывались углы из краевых нуклеотидов (см. табл.1).
Таблица 2.Средние значения торсионных углов в ДНК и тРНК
| α (P - O5') | β (O5' - C5') | γ (C5' - C4') | δ (C4' - C3') | ε (C3' - O3') | ζ (O3' - P) | χ (C1' - N) | |
| ДНК | ±51,04° | ±160,47° | ±44,38° | ±139,98° | ±153,21° | ±121,83° | ±104,73° |
| тРНК | ±81,16° | ±168,98° | ±71,35° | ±89,58;° | ±150,05° | ±72,8° | ±159,11° |
* Средниe значения расчитывались без учета знака угла.
Также были найдены самые "деформированные" нуклеотиды, у которых значения одного или нескольких торсионных углов сильно отклоняется от среднего. Таковыми являются 2T из ДНК и 15U из тРНК(см.табл.2).
Таблица 2.Значения торсионных углов у самых "деформированных" нуклеотидов из ДНК и тРНК
| α (P - O5') | β (O5' - C5') | γ (C5' - C4') | δ (C4' - C3') | ε (C3' - O3') | ζ (O3' - P) | χ (C1' - N) | |
| ДНК | -64,1° | 170,8° | 34,0° | 139,5° | -95,9° | -177,8° | -99,4° |
| тРНК (15U) | 158,2° | -155,3° | 172,9° | 84,7° | -123,6° | -70,2° | 174,2° |
Дальше из файла формата .out была рассмотрена информация о водородных связях
в структуре тРНК.
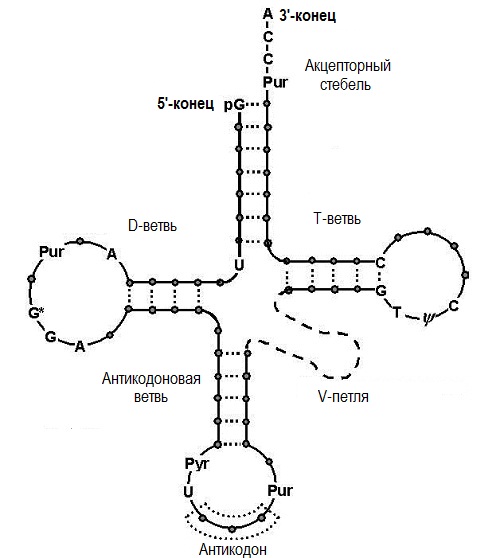
Для того чтобы понимать, как устроена данная структура рассмотрим схему строения вторичной структуры тРНК (рис.1).
Транспортная рибонуклеиновая кислота представляет собой однуцепочечную молекулу РНК, которая во вторичной структуре
имеет вид «клеверного листа». «Клеверный лист» состоит из 3(4) петлей и 4 стеблей. Стебель с петлей формирует ветвь.
В структуре тРНК различают следующие элементы:
- акцепторный стебель;
- D-ветвь, в состав которой входят D-стебель и D-петля. В петлю входит необычный нуклеозид дигидроуридин (D);
- T-ветвь, в состав петли которой входит необычный нуклеозид тимидин (T);
- антикодоновая ветвь, которая содержит антикодоновый триплет в составе своей петли;
Теперь взглянем на информацию, которую мы имеем, о водородных связях в рассматриваемой структуре тРНК. По порядку соединения нуклеотидов мы можем установить, какие пары нуклеотидов относятся к каждому стеблю (см.рис.2).
|

|
Рис.1. Схема строения вторичной структуры тРНК. Рисунок получен из интернета. |
Рис.1. Информация о водородных связях в тРНК. Голубым цветом выделены пары нуклеотидов, относящиеся к акцепторному стеблю, фиолетовым — к Т-стеблю, зеленым — к антикодоновому стеблю и желтым — к D-стеблю. |
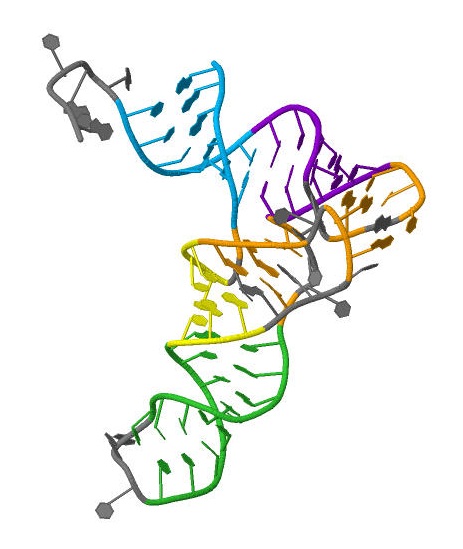
Таким образом, получаем, что в структуре рассматриваемой тРНК акцепторный стебель состоит из 6 пар нуклеодидов (номера нуклеотидов 902-907 и 966-971), Т-стебель из 5 пар нуклеотидов (номера нуклеотидов 949-953 и 961-965) антикодоновый стебель из 8 пар нуклеотидов (номера нуклеотидов 937-944 и 926-933), а D-стебль из 3 пар нуклеотидов (номера нуклеотидов 910-912 и 923-925). Заметим, что еще остались комплементарные пары, которые не относятся к стеблям (на рис.2 они не выделены цветом). Эти пары (954-958, 955-918, 913-945, 914-908, 915-948, 919-956) стабилизируют третичную стуктуру тРНК. На рисунке 3 представлена структура рассматриваемой тРНК и оранжевым цветом на нем выделены именно эти пары.
Также мы можем определить неканонические пары в структуре тРНК: они обозначены «*» на рисунке 2. А именно таковыми являются: 12 U-A, 13 U-G, 14 A-U, 15 U-U, 17 C-G, 21 C-A, 25 A-A, 26 A-U, 27 G-C. Всего в стуктуре присутствует 9 неканонических пар, среди которых есть пары некоплементарные и пары с неканоническими водородными связями.
|
Рис.3. Структура тРНК. Голубым цветом выделены пары нуклеотидов, относящиеся к акцепторному стеблю, фиолетовым — к Т-стеблю, зеленым — к антикодоновому стеблю, желтым — к D-стеблю. Оранжевым обозначены пары нуклеотидов, которые стабилизируют третичную структуру тРНК.Рисунок получен с помощью программы Jmol.
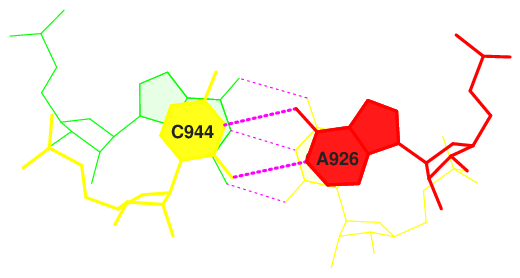
И последняя информация, которую мы рассматривали из полученного файла формата .out, были данные о величине площади "перекрывании" 2-х последовательных пар азотистых оснований. Наибольшей площадью перекрывания обладают пары 20 GC/AC (суммарно 12.28).
ex_str -20 stacking.pdb step20.pdb
stack2img -cdolt st20.pdb step20.ps
В результате работы которых была получена картинка стекинг-взаимодействия этих пар в формате .ps. В дальнейшем эта картинка была конвертирована в формат .png (рис.4).
|
Рис.2. Взаимное расположение пар 20 GC/AC. Изображение получено с помощью онлайн конвертера http://image.online-convert.com/ru/convert-to-png.
Смотрите также другие работы:
Дата последнего изменения: 05.10.2014