
|
Учебный сайт Алены Корягиной |

|
Главная |
Oбо мне |
Cеместры |
Cсылки |
Реконструкция филогении по нуклеотидным последовательностям. Паралоги.
1. Реконструкция филогении по нуклеотидным последовательностям
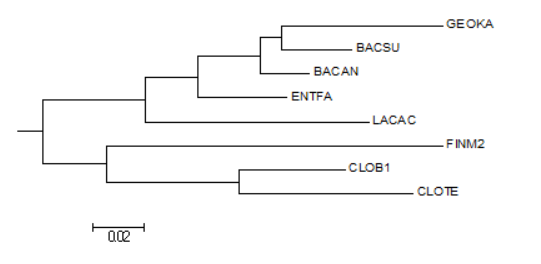
Было построено филогенетическое дерево тех же бактерий, что в предыдущих заданиях (см. здесь и здесь), на основе последовательностей РНК малой субъединицы рибосомы (16S rRNA).
Последовательности 16S рибосомальной РНК каждой бактерии были скачаны из базы полных геномов NCBI, а именно из файла, содержащего все последовательности РНК, расширение которого .frn (скачать файл с последовательностями). Далее полученные последовательности были выровнены с помощью сервера Muscle (скачать выравнивание), и по этому выравниванию построено дерево методом «Maximum likelihood» в программе MEGA (Рис.1.А). Также это дерево было укоренено в среднюю точку с помощью программы retree пакета PHYLIP.
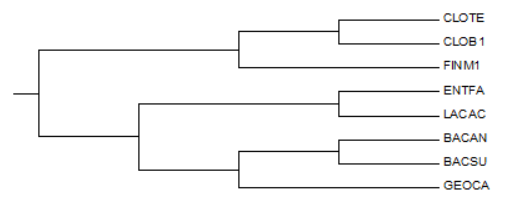
При сравнении с ранее построенным деревом на основе таксономии (рис.1.С) было выявлено, что правильно реконструированы только две следующие нетривиальные ветви:
{CLOTE, CLOB1} против {FINM1, ENTFA, LACAC, BACAN, BACSU, GEOCA}
{CLOTE, CLOB1, FINM1} против {ENTFA, LACAC, BACAN, BACSU, GEOCA}.
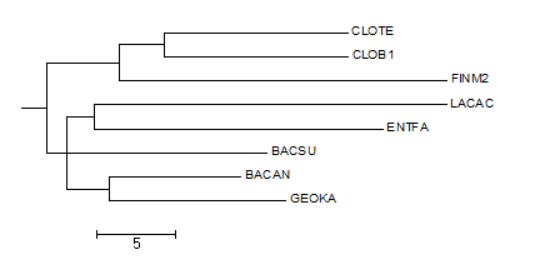
Дерево, построенное на основе нуклеотидных последовательностей значительно хуже отражает действительность, чем деревья, построенные на основе выравнивания белков (рис.1.В). Это связано с тем, что выравнивания нуклеотидных последовательностей сами по себе менее достоверны, чем выравнивания соответствующих белков, так как нуклеотидные последовательности состоят только из 4 букв в то время как, белковые из 20 и больше, и, следовательно, вероятность случайного совпадения позиций намного больше, чем для белковых последовательностей.
|
|
|
A |
B |
C |
Рис.1. Филогенетическое дерево восьми выбранных видов бактерий отдела Firmicutes, А - построенное на основании выравнивания нуклеотидных последовательностей 16S рРНК, В – построенное на основании выравнивания факторов инициации транляции 2, С – построенное на основании таксономии. Изображения получены с помощью программы MEGA.
2. Построение и анализ дерева, содержащего паралоги
Был проведен поиск гомологов белка CLPX_BACSU среди белков восьми выбранных бактерий. Он осуществлялся с помощью программы blastp на сервере kodomo. Команды:
makeblastdb -in proteomes.fasta -dbtype prot
blastp -query CLPX_BACSU.fasta -db proteomes.fasta -evalue 0.001 -outfmt 7 -out blastp.out
На основе полученного файла, содержащего информацию о гомологах, был создан файл-список идентификаторов белков. Команда:
cat blastp.out | egrep -v '#' | awk ' {print $2}' | awk ' BEGIN {FS = "|"} {print "fasta::proteomes.fasta:" $3}' > homol_id.bs
Этот список был подан на вход программе seqret:
seqret @homol_id.bs
В результате был получен файл, содержащий аминокислотные последовательности гомологов белка CLPX_BACSU, которые были выровнены с помощью онлайн программы Muscle (файл с выравниванием). Даллее было построено дерево (рис.1) полученных гомологов с помощью метода Maximum likelihood (скачать файл в формате .nwk).
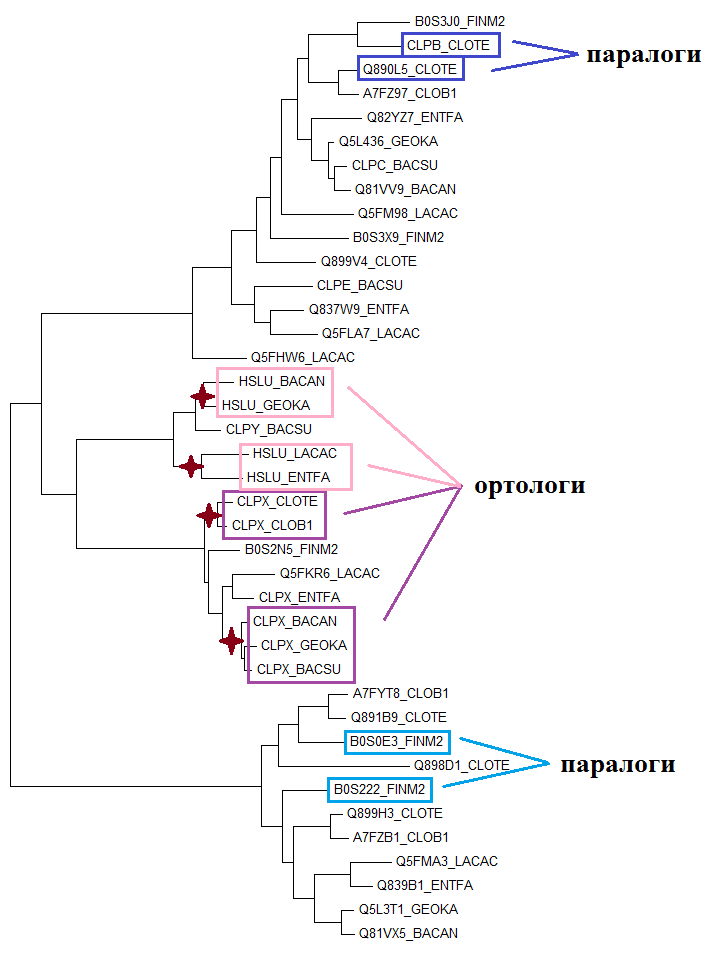
Рис. 2. Филогенетическое дерево гомологов белка CLPX_BACSU среди белков восьми бактерий отдела Firmicutes. Построено методом Maximum likelihood. Розовыми и фиолетовыми рамочками выделены 2 группы попарно ортологичных белков, синим и голубым - паралоги. Красными звёздочками помечены примеры разделения путей эволюции белков в результате видообразования.
На основании построенного дерева были выявлены ортологичные белки и белки паралоги. Ортологичные белки относятся а разным организмам и разделение их общего предка на линии, ведущие к ним, произошло в результате видообразования. На рисунке 1 выделено розовой и фиолетовой рамочкой две группы гомологичных белков. Внутри каждой группы белки попарно являются ортологами. Если два гомологичных белка из одного организма, то такие белки являются паралогами. На рисунке 1 синими и голубыми рамочками отмечены 2 пары паралогов. Также на дереве красными звездочками обозначены разделение путей эволюции в результате видообразованя. Такого эволюционного события, как дупликация гена на рассматриваемом дереве не найдено.
Дата последнего изменения: 03.09.15